GEO转录组芯片数据分析全流程:无gene symbol的R处理技巧(以GSE69063为例)
GEO数据库转录组芯片数据处理与R分析:以GSE65682为例中我们提到过,GEO数据集如果只包含探针ID和对应的ENTREZID,则需要先将探针ID 转换为ENTREZID,再将ENTREZID转换为gene symbol。那么具体是需要进行什么操作呢?以数据集GSE69063为例,为大家详细演示下如何使用R script窗口的脚本进行下载和分析。
1.数据集获取
我们先进入GEO网站官网,根据GEO数据库转录组芯片数据处理与R分析:以GSE65682为例中的方法找到数据集GSE69063的注释文件GPL19983。

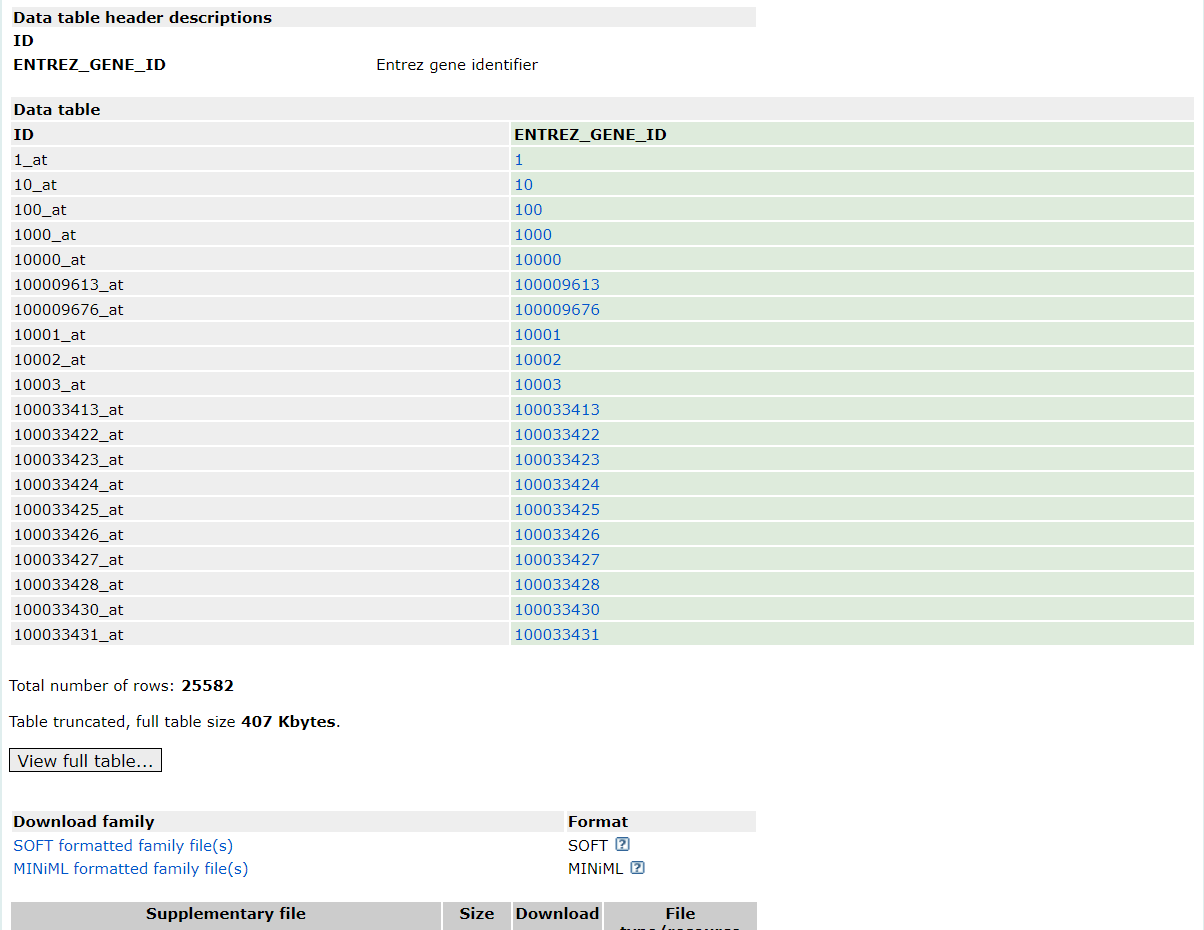
点击GPL19983后,将页面滚动至“Data table header descriptions”部分,发现注释文件中只有探针ID和ENTREZID,并没有我们需要的gene symbol,那这个时候我们该怎么处理呢?其实处理方法很简单,只需要先将探针ID 根据注释文件转换为ENTREZID,再将ENTREZID转换为gene symbol。
2. 数据下载与处理
获取以上信息后即可直接进入R
获取数据
#下载安装相关的R包; #library(BiocManager) #install("GEOquery") #加载所需R包; library(GEOquery) library(limma) library(affy) library(data.table) library(dplyr) # 从GEO数据库下载GSE69063数据集 # destdir="." 表示下载到当前工作目录 # AnnotGPL = T 表示下载注释文件 # getGPL = T 表示获取平台信息 # GSEMatrix = T 表示以GSEMatrix格式获取数据 gset <- getGEO('GSE69063', destdir=".", AnnotGPL = T, ## 注释文件 getGPL = T, GSEMatrix = T) ## 平台文件 # 获取表达矩阵(基因表达数据) exp <- exprs(gset[[1]]) # 获取样本的临床信息 cli <- pData(gset[[1]]) ## 获取临床信息 # 获取平台注释信息(探针与基因的对应关系) GPL <- fData(gset[[1]]) ## 获取平台信息
获取到的表达矩阵行名为芯片的ID,列名为样本ID。
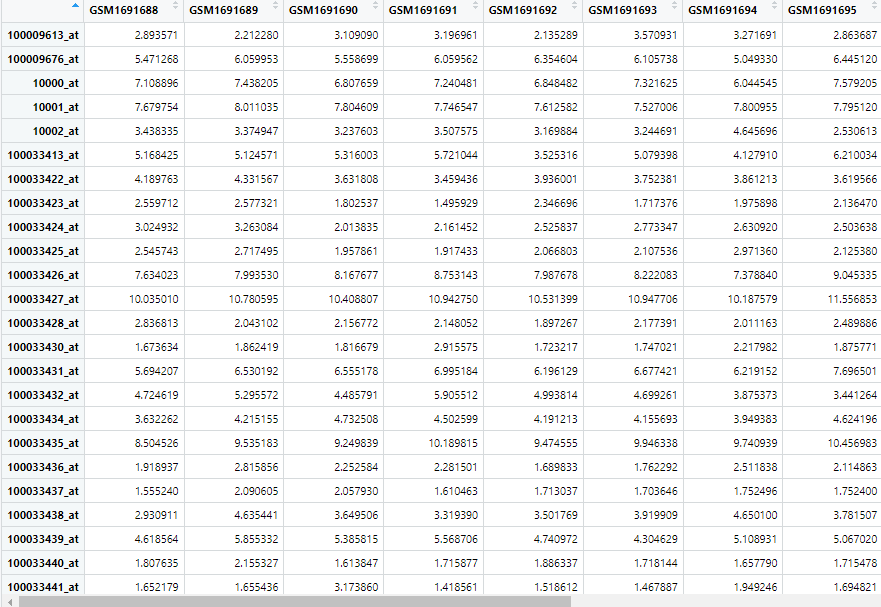
获取到的临床信息和注释文件
临床信息

注释文件
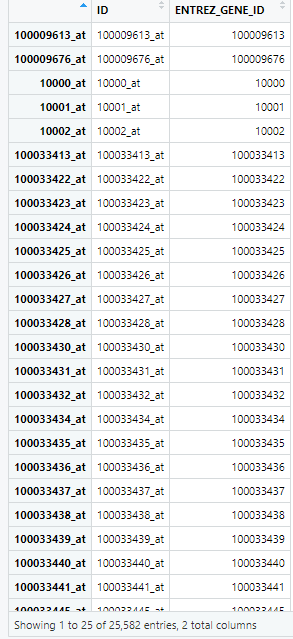
探针ID到ENTREZ ID的转换
# 从平台信息中提取探针ID和ENTREZ基因ID列 gpl <- GPL[, c("ID", "ENTREZ_GENE_ID")] # 创建新的ENTREZID列,从ENTREZ_GENE_ID列提取数据 gpl$"ENTREZID" <- data.frame(gpl$"ENTREZ_GENE_ID", stringsAsFactors = F)[, 1] # 将表达矩阵转换为数据框格式 exp <- as.data.frame(exp) # 在表达矩阵中添加探针ID列,使用行名作为探针ID exp$ID <- rownames(exp) # 将表达矩阵与平台注释信息按探针ID合并,添加ENTREZ基因ID信息 exp_ENTREZ <- merge(exp, gpl, by = "ID") # 移除包含NA值的行 exp_ENTREZ <- na.omit(exp_ENTREZ)
ENTREZ ID到基因符号的转换
# 加载clusterProfiler包,用于基因ID转换 library(clusterProfiler) # 使用bitr函数将ENTREZ基因ID转换为基因符号(SYMBOL) # fromType = "ENTREZID" 指定输入ID类型为ENTREZ # toType = "SYMBOL" 指定输出ID类型为基因符号 # OrgDb = "org.Hs.eg.db" 指定使用人类基因注释数据库 gene_symbol <- bitr(exp_ENTREZ$ENTREZID, # 需要转换的基因ID fromType = "ENTREZID", # 需要转换的类型 toType = c("SYMBOL"), # 需要转换为的类型 OrgDb = "org.Hs.eg.db") # 注释包 # 将表达数据与基因符号信息按ENTREZID合并 exp_symbol <- merge(exp_ENTREZ, gene_symbol, by = "ENTREZID")
数据整理和去重处理
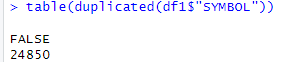
# 将数据框转换为data.table格式,便于数据处理 df <- exp_symbol setDT(df) # 移除ENTREZ_GENE_ID列,因为已经有ENTREZID列 df1 <- df[, !(names(df) == "ENTREZ_GENE_ID"), with = FALSE] # 将data.table转换回数据框格式 df1 <- as.data.frame(df1) # 将基因符号设置为数据框的行名 rownames(df1) <- df1$SYMBOL # 检查基因符号的重复情况 table(duplicated(df1$"SYMBOL"))
# 对重复的基因符号取平均值(去重处理) # 移除SYMBOL列(最后一列),然后对重复的基因符号取平均值 exp_unique1 <- avereps(df1[, -c(ncol(df1))], ID = df1$"SYMBOL") # 将处理后的唯一基因表达矩阵保存为CSV文件 write.csv(exp_unique1, "GSE69063_exp_unique.csv")
最后得到的exp_unique1行名为gene symbol,第一列为ENTREZID,第二列为探针ID,后面的数据就是这个基因在每个样本中的表达水平了。

感谢大家的观看!如果你在学习过程中遇到任何疑问,或者想要进一步探讨相关话题,欢迎随时私信我,或者在评论区留言交流。如果你觉得这个教程对你有所帮助,希望你能动动手指点个赞,收藏起来。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号