GEO数据库数据下载实战:从关键词搜索到原始数据获取
GEO数据库构成:
1. GEO Platform (GPL)芯片平台
定义:描述实验使用的芯片或测序技术平台。
内容:包含探针与基因的对应关系、平台设计参数等元数据。
示例:GPL570(Affymetrix Human Genome U133 Plus 2.0 Array)。
2. GEO Sample (GSM)样本ID号
定义:单个生物样本的实验数据。
内容:原始数据(如CEL文件)、处理后的表达矩阵及样本描述(如疾病状态、处理条件)。
示例:GSM12345(肺癌组织样本的基因表达谱)。
3. GEO Series (GSE)study的ID号
定义:关联多个样本(GSM)的完整研究项目。
内容:实验设计、分析方法及所有相关GSM和GPL的索引。
示例:GSE12345(包含20个样本的肺癌转录组研究)。
4. GEO Dataset(GDS)数据集的ID号
定义:经NCBI整合和标准化的数据集。
内容:统一格式的表达矩阵、实验注释及差异分析工具。
示例:GDS1234(标准化后的乳腺癌基因表达数据集)。
最需要关注的就是数据集对应的GSE号(例如GSE116959)以及数据集的GPL采集平台(例如GPL17077)。
进入GEO数据库官网(https://www.ncbi.nlm.nih.gov/geo/)
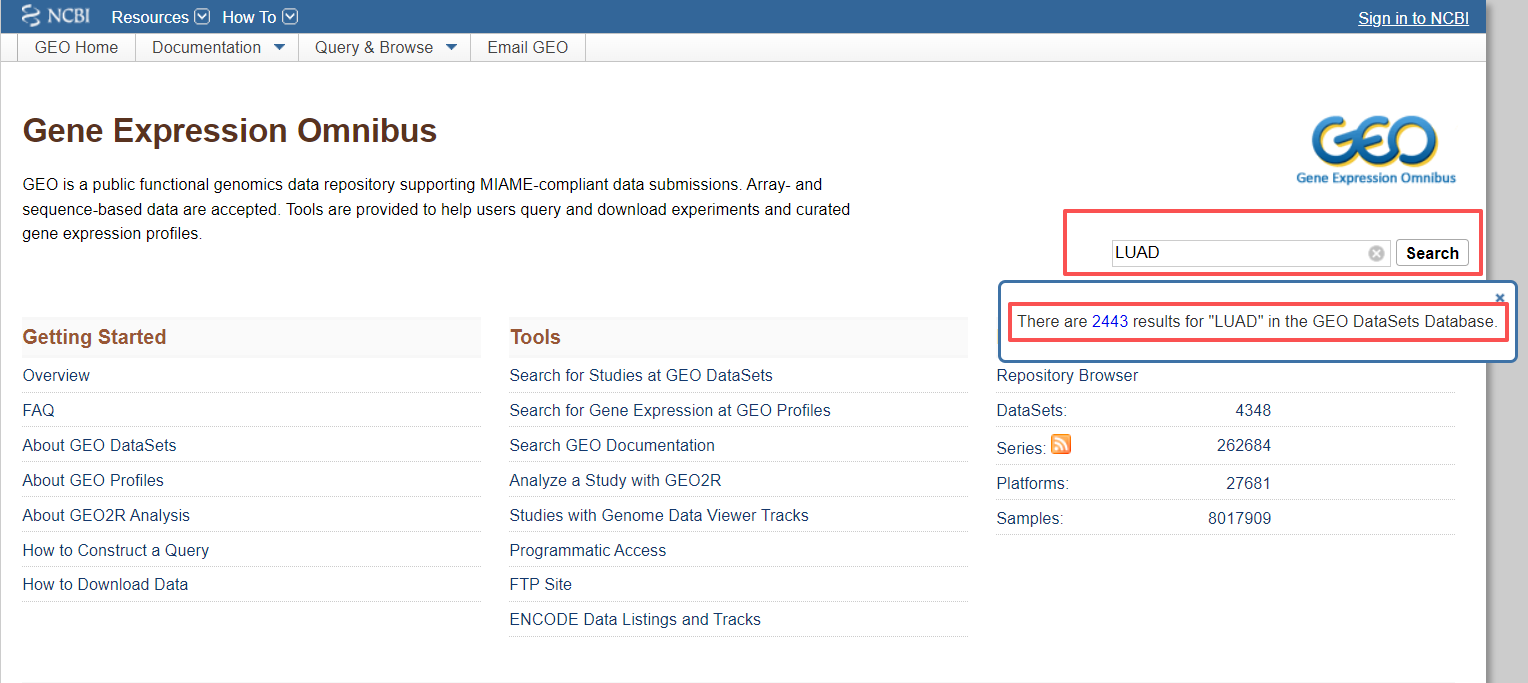
通过搜索所研究目标的关键词或者数据集的GSE的ID可以寻找到相关的数据集,例如我们想寻找肺癌患者的转录组测序数据,可以搜索LUAD
在这个页面可以根据需要筛选数据,物种,数据类型,研究类型
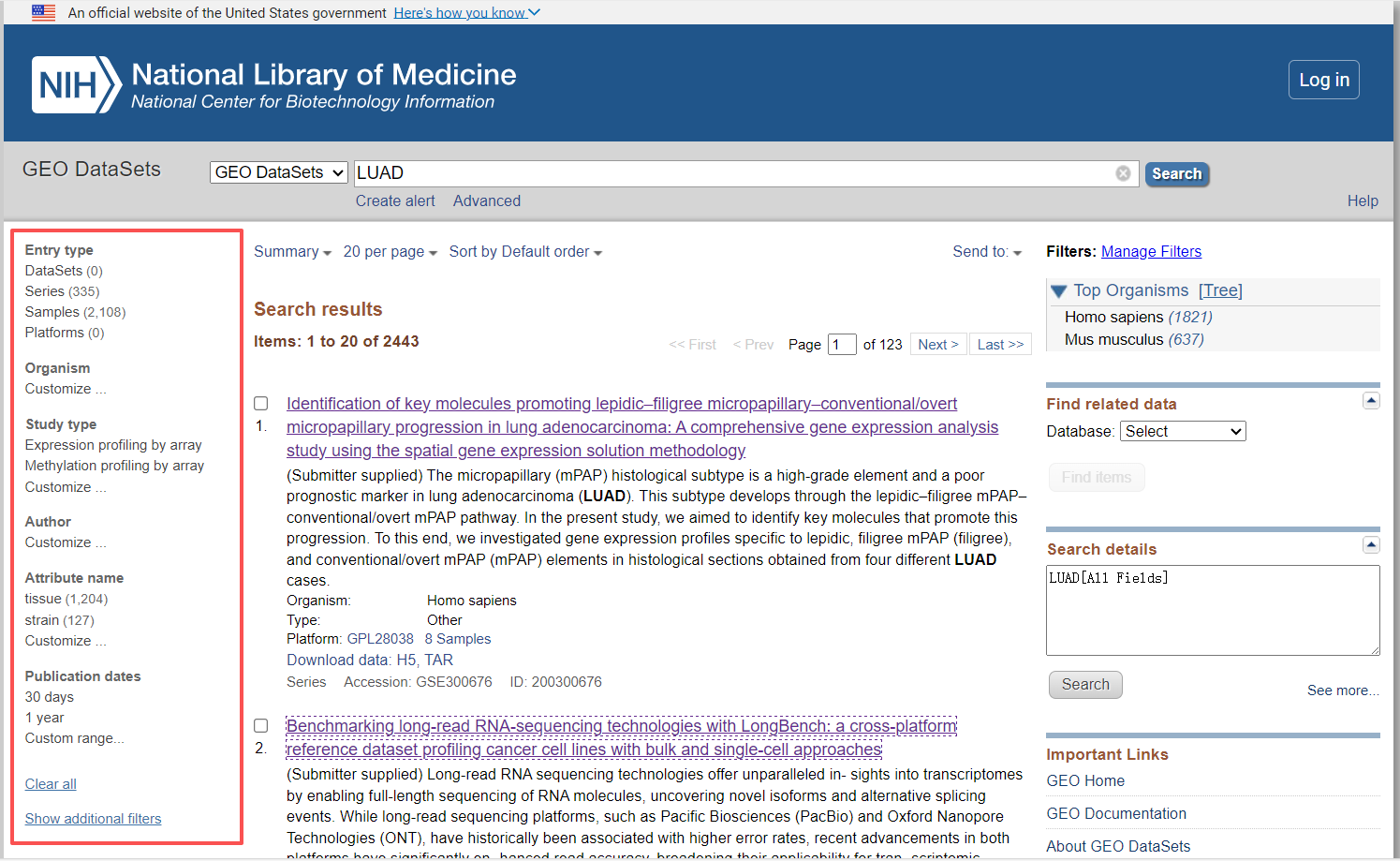
在输入关键词后,需要选择感兴趣的数据集,点击数据集后进入以下页面

首先我们可以看到是关于该study的描述信息,包括文章信息、测序物种、实验类型等等,注意如果转录组数据的Experiment type是Expression profiling by array的话,需要下载对应的GPL采集平台注释文件(例如GPL17077),将探针ID转换为基因ID。如果是Expression profiling by high throughput sequencing,则不需要这一步操作。
在页面的下方,如果我们想下载作者标准化后的数据,可以直接在这个页面中Supplementary file中进行下载,选择Series Matrix File(s),这里会有三个部分信息,一是GSE的一些信息,二是临床信息,三是表达矩阵。
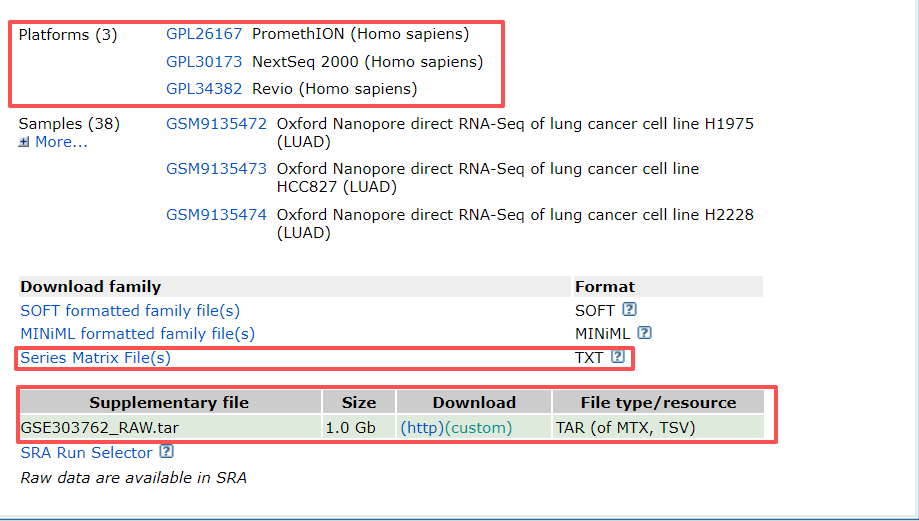
如果想下载原始数据,我们点击页面中Sample对应的GSM的ID,每个样本都对一个GSM,我们以第一个为例,点击后进入以下界面
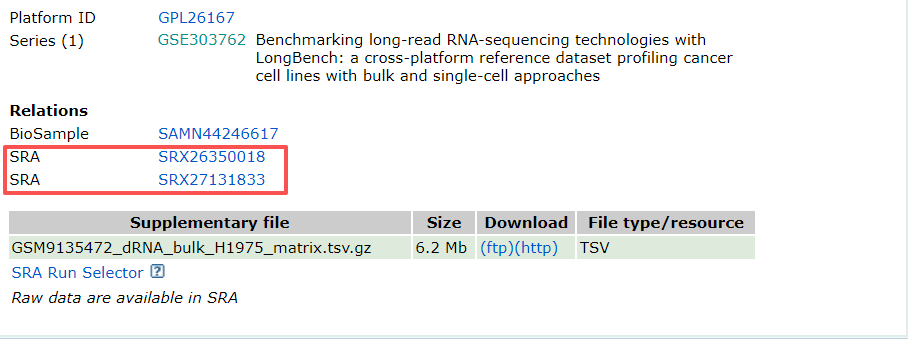
然后点击最下方SRA编号,进入下面的页面
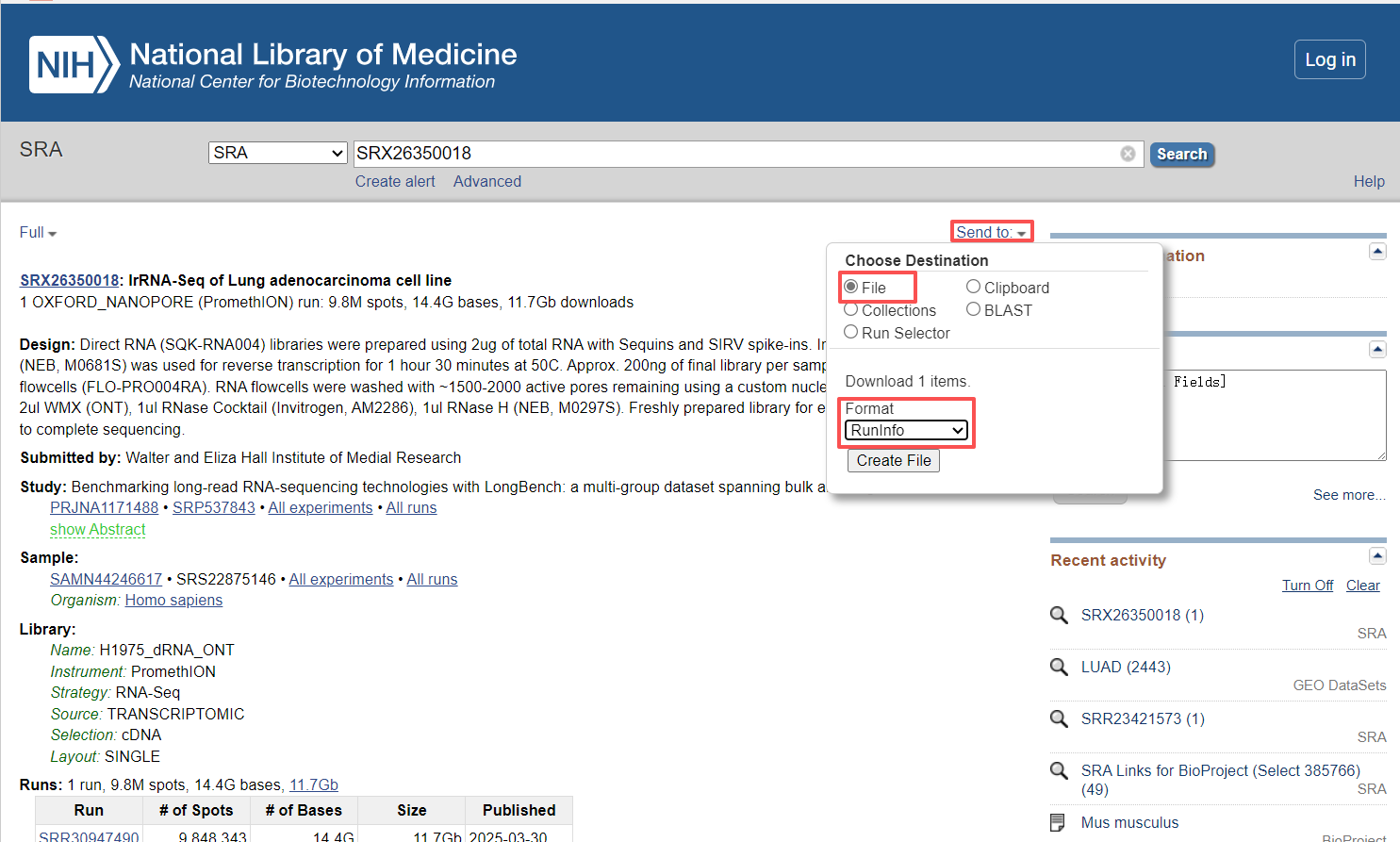
这里面包含了这个样本的基本信息,包括测序平台、文库类型、数据量等基本信息,然后点击页面右上方的Send to按钮,选择File,Format选择RunInfo,然后点击Creat file下载一个csv文件,打开文件,可以看到一个下载链接,点击链接就可以直接下载数据了。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号