GEO单细胞数据建立Seurat对象全过程与错误修复示例
进行单细胞转录组分析,有时候用到公共数据集,尤其是从GEO下载的10X数据,很多研究者常常会遇到一个令人头疼的问题——数据明明能读入,却无法正常创建Seurat对象。本文通过一个真实案例,带大家一起排查并解决这一问题。
这次遇到的问题发生在对数据集“GSM533785HC2”进行分析时。和往常一样,首先加载Seurat包,然后使用CreateSeuratObject()函数创建Seurat对象:
library(Seurat) SeuratObject = CreateSeuratObject(counts = Read10X('GSM533785HC2',gene.column = 2), project = 'GSM533785HC2', min.cells = 3, min.features = 200)
结果报错Error: No feature overlap between existing object and new layer data.
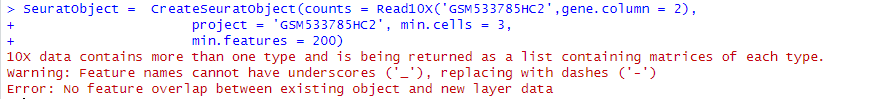
但是有运行的一些信息
10X data contains more than one type and is being returned as a list containing matrices of each type.Warning: Feature names cannot have underscores ('_'), replacing with dashes ('-')
说明数据已经能读取,但是并不能建立seruat对象,查看matrix矩阵信息与features信息、barcodes细胞数据是否一致,细胞数量,基因数量都对应上了,matrix第一列基因,第二列细胞,也都对应上了,到底哪里出了问题?


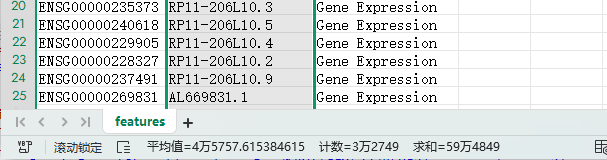
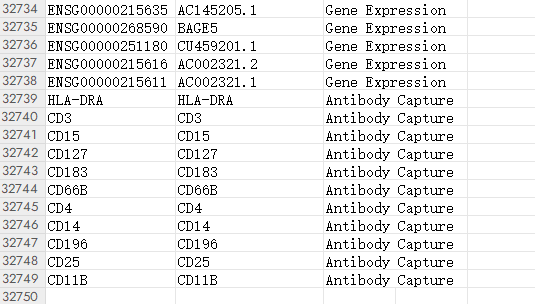
经过查看features文件,在文件末尾发现基因类型不一样,正常情况下都是Gene Expression,多出了Antibody Capture.
既然前面发现了数据能读取,那么先使用Read10X读取文件。

查看一下counts,发现counts是一个list,并不是创建seurat对象的矩阵文件,当中有Gene Expression和Antibody Capture两个矩阵,问题就是这里了。

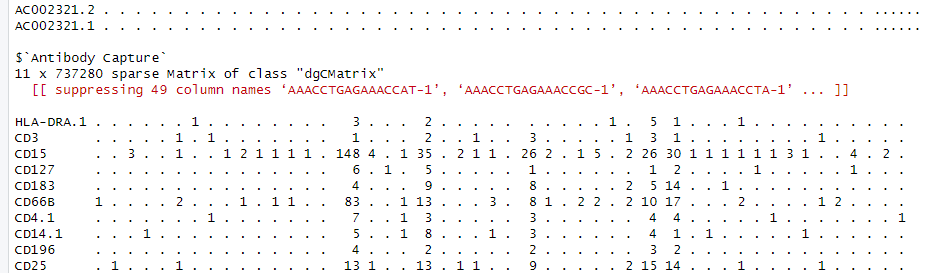
那么接下来从counts中提取Gene Expression矩阵进行创建即可
SeuratObject = CreateSeuratObject(counts = counts$`Gene Expression`, project = 'GSM533785HC2', min.cells = 3, min.features = 200)
运行结果没问题,并且能正常的NormalizeData。
从这个案例可以看出,Seurat读取10X数据时会自动识别不同类型的数据层,如果数据中既包含RNA表达量,又包含抗体捕获信号或其他组学类型,就会返回一个list结构而不是单一矩阵。若不加注意直接传入CreateSeuratObject,就会触发“feature不匹配”的报错。
因此,在处理公共数据或多模态数据时,建议在创建对象之前,始终先用str()或names()查看Read10X()的返回结构,明确其中包含哪些数据类型,再提取合适的那一部分用于构建Seurat对象。
下载GEO或ArrayExpress中的单细胞数据时都会遇到类似困扰。错误信息中的“feature overlap”通常意味着输入数据结构与Seurat期望的矩阵格式不符,当Read10X()输出为list时,一定要注意手动提取Gene Expression矩阵。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号