2026-01-13-Nature Genetics | 本周最新文献速递

Article Title: A breakage–replication-fusion process explains complex rearrangements and segmental DNA amplification
中文标题: 染色体碎裂机制重塑!“断裂-复制-融合”过程揭示复杂基因组重排与DNA扩增之谜
关键词: 染色体碎裂、基因组重排、DNA复制、基因扩增、癌症基因组学
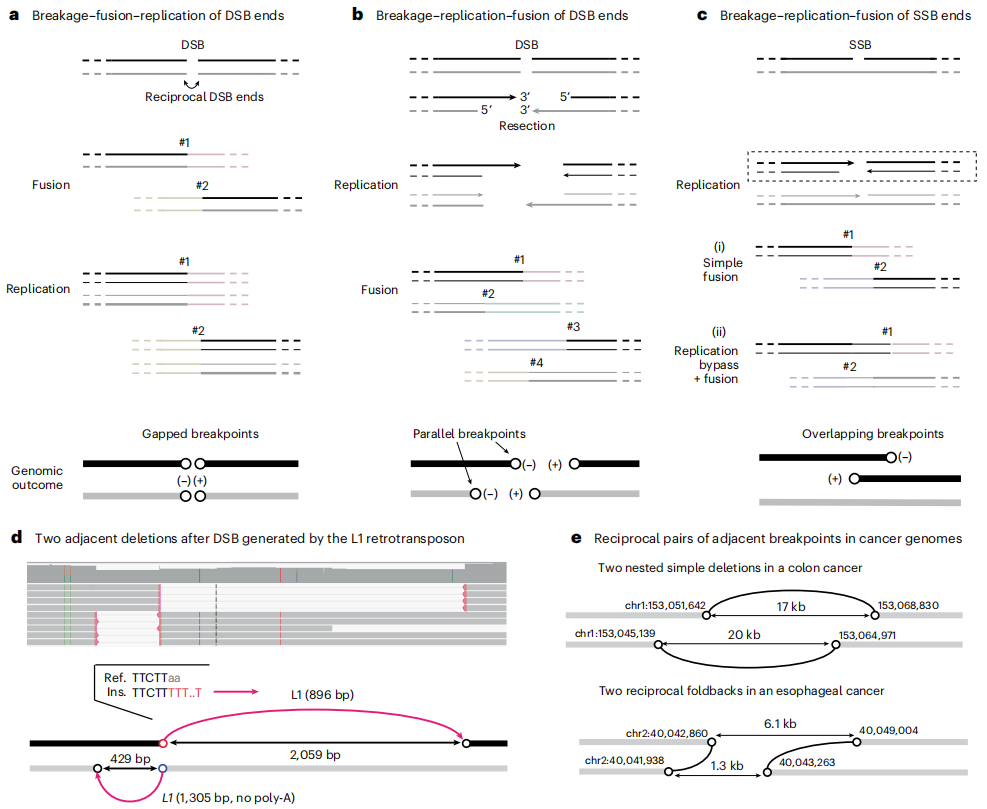
摘要总结: 这篇文章通过分析癌症基因组和实验诱导的染色体碎裂模型,探索了基因组复杂重排和拷贝数增加的产生机制,这对于理解癌症基因组的不稳定性具有重要意义。作者提出了一种新的“断裂-复制-融合”(breakage–replication/fusion)模型,即断裂的DNA末端在S期进行复制,生成的姐妹DNA末端再进行融合。该模型成功解释了癌症基因组中常见的三个特征:相邻的平行断点(adjacent parallel breakpoints)、长片段的低水平扩增、以及倒位重复形式的高水平基因扩增(如双微体)。此外,该机制还解释了重排连接处出现的短插入序列的来源。
文章亮点:
- 提出了“断裂-复制-融合”新机制,修正了传统的断裂-融合-桥(BFB)模型。
- 解释了染色体碎裂后如何在一个细胞周期内产生复杂的节段性扩增。
- 揭示了癌症基因组中“相邻平行断点”这一广泛存在的特征。
- 阐明了重排连接处短插入序列的来源(来自断裂末端的单链DNA)。
文章局限:
- 插入序列的具体生成模型(如聚合酶模板转换与填补合成的竞争)仍有推测成分。
- 主要基于序列特征推断,部分中间过程难以直接实时观测。

Article Title: p53 inactivation drives breast cancer metastasis to the brain through SCD1 upregulation and increased fatty acid metabolism
中文标题: p53失活通过上调SCD1重塑脂肪酸代谢驱动肿瘤脑转移
关键词: 乳腺癌脑转移、p53失活、脂肪酸代谢、SCD1、星形胶质细胞
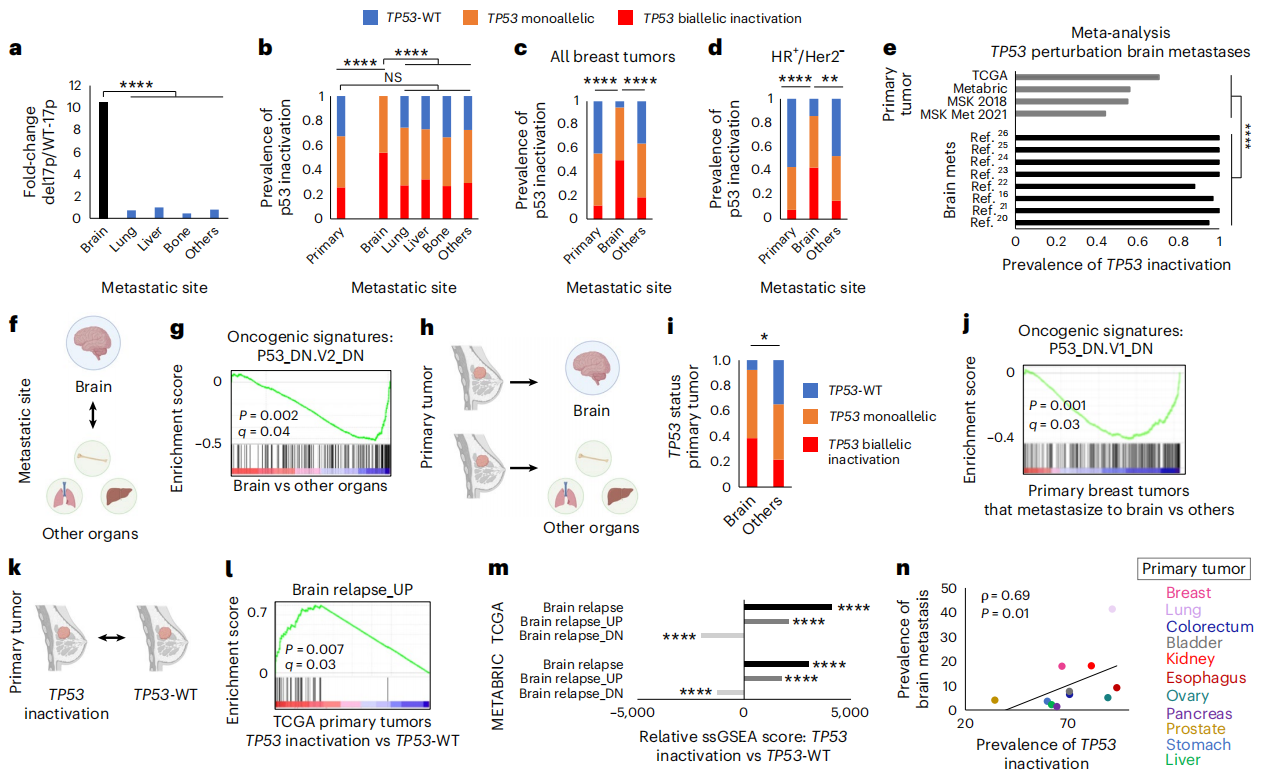
摘要总结: 这篇文章通过结合临床数据分析与动物模型实验,探索了乳腺癌脑转移(BCBM)的分子驱动力,这对于开发针对脑转移的治疗策略具有重要意义。研究发现,TP53基因的失活(突变或缺失)在BCBM中极为普遍。机制上,p53失活解除了对硬脂酰辅酶A去饱和酶1(SCD1)的抑制,导致脂肪酸合成增加。此外,脑微环境中的星形胶质细胞分泌因子进一步促进了p53缺陷细胞的脂质代谢和生长。抑制SCD1可以特异性地抑制p53缺陷型肿瘤在脑内的生长。
文章亮点:
- 确立了p53失活是乳腺癌脑转移的关键驱动因素。
- 揭示了“p53-SREBP1/DEPDC1-SCD1”轴在调控脂肪酸代谢中的作用。
- 发现了星形胶质细胞与癌细胞代谢互作的新机制。
- 证明了SCD1抑制剂是治疗p53缺陷型脑转移瘤的潜在药物。
文章局限:
- 虽然在小鼠模型中验证了SCD1抑制剂的效果,但临床转化仍需验证。
- 星形胶质细胞分泌的具体脂质成分及其摄取机制细节仍需深入研究。

Article Title: Compressive pangenomics using mutation-annotated networks
中文标题: 利用突变注释网络(PanMAN)实现海量基因组的高效压缩与分析
关键词: 泛基因组、数据压缩、突变注释网络、系统发育树、生物信息学
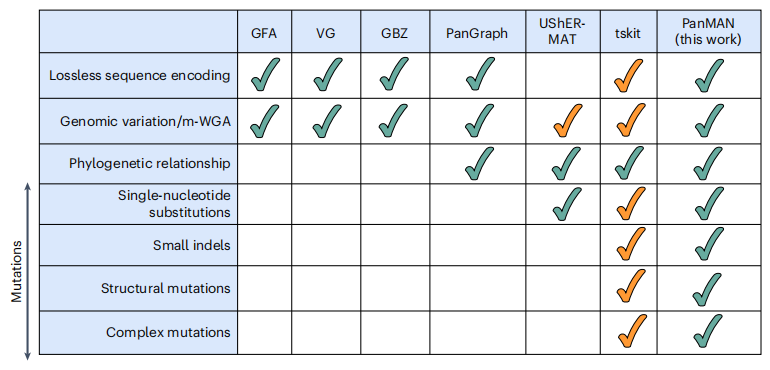
摘要总结: 这篇文章通过开发名为PanMAN(Pangenome Mutation-Annotated Network)的新型数据结构,探索了如何高效存储和分析包含数百万个基因组的泛基因组数据,这对于应对测序数据爆炸式增长具有重要意义。PanMAN结合了系统发育树和突变注释,能够无损地压缩存储基因组序列、变异信息及进化关系。在SARS-CoV-2和微生物基因组数据集上的测试表明,PanMAN相比现有格式(如GFA、VCF)在压缩率上提高了数倍至上千倍,且支持快速的序列检索、进化分析和变异提取。
文章亮点:
- 提出了PanMAN数据结构,实现了泛基因组的超高压缩率(SARS-CoV-2数据压缩3032倍)。
- 同时保留了序列变异、单倍型和系统发育信息。
- 提供了panmanUtils工具箱,支持多种下游分析。
- 解决了现有图泛基因组格式在大规模数据下扩展性差的问题。
文章局限:
- 目前主要应用于微生物和病毒基因组,对结构更复杂的真核生物基因组的适用性有待优化。
- 构建过程依赖于现有的多序列比对和建树工具,构建速度可能受限。

Article Title: Graph pan-genome illuminates evolutionary trajectories and agronomic trait architecture in allotetraploid cotton
中文标题: 棉花育种新篇章!构建高质量图泛基因组揭示异源四倍体棉花的进化轨迹与农艺性状
关键词: 棉花、图泛基因组、结构变异、驯化、全基因组关联分析
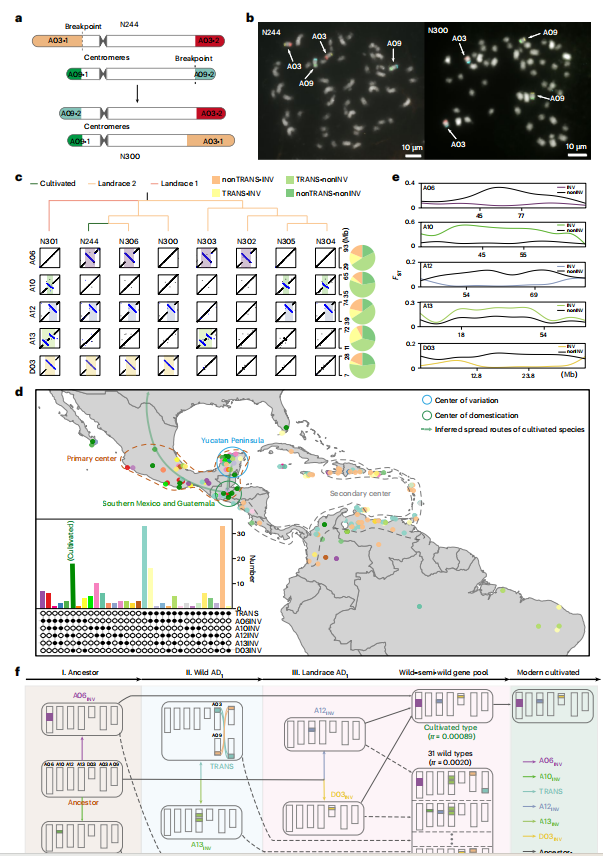
摘要总结: 这篇文章通过对107份代表性陆地棉(Gossypium hirsutum)种质资源进行高深度PacBio HiFi测序和组装,探索了棉花的基因组多样性、驯化历史及重要农艺性状的遗传基础,这对于棉花分子育种具有重要意义。研究构建了高质量的图泛基因组,鉴定了大量的结构变异(SVs),包括一个区分野生和栽培种的大型染色体易位(A03-A09)和五个大倒位。基于存在/缺失变异(PAV)的GWAS分析发现了多个控制纤维品质和抗病性的新位点(如 VWD11 ),揭示了野生种质资源中未被利用的遗传潜力。
文章亮点:
- 构建了包含107个高质量组装的陆地棉图泛基因组。
- 发现并验证了A03-A09染色体易位及多个大倒位是棉花驯化的重要遗传标记。
- 建立了泛-NLR组(Pan-NLRome),解析了抗病基因的演化规律。
- 利用PAV-GWAS挖掘出SNP-GWAS遗漏的关键性状调控基因(如纤维长度和抗黄萎病基因)。
文章局限:
- 尽管组装质量高,但部分复杂的rDNA簇和端粒区域仍未完全解析。
- 半野生/野生种质样本量相对较少,可能未完全覆盖该群体的遗传多样性。
致谢橙子牛奶糖(陈文燕),请用参考模版:We thank the blogger (orange_milk_sugar, Wenyan Chen) for XXX
感谢小可爱们多年来的陪伴, 我与你们一起成长~

本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/19489157



 浙公网安备 33010602011771号
浙公网安备 33010602011771号