Nature Genetics | 解码免疫细胞动态遗传调控机制及其与疾病的关联
关键词
单细胞转录组学、免疫细胞、多组学、遗传调控、COVID-19
摘要总结
这篇文章是2025年8月发表在《Nature Genetics》杂志上的一篇研究,标题为“Deciphering state-dependent immune features from multi-layer omics data at single-cell resolution”。这篇文章通过整合单细胞转录组学、宿主遗传学、血浆蛋白质组学和肠道宏基因组学数据,探索了日本人群中免疫细胞的动态遗传调控及其与COVID-19等疾病的关联,这对于理解复杂疾病的分子机制和推动精准医学具有重要意义。
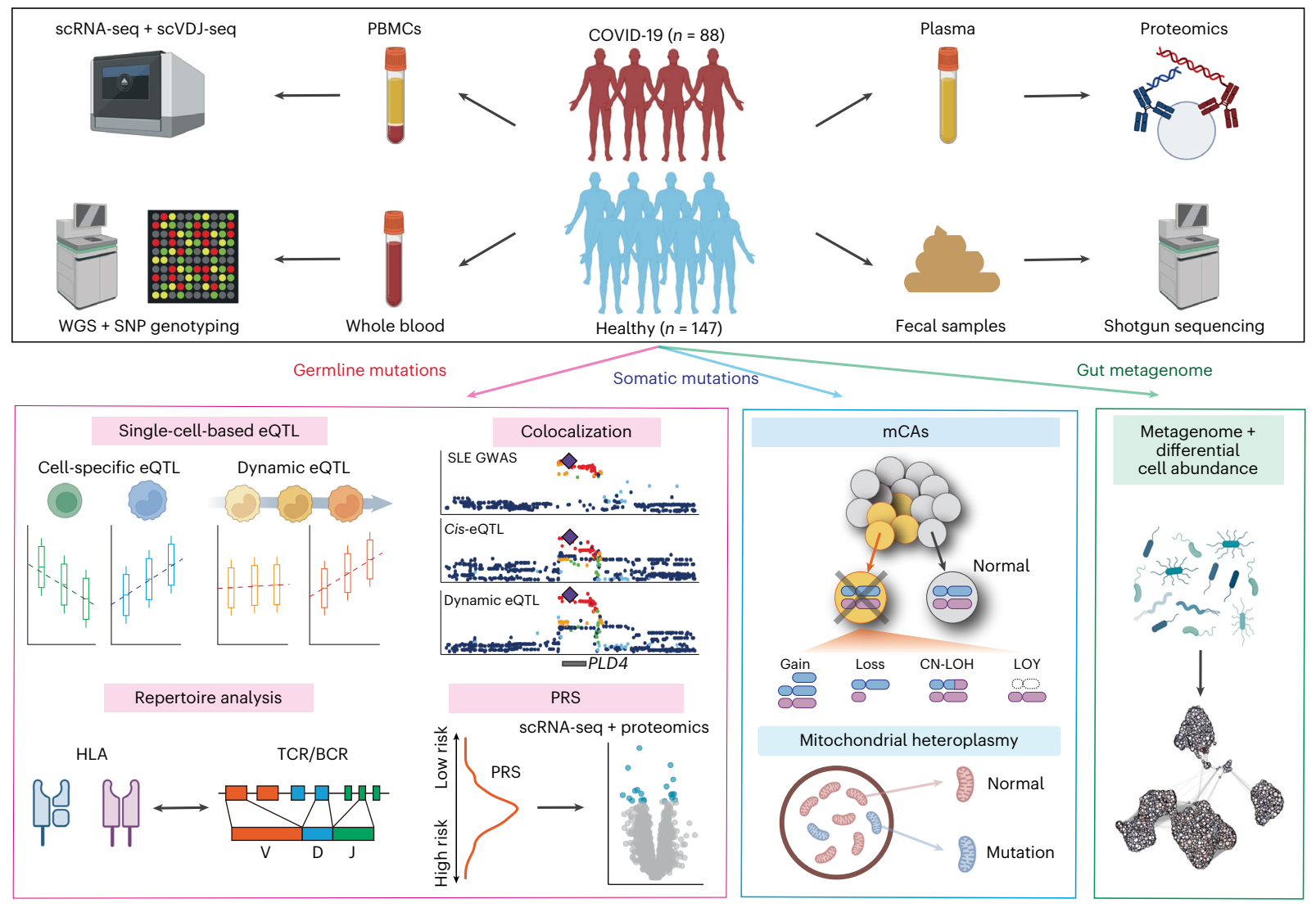
研究背景
人类免疫系统在健康和疾病状态下表现出高度动态的调控特性,但现有分子定量性状位点(mQTL)研究多集中于欧洲人群,且以整体组织水平分析为主,缺乏非欧洲人群的单细胞分辨率数据。此外,体细胞突变(如克隆性造血)在健康个体中的作用及其与疾病(如COVID-19)的关联机制尚不清楚。因此,研究团队构建了Osaka Atlas of Immune Cells(OASIS),旨在通过多组学整合分析揭示日本人群中免疫细胞的动态调控机制。
研究方法
研究对象为235名日本个体(88名COVID-19患者和147名健康个体),通过10x Genomics Chromium平台对超过150万个外周血单核细胞(PBMCs)进行单细胞转录组学(scRNA-seq)和VDJ测序(scVDJ-seq)。同时,结合全基因组测序(WGS)、SNP基因分型、血浆蛋白质组学(Olink assay)和肠道宏基因组学数据,研究团队分析了:1)细胞类型特异性和状态依赖的表达定量性状位点(eQTL);2)HLA及全基因组变异与T/B细胞受体(TCR/BCR)谱系的关联;3)体细胞突变(如镶嵌染色体异常mCAs、Y染色体丢失LOY和线粒体DNA异质性)的单细胞特征;4)肠道微生物群与免疫细胞丰度的关系。分析方法包括伪批量(pseudobulk)eQTL映射、动态eQTL建模、GWAS共定位分析和Milo单细胞邻域分析。
研究结果
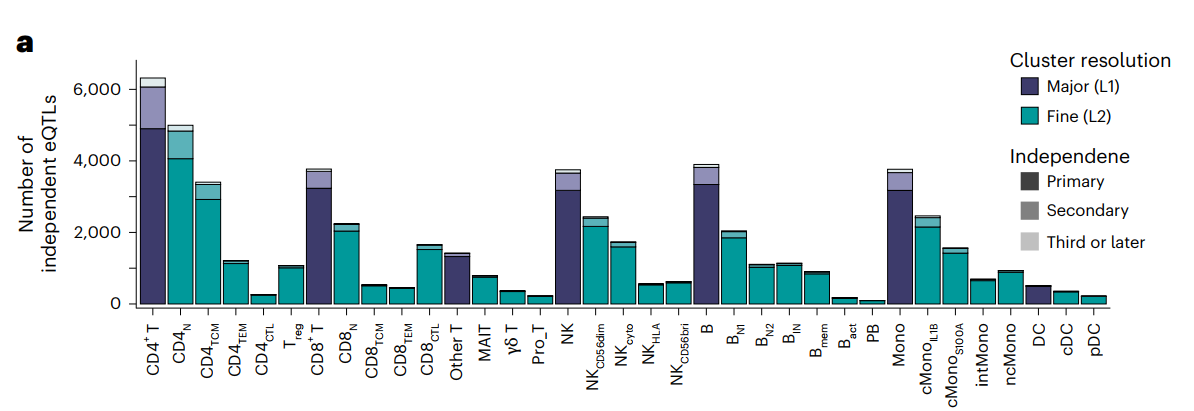
- 单细胞eQTL映射:研究鉴定出23,443个L1级和34,297个L2级eQTL,发现eQTL效应在不同细胞类型和状态间高度异质,特别是在东亚人群中未被欧洲人群数据复制的eQTL与次要等位基因频率(MAF)差异相关。
L1级eQTL是指在主要细胞类型(major cell types)水平上映射的eQTL。文章中将外周血单核细胞(PBMCs)分为七个主要细胞类型(L1),包括CD4+ T细胞、CD8+ T细胞、其他T细胞、NK细胞、B细胞、单核细胞(Mono)和树突状细胞(DC)。
L2级eQTL是指在更细分的细胞状态或亚型(fine cell types/states)水平上映射的eQTL。文章中进一步将PBMCs分为28个细细胞类型(L2),如CD4+初级T细胞(CD4N)、CD8+效应记忆T细胞(CD8TEM)、激活B细胞(Bact)等。
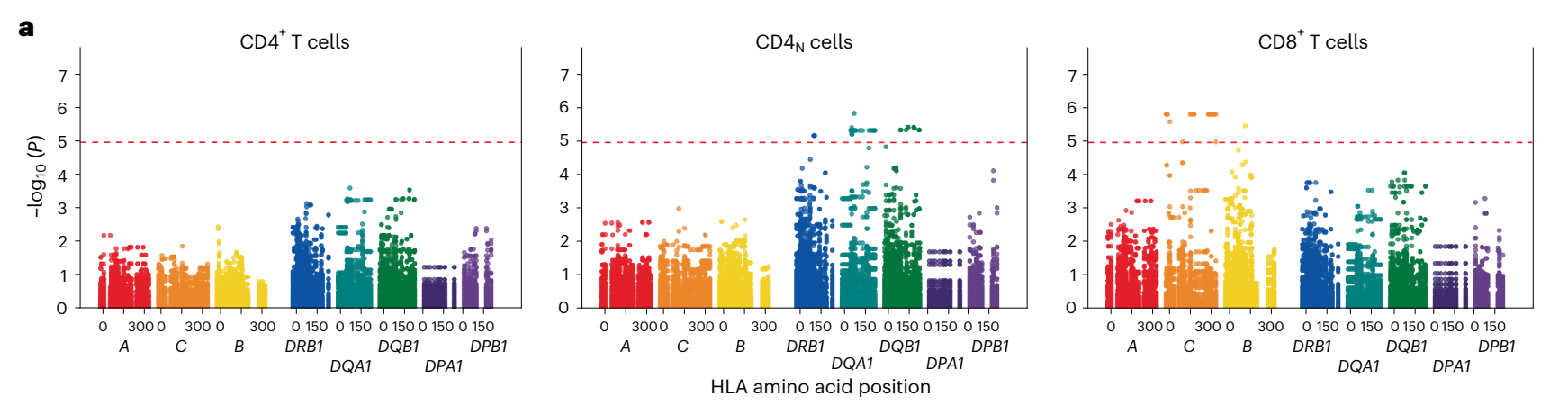
- HLA与TCR/BCR关联:HLA变异与TCR V基因使用在CD4+和CD8+ T细胞中显示特异性关联,尤其在COVID-19患者中观察到HLA I类基因与TRAV基因使用的显著交互作用。
-
动态eQTL分析:在髓系细胞中,530和568个基因在两个免疫相关基因模块中显示动态eQTL效应,与先天免疫和抗原呈递通路相关。
-
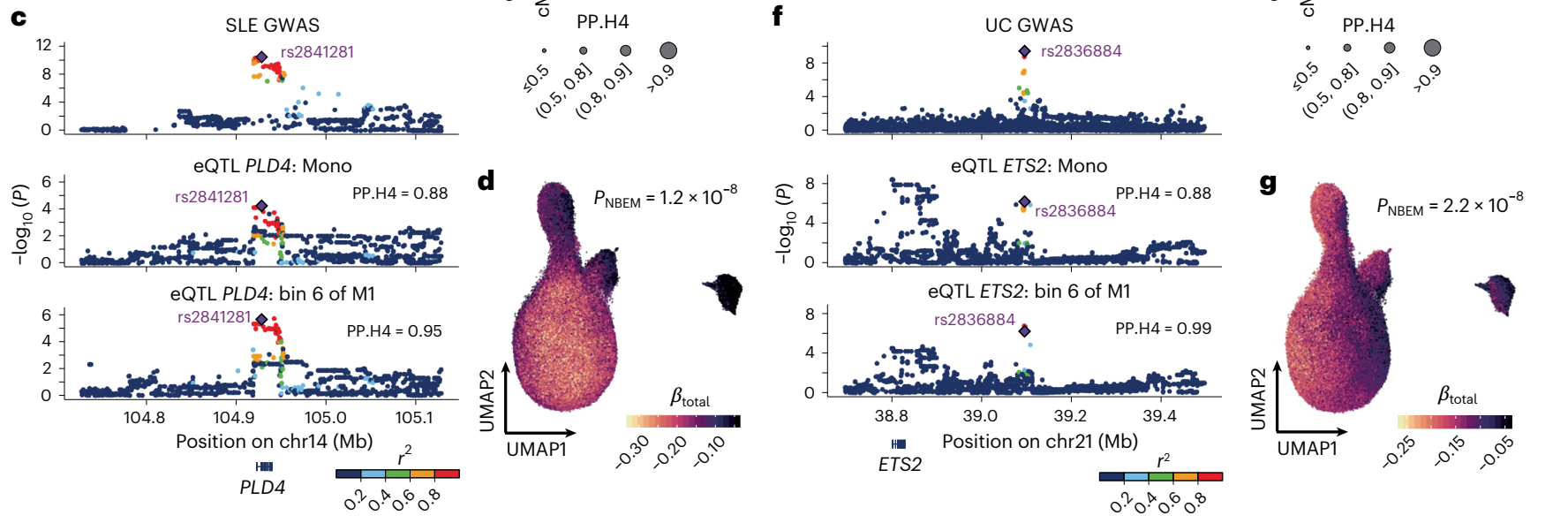
GWAS共定位:121个GWAS位点与eQTL共定位,揭示179个候选疾病相关基因,动态eQTL在解释系统性红斑狼疮和溃疡性结肠炎等疾病信号中具有更高分辨率。
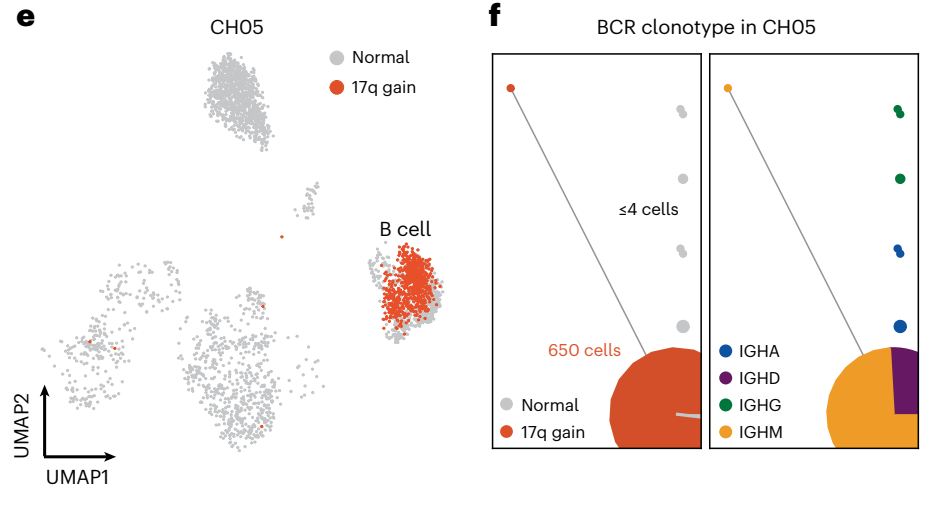
- 体细胞突变:mCAs和LOY在单细胞水平显示细胞类型特异性,例如17q增益在B细胞中富集,伴随免疫通路上调;LOY与COVID-19住院风险显著相关(OR=6.6)。
- 肠道微生物群:Ruminococcus gnavus等菌种丰度与特定免疫细胞(如CD4+毒性T细胞和浆细胞)的差异性丰度相关。
文章的亮点和局限
亮点:
-
首次在非欧洲人群中构建了多组学单细胞免疫图谱,填补了全球基因组学多样性的空白。
-
通过单细胞分辨率揭示了动态遗传调控和体细胞突变对免疫功能的复杂影响,为COVID-19和自身免疫疾病的机制研究提供了新视角。
-
整合多组学数据(转录组、遗传学、蛋白质组、宏基因组)展示了强大的系统生物学分析能力。
局限:
-
体细胞突变分析依赖于高深度测序,可能受限于数据质量和覆盖度。
-
研究主要集中于PBMCs,其他免疫细胞类型的调控机制未被探索。
公开数据
sc-eQTL summary statistics are available at the NBDC under accession ID hum0197 and are also available in an interactive browser at https://japan-omics.jp/. Processed OneK1K scRNA-seq data (h5ad file) were obtained from the Human Cell Atlas (https://explore.data.humancellatlas.org/projects/f2078d5f-2e7d-4844-8552-f7c41a231e52). OneK1K eQTL summary statistics were obtained from https://onek1k.org/.
本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/19032795




 浙公网安备 33010602011771号
浙公网安备 33010602011771号