退而求其次(3)——宿管员的烦恼
书接上文,在《退而求其次(1)——随机法》中宿管员使用了随机法分配宿舍,现在尝试使用遗传算法。
顺序编码和初始种群
遗传算法的首要问题是基因编码。对于分宿舍问题,每种分配方案是一个个体,其基因序列的每一个编码代表一个同学,要求处于同一基因序列中的所有基因代码均不能重复,也就是每个同学都是独一无二的。在这种规则下,使用二进制编码就显得笨拙了。一种简单的编码方案是直接使用同学的序号作为基因编码,这种编码称为顺序码。
顺序码又称自然数编码,使用从1到n 的自然数进行编码,且不允许重复。例如[1,2,3,4,5,6,7,8,9,10,11,12]是一个合法的个体,表示按编码从左到右的顺序四人一个宿舍,而[1,1,3,3,6,6,8,8,9,10,11,12]则不是。
随机选择1000个个体作为初始种群,约占解空间的20%:
1 POPULATION_SIZE = 1000 # 种群数量 2 3 def init_population(): 4 ''' 构造初始种群 ''' 5 population = [] 6 code_len = len(base_data.STUDENTS_NAME) # 编码长度 7 for i in range(POPULATION_SIZE): 8 population.append(base_data.upset()) 9 return population
适应度评估和种群选择
可以利用成本函数cost_fun来进行适应度评估。由于cost_fun识别的是解而不是基因编码,因此在使用cost_fun之前还需要通过solution_adapter将基因编码适配成解,将基因编码[1,2,3,4,5,6,7,8,9,10,11,12]翻译成cost_fun能够有效计算的[[1,2,3,4],[5,6,7,8],[9,10,11,12]]。
1 def solution_adapter(code): 2 ''' 将基因编码翻适配成cost_fun能够识别的解 ''' 3 solution = [] 4 for i in range(0, len(base_data.STUDENTS_NAME), base_data.NUM_PER_DROM): 5 solution.append(code[i:i + base_data.NUM_PER_DROM]) 6 return solution 7 8 def fitness_fun(code): 9 ''' 10 适应度评估 11 :param code: 二进制基因编码 12 :return: 适应度评估值, 二元组, (宿舍总成本, 每个宿舍的成本) 13 ''' 14 return base_data.cost_fun(solution_adapter(code))
我们依然使用锦标赛法选择种群。由于成本越低表示适应度越越高,因此在锦标赛中适应度值低的是胜出者。
1 def selection(population): 2 '''选择策略, 锦标赛法''' 3 pop_next = [] # 下一代种群 4 for i in range(POPULATION_SIZE): 5 tour_list = random.choices(population, k=2) # 二元锦标赛 6 winner = min(tour_list, key=lambda x: fitness_fun(x)) # 成本低的胜出 7 pop_next.append(winner) 8 return pop_next
部分匹配交叉和循环交叉
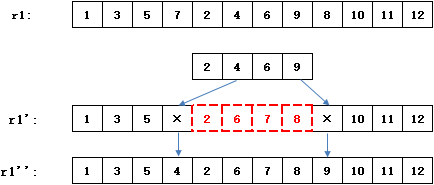
对于宿舍编码来说,单点交叉和两点交叉都无法产生合法的个体,下图展示了一个不合法的单点交叉。
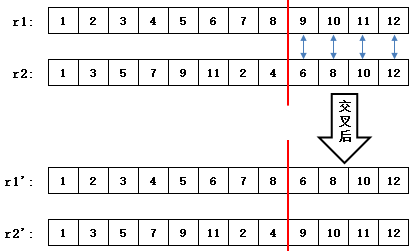
在交叉后的个体 r1' 中,基因代码6和8出现了两次,表示6号同学和8号同学同时住在两个宿舍,这显然不是一个合法的解。反过来,如果交叉后得到了合法的个体,那么新个体又会和它们的父代没有任何区别,即没有产生任何新个体:
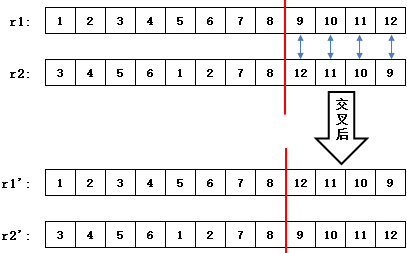
r1和 r1' 没有任何区别,最后一个宿舍的四个同学仅仅是交换了一下床位。看来必须另辟他径,寻找其它的交叉策略。
部分匹配交叉
部分匹配交叉(Partially Matched Crossover,PMC)在1985年被提出,是由两点交叉改进而来的。部分匹配交叉的第一步和两点交叉一样,首先在个体基因序列中随机设置两个交叉点,然后随机选择两个个体做为父代个体,相互交换它们交叉点之间的那部分基因块
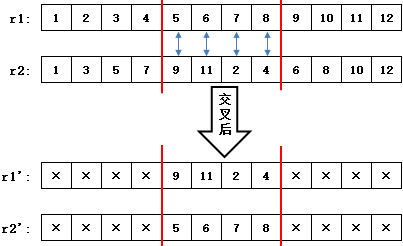
在交叉时需要记住交叉前的基因块r1→[5,6,7,8],r2→[9,11,2,4] 。
接下来对交叉后生成的新个体 r1' 和 r2' 中的×部分,分别继承r1和 r2 中对应位置的编码,如果待继承的编码在交换后的基因块中,则不做继承:
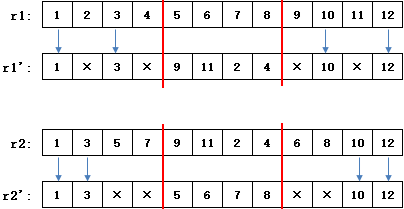
最后,把交叉前记住的基因块按顺序依次填入×部分,得到最终的r1'和 r2' :
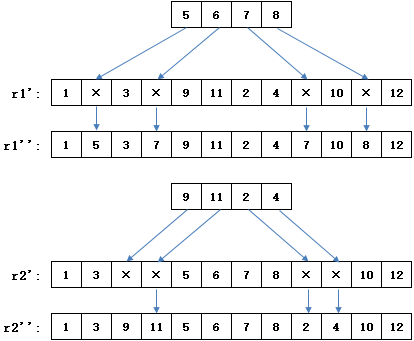
如果交叉前的编码已经在 r1' 中,则略过该编码。下图在替换x时需要略过2和6:
r1 是初始个体,它的基因块[2,4,6,9]与另一个个体的对应基因块[2,6,7,8]交叉,得到[×,×,×,×.2,6,7,8,×,×,×,×]。继承 r1 后得到 r1' = [1,3,5,×,2,6,7,8,×,10,11,12]。在[2,4,6,9]中,编码2和6已经在 r1' 中,因此只有4和9可以替换对应的×。
部分匹配交叉的编码如下。
1 def crossover_pmc(population): 2 ''' 部分匹配交叉(PMC)''' 3 4 def create_mapping(cross_code_1, cross_code_2): 5 ''' 6 建立两个交叉片段间的映射关系 7 :param cross_code_1: 8 :param cross_code_2: 9 :return: 映射关系set 10 ''' 11 mapping = set() 12 for i in range(len(cross_code_1)): 13 c1, c2 = cross_code_1[i], cross_code_2[i] 14 if (c1, c2) not in mapping and (c2, c1) not in mapping: 15 mapping.add((c1, c2)) 16 return mapping 17 18 def code_extends(child, parent, start, end): 19 ''' 20 继承父代的编码 21 :param child: 子代个体 22 :param parent: 父代个体 23 :param start: 基因编码起始位置 24 :param end: 基因编码终止位置 25 ''' 26 for i in range(start, end): 27 if parent[i] not in child: 28 child[i] = parent[i] 29 30 def code_rest(child, cross_code): 31 ''' 32 通交叉前的基因片段修改子代的编码 33 :param child: 子代个体 34 :param cross_code: 交叉前的的基因片段 35 :return: 36 ''' 37 for i, x in enumerate(child): 38 if x != -1: 39 continue 40 for x_old in cross_code: 41 if x_old not in child: 42 child[i] = x_old 43 break 44 45 pop_new = [] # 新种群 46 code_len = len(population[0]) # 基因编码的长度 47 for i in range(POPULATION_SIZE): 48 # 选择两个随机的交叉点 49 p1, p2 = random.randint(0, code_len - 1), random.randint(0, code_len - 1) 50 if p1 > p2: 51 p1, p2 = p2, p1 52 parent1, parent2 = random.choices(population, k=2) # 选择两个随机的个体 53 cross_code_1 = parent1[p1:p2] # 交叉前的编码块 54 cross_code_2 = parent2[p1:p2] # 交叉后的编码块 55 # 构造新的个体,-1表示基因编码尚未确定 56 r = [-1] * p1 + cross_code_2 + [-1] * (code_len - p2) 57 code_extends(r, parent1, 0, p1) # 继承父代的编码 58 code_extends(r, parent1, p2, code_len) # 继承父代的编码 59 mapping = create_mapping(cross_code_1, cross_code_2) # 两个交叉块的映射关系 60 code_rest(r, cross_code_1) # 通过交换前的基因片段确定剩余编码 61 pop_new.append(r) 62 return pop_new
循环交叉
循环交叉(Cycle Crossover,CX)是另一种适合顺序编码的交叉策略。不同于其它交叉策略,循环交叉不需事先要选择交叉点。
假设有两个父代个体
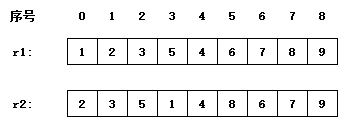
先从 r1 中选择第0个编码,作为子代 r1' 的第一个编码:
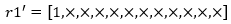
r2 的第0个编码是2,因此 r1' 中第2个被确定的编码是2:
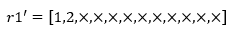
2在 r1 中的序号是1, r2[1]=3, r1' 中第3个确定的编码是3:
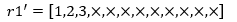
3在 r1 中的序号是2, r2[2]=5, r1' 中第4个确定的编码是5:

5在 r1 中的序号是3, r2[3]=1 , 和 r1' 中第1个编码相同,至此称为一个循环。剩余未确定的编码从 r2 的对应位置映射即可
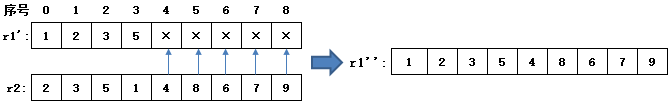
循环交叉匹配的代码如下:
1 def crossover_cx(population): 2 ''' 循环交叉匹配 ''' 3 pop_new = [] # 新种群 4 code_len = len(population[0]) # 基因编码的长度 5 for i in range(POPULATION_SIZE): 6 parent1, parent2 = random.choices(population, k=2) # 选择两个随机的个体 7 r_new = [-1] * code_len # 新个体 8 r_new[0] = parent1[0] 9 i = 0 10 while True: # 循环交叉 11 x = parent2[i] 12 if r_new[0] == x: 13 break 14 i = parent1.index(x) 15 r_new[i] = x 16 # r_new中剩余未确定的编码直接从parent2中继承 17 for i, x in enumerate(r_new): 18 if x == -1: 19 r_new[i] = parent2[i] 20 pop_new.append(r_new) 21 return pop_new
变异
顺序编码的变异策略很简单,仅仅是将两个随机变异点的编码互相交换:
1 def mutation(population): 2 ''' 变异 ''' 3 code_len = len(population[0]) # 基因编码的长度 4 mp = 0.2 # 变异率 5 for i, r in enumerate(population): 6 if random.random() < mp: 7 # 两个随机变异点 8 p1, p2 = random.randint(0, code_len - 1), random.randint(0, code_len - 1) 9 # 交换两个变异点的数据 10 population[p1], population[p2] = population[p2], population[p1]
分配宿舍
准备工作已经就绪,可以开始使用遗传算法分配宿舍:
1 def sum_fitness(population): 2 ''' 计算种群的总适应度 ''' 3 return sum([fitness_fun(code)[0] for code in population]) 4 5 def ga(): 6 ''' 遗传算法分配宿舍 ''' 7 population = init_population() # 构建初始化种群 8 s_fitness = sum_fitness(population) # 种群的总适应度 9 i = 0 10 while i < 10: # 如果连续10代没有改进,结束算法 11 pop_next = selection(population) # 选择种群 12 pop_new = crossover_cx(pop_next) # 交叉 13 mutation(pop_new) # 变异 14 s_fitness_new = sum_fitness(pop_new) # 新种群的总适应度 15 if s_fitness > s_fitness_new: # 成本越低,适应度越高 16 s_fitness = s_fitness_new 17 i = 0 18 else: 19 i += 1 20 population = pop_new 21 # 按适应度值从大到小排序 22 population = sorted(population, key=lambda x: fitness_fun(x), reverse=True) 23 # 返回最优的个体 24 return population[0] 25 26 if __name__ == '__main__': 27 best = ga() 28 solution = solution_adapter(best) 29 total_cost, dorms_cost = base_data.cost_fun(solution) 30 base_data.print_solution(solution, total_cost, dorms_cost)
需要注意的是第15行的适应度比较,由于这里使用的是成本函数,因此种群的成本值越低,适应度越高,越应该被保留。一种可能的运行结果:

可以看到,在总成本较低的同时,“不均”的问题也得到了解决。
总体代码
base_data.py:
1 import random 2 3 # 学生调查表数据 4 STUDENTS = [ 5 [32, 1, 2, 2, [11, 33, 42], 5], 6 [32, 1, 2, 1, [11], 5], 7 [41, 1, 2, 5, [21, 22], 4], 8 [43, 2, 3, 3, [11, 21], 3], 9 [36, 2, 3, 4, [11, 33], 3], 10 [44, 2, 3, 4, [41, 42], 2], 11 [42, 1, 2, 1, [11, 12], 1], 12 [32, 1, 1, 2, [31, 32], 2], 13 [61, 1, 1, 3, [51], 3], 14 [61, 1, 1, 2, [13], 3], 15 [44, 3, 4, 1, [21, 43], 1], 16 [22, 3, 4, 4, [22, 43], 2] 17 ] 18 # 学生姓名 19 STUDENTS_NAME = ['蕾娜', '琪琳', '蔷薇', '炙心', 20 '灵犀', '莫伊', '怜风', '语琴', 21 '凉冰', '鹤熙', '瑞萌萌', '何蔚蓝'] 22 DROM_SIZE = 3 # 宿舍数量 23 NUM_PER_DROM = 4 # 每个宿舍的人数 24 MAX_COST = 5 # 同一维度间的最大差异 25 26 def cost_stu(stu_1, stu_2): 27 ''' 以stu_1为主,计算stu_1与stu_2的差异 ''' 28 cost = [] # 各维度的成本值(差异度) 29 cost.append(cost_equal(stu_1[0], stu_2[0])) # 籍贯成本 30 cost.append(cost_equal(stu_1[1], stu_2[1])) # 专业成本 31 cost.append(cost_class(stu_1[2], stu_2[2], stu_1[1], stu_2[1])) # 班级成本 32 cost.append(cost_get_up(stu_1[3], stu_2[3])) # 起床成本 33 cost.append(cost_interest(stu_1[4], stu_2[4])) # 爱好成本 34 w_idx_1, w_idx_2 = stu_1[len(stu_1) - 1] - 1, stu_2[len(stu_2) - 1] - 1 # 权重序号 35 w_cost_1, w_cost_2 = cost[w_idx_1] * 1.5, cost[w_idx_2] * 1.5 # 加权处理 36 # 判断二者最在意的是否相同 37 if w_idx_1 == w_idx_1: 38 cost[w_idx_1] = w_cost_1 + w_cost_2 39 else: 40 cost[w_idx_1], cost[w_idx_2] = w_cost_1, w_cost_2 41 42 return sum(cost) 43 44 def cost_equal(d_1, d_2): 45 ''' 同质化比较成本 ''' 46 return 0 if d_1 == d_2 else MAX_COST 47 48 def cost_class(d_1, d_2, sub_1, sub_2): 49 ''' 班级成本 ''' 50 if d_1 == d_1: # 班级相同 51 return 0 52 elif sub_1 == sub_1: # 不同班级,同一专业 53 return 1 54 else: # 不同班级,不同专业 55 return MAX_COST 56 57 def cost_get_up(d_1, d_2): 58 ''' 起床成本 ''' 59 return 1.2 * (d_2 - d_1) 60 61 def cost_interest(d_1, d_2): 62 ''' 爱好成本 ''' 63 for t_1 in d_1: 64 # 如果两个同学都有一个共同的爱好,二者就是零距离 65 if t_1 in d_2: 66 return 0 67 68 obj_1 = t_1 // 10 # 爱好的“大类” 69 # 如果两个同学都有一个共同的大类,二者距离是2 70 for t_2 in d_2: 71 if obj_1 == t_2 // 10: 72 return 2 73 return MAX_COST 74 75 def cost_fun(solution): 76 ''' 77 计算方案中每个宿舍的成本 78 :param solution: 宿舍分配方案 79 :return: 宿舍总成本和每个宿舍的成本 80 ''' 81 droms_cost = [] 82 for drom in solution: 83 # 同一宿舍中的四个同学两两比对 84 d_cost = 0 85 d_cost += cost_stu(STUDENTS[drom[0]], STUDENTS[drom[1]]) 86 d_cost += cost_stu(STUDENTS[drom[0]], STUDENTS[drom[2]]) 87 d_cost += cost_stu(STUDENTS[drom[0]], STUDENTS[drom[3]]) 88 d_cost += cost_stu(STUDENTS[drom[1]], STUDENTS[drom[2]]) 89 d_cost += cost_stu(STUDENTS[drom[1]], STUDENTS[drom[3]]) 90 d_cost += cost_stu(STUDENTS[drom[2]], STUDENTS[drom[3]]) 91 droms_cost.append(d_cost) 92 diff_cost = max(droms_cost) - min(droms_cost) # 宿舍之间的贫富差 93 total_cost = sum(droms_cost) + diff_cost * DROM_SIZE # 该方案的总成本 94 return total_cost, droms_cost 95 96 def upset(): 97 ''' 打乱学生顺序 ''' 98 n = len(STUDENTS_NAME) 99 stu_list = list(range(n)) 100 # 打乱学生顺序 101 for i in range(n): 102 rand_idx = random.randint(0, n - 1) 103 stu_list[i], stu_list[rand_idx] = stu_list[rand_idx], stu_list[i] 104 return stu_list 105 106 def print_solution(solution, total_cost, dorms_cost): 107 for i, drom in enumerate(solution): 108 print('宿舍%d:\t' % i, end='') 109 for j in drom: 110 print('%-8s' % STUDENTS_NAME[j], end='') 111 print('\tcost=%f' % dorms_cost[i]) 112 print('total=%f' % total_cost)
genetic_optimize.py
1 from __future__ import division 2 import random 3 import os 4 import sys 5 parent_dir_name = os.path.dirname(os.path.realpath(__file__)) 6 sys.path.append(parent_dir_name ) 7 import base_data 8 9 POPULATION_SIZE = 1000 # 种群数量 10 11 def init_population(): 12 ''' 构造初始种群 ''' 13 population = [] 14 code_len = len(base_data.STUDENTS_NAME) # 编码长度 15 for i in range(POPULATION_SIZE): 16 population.append(base_data.upset()) 17 return population 18 19 def solution_adapter(code): 20 ''' 将基因编码翻适配成cost_fun能够识别的解 ''' 21 solution = [] 22 for i in range(0, len(base_data.STUDENTS_NAME), base_data.NUM_PER_DROM): 23 solution.append(code[i:i + base_data.NUM_PER_DROM]) 24 return solution 25 26 def fitness_fun(code): 27 ''' 28 适应度评估 29 :param code: 二进制基因编码 30 :return: 适应度评估值, 二元组, (宿舍总成本, 每个宿舍的成本) 31 ''' 32 return base_data.cost_fun(solution_adapter(code)) 33 34 def selection(population): 35 '''选择策略, 锦标赛法''' 36 pop_next = [] # 下一代种群 37 for i in range(POPULATION_SIZE): 38 tour_list = random.choices(population, k=2) # 二元锦标赛 39 winner = min(tour_list, key=lambda x: fitness_fun(x)) # 成本低的胜出 40 pop_next.append(winner) 41 return pop_next 42 43 def crossover_pmc(population): 44 ''' 部分匹配交叉(PMC)''' 45 46 def create_mapping(cross_code_1, cross_code_2): 47 ''' 48 建立两个交叉片段间的映射关系 49 :param cross_code_1: 50 :param cross_code_2: 51 :return: 映射关系set 52 ''' 53 mapping = set() 54 for i in range(len(cross_code_1)): 55 c1, c2 = cross_code_1[i], cross_code_2[i] 56 if (c1, c2) not in mapping and (c2, c1) not in mapping: 57 mapping.add((c1, c2)) 58 return mapping 59 60 def code_extends(child, parent, start, end): 61 ''' 62 继承父代的编码 63 :param child: 子代个体 64 :param parent: 父代个体 65 :param start: 基因编码起始位置 66 :param end: 基因编码终止位置 67 ''' 68 for i in range(start, end): 69 if parent[i] not in child: 70 child[i] = parent[i] 71 72 def code_rest(child, cross_code): 73 ''' 74 通交叉前的基因片段修改子代的编码 75 :param child: 子代个体 76 :param cross_code: 交叉前的的基因片段 77 :return: 78 ''' 79 for i, x in enumerate(child): 80 if x != -1: 81 continue 82 for x_old in cross_code: 83 if x_old not in child: 84 child[i] = x_old 85 break 86 87 pop_new = [] # 新种群 88 code_len = len(population[0]) # 基因编码的长度 89 for i in range(POPULATION_SIZE): 90 # 选择两个随机的交叉点 91 p1, p2 = random.randint(0, code_len - 1), random.randint(0, code_len - 1) 92 if p1 > p2: 93 p1, p2 = p2, p1 94 parent1, parent2 = random.choices(population, k=2) # 选择两个随机的个体 95 cross_code_1 = parent1[p1:p2] # 交叉前的编码块 96 cross_code_2 = parent2[p1:p2] # 交叉后的编码块 97 # 构造新的个体,-1表示基因编码尚未确定 98 r = [-1] * p1 + cross_code_2 + [-1] * (code_len - p2) 99 code_extends(r, parent1, 0, p1) # 继承父代的编码 100 code_extends(r, parent1, p2, code_len) # 继承父代的编码 101 mapping = create_mapping(cross_code_1, cross_code_2) # 两个交叉块的映射关系 102 code_rest(r, cross_code_1) # 通过交换前的基因片段确定剩余编码 103 pop_new.append(r) 104 return pop_new 105 106 def crossover_cx(population): 107 ''' 循环交叉匹配 ''' 108 pop_new = [] # 新种群 109 code_len = len(population[0]) # 基因编码的长度 110 for i in range(POPULATION_SIZE): 111 parent1, parent2 = random.choices(population, k=2) # 选择两个随机的个体 112 r_new = [-1] * code_len # 新个体 113 r_new[0] = parent1[0] 114 i = 0 115 while True: # 循环交叉 116 x = parent2[i] 117 if r_new[0] == x: 118 break 119 i = parent1.index(x) 120 r_new[i] = x 121 # r_new中剩余未确定的编码直接从parent2中继承 122 for i, x in enumerate(r_new): 123 if x == -1: 124 r_new[i] = parent2[i] 125 pop_new.append(r_new) 126 return pop_new 127 128 def mutation(population): 129 ''' 变异 ''' 130 code_len = len(population[0]) # 基因编码的长度 131 mp = 0.2 # 变异率 132 for i, r in enumerate(population): 133 if random.random() < mp: 134 # 两个随机变异点 135 p1, p2 = random.randint(0, code_len - 1), random.randint(0, code_len - 1) 136 # 交换两个变异点的数据 137 population[p1], population[p2] = population[p2], population[p1] 138 139 def sum_fitness(population): 140 ''' 计算种群的总适应度 ''' 141 return sum([fitness_fun(code)[0] for code in population]) 142 143 def ga(): 144 ''' 遗传算法分配宿舍 ''' 145 population = init_population() # 构建初始化种群 146 s_fitness = sum_fitness(population) # 种群的总适应度 147 i = 0 148 while i < 10: # 如果连续10代没有改进,结束算法 149 pop_next = selection(population) # 选择种群 150 pop_new = crossover_cx(pop_next) # 交叉 151 mutation(pop_new) # 变异 152 s_fitness_new = sum_fitness(pop_new) # 新种群的总适应度 153 if s_fitness > s_fitness_new: # 成本越低,适应度越高 154 s_fitness = s_fitness_new 155 i = 0 156 else: 157 i += 1 158 population = pop_new 159 # 按适应度值从大到小排序 160 population = sorted(population, key=lambda x: fitness_fun(x), reverse=True) 161 # 返回最优的个体 162 return population[0] 163 164 if __name__ == '__main__': 165 best = ga() 166 solution = solution_adapter(best) 167 total_cost, dorms_cost = base_data.cost_fun(solution) 168 base_data.print_solution(solution, total_cost, dorms_cost)
作者:我是8位的



 浙公网安备 33010602011771号
浙公网安备 33010602011771号