用于增强抗癌肽特征提取的对比学习
用于增强抗癌肽特征提取的对比学习
李炳卓(Byungjo Lee)\({}^{1}\) 和申东宽(Dongkwan Shin)\({}^{1,2, * }\)
\({}^{1}\) 韩国国立癌症中心研究所,高阳11산동구11산로32号,邮编10408
\({}^{2}\) 韩国国立癌症中心癌症科学与政策研究生院癌症生物医学科学系,高阳11산동구11산로323号,邮编10408
*通讯作者。申东宽,韩国国立癌症中心研究所,高阳10408。电话:+82 - 31 - 920 - 2433;电子邮箱:dshin@ncc.re.kr
摘要
癌症是全球公认的主要死因,对健康有着深远影响,并带来巨大的社会负担。人们为开发癌症治疗方法付出了诸多努力,其中抗癌肽(ACPs)因其潜在应用而受到认可。虽然抗癌肽筛选既耗时又昂贵,但计算机预测工具提供了克服这些挑战的途径。在此,我们提出一种仅使用肽序列筛选抗癌肽的深度学习模型。应用对比学习技术来提高模型性能,其产生的结果优于仅基于二元分类损失训练的模型。此外,使用两个独立编码器替代对比学习中常用的数据增强技术。在六个基准数据集中的五个上,我们的模型相对于先前的最先进模型表现出卓越性能。随着预测工具的进步,基于肽的癌症治疗潜力增加,为肿瘤学研究和患者护理带来更光明的未来。
关键词:抗癌肽;深度学习;对比学习;治疗筛选
引言
癌症是全球主要死因,对健康有严重影响,并给社会带来巨大负担[1]。2020年,全球约出现1930万例新癌症病例和1000万例癌症相关死亡[2]。尽管全球都在努力,但由于与经济和社会发展相关的风险因素不断演变,癌症负担或死亡率仍在上升[3,4]。因此,建立合适的医疗保健系统以进行早期检测和有效治疗对于降低与癌症相关的社会成本至关重要。
由于肽合成技术的进步,新型基于肽的治疗药物的开发正在显著加速[5]。肽治疗药物因其独特性质,如高选择性、灵活的骨架和增强的渗透性,可作为小分子的有效替代品[6]。通过利用这些有益特性,抗癌肽(ACPs)已被开发用于治疗各种癌症,并在市场上成功崭露头角[7,8]。然而,开发肽药物仍然既耗时又耗费人力和资源。为应对这些挑战,基于计算机的方法可以加速有前景的抗癌肽的发现[9]。
研究人员最近开发了各种计算机预测工具,以促进治疗肽的发现[10 - 13]。为模型训练获取合适的数据集需要进行生物学实验,而这种内在复杂性限制了肽数据集的可用性。因此,基于肽的预测模型使用有限数量的样本进行训练,从而限制了预测性能。因此,人们采取了多种方法通过预处理或不同的学习策略从肽数据中提取突出特征[14 - 16]。关于抗癌肽,各种模型利用传统机器学习或深度学习模型,并在数据预处理期间提出了新的模型结构或特征工程方法[15,17 - 28]。
为提高预测性能,对比学习可用于特征表示学习,这是深度学习中广泛使用的一种技术,以增强这些方法的效果[29]。该技术采用“相似”样本之间的接近度,并增加嵌入空间中“不相似”样本之间的距离[30]。对比学习通过比较批次或数据集中的样本,有助于捕获和提取有价值的潜在特征表示。为利用对比学习,数据增强技术建立“相似”和“不相似”样本:“相似”样本是来自同一原始来源的成对增强实例,而“不相似”样本来自不同的原始来源[29]。该技术在语言、视觉和音频领域显著提高了模型性能[31 - 34]。然而,增强肽或蛋白质序列(这对对比学习至关重要)仍然是一个挑战,需要一种克服这一挑战的方法来应用对比学习[35]。
李炳卓是韩国国立癌症中心研究所的博士后研究员。他的研究兴趣集中在人工智能驱动的药物靶点发现。
申东宽是韩国国立癌症中心研究所的高级研究员。他的研究兴趣集中在癌症系统生物学领域。收到日期:2024年1月8日。修订日期:2024年3月28日。接受日期:2024年4月21日
© 作者(s) 2024。由牛津大学出版社出版。
这是一篇开放获取文章,根据知识共享署名许可协议(https://creativecommons.org/licenses/by/4.0/)分发,允许在任何媒介中进行无限制的再利用、分发和复制,前提是对原始作品进行适当引用。
在本研究中,我们开发了一种仅使用肽序列筛选抗癌肽(ACP)的精确工具。为了提高模型预测性能,同时实施了对比损失和交叉熵损失。我们的方法结合使用两个不同的编码器和两个不同的分词器,实现对比学习。这种新颖的设置允许相同的肽序列通过各自的分词器转换为两个独特的索引表示,增强了模型从数据中捕获和学习潜在特征表示的能力。这些编码器的输出分别转换为用于分类的分类概率向量和用于对比损失的投影向量。通过调整交叉熵损失和对比损失之间比例的实验,我们的方法在六个基准数据集中的五个上表现优于最新模型。
方法
基准数据集
为了与先前开发的模型进行性能基准测试,我们选择了多个预测模型中使用过的各种数据集。在用于预测抗癌肽的基准数据集中,我们将模型训练集中在由He等人整理的六个基准数据集上[15, 24, 27, 36, 37]。表S1展示了这六个基准数据集的统计信息。这些数据集预先分为模型训练和独立测试部分。因此,在训练数据集内、测试数据集内以及训练和测试数据集之间评估了序列冗余性。通过在每个训练和测试数据集内部及之间使用基本局部比对搜索工具(BLAST)进行同源性搜索来获得序列冗余性,以确定每个数据集的可靠性[38]。在每个序列中,如果查询序列与数据库中的任何序列(不包括序列本身)相比,其e值为1E - 5或更低,则该查询序列被视为冗余序列。在此背景下,对于训练或测试数据集内的冗余性,比较是在查询序列和作为数据库的相同数据集中进行。相反,在评估训练和测试数据集之间的冗余性时,比较结构是一个数据集作为查询,另一个作为数据库。
序列表示
肽由一系列氨基酸表示。有21种独特的氨基酸,包括用字母'X'表示的未指定氨基酸。分词器将氨基酸序列转换为输入索引,以便将肽数据输入模型。有两个分词器:一个将单个氨基酸转换为单个索引,另一个将氨基酸对转换为一个索引。第一个分词器的范围是0到22,它将单个氨基酸转换为一个包括序列起始令牌和填充令牌的单个索引。第二个分词器将两个氨基酸映射为一个范围从0到442的单个索引,其中也包括序列起始令牌和填充令牌。通过这两个分词器,相同的肽序列被转换为两个不同的索引序列,从而产生数据增强效果。由两个分词器转换的索引准备好输入到各自的嵌入层,嵌入层将离散的索引向量转换为更压缩的向量。
模型架构
为了进行对比学习,开发了两个独立的编码器。每个编码器层被配置为从嵌入层的输出中提取特征表示向量,其中各自的分词器将一个或两个氨基酸长度的令牌转换为相应的索引。当使用单个氨基酸长度的令牌时,由每个分词器编码的向量输入到嵌入1中,当使用两个令牌时输入到嵌入2中。嵌入后的向量被输入到各自的编码器层1和2中,输出用于分类和特征投影。线性头确定分类结果,而投影层进行特征投影。
卷积神经网络(Convolutional neural networks,CNNs)在分析空间数据方面已取得显著成功,而长短期记忆(long short - term memory,LSTM)网络则专门用于处理序列数据,能够有效地从序列中的过去和当前状态捕获信息[39]。此外,Transformer编码器架构以其自注意力机制有效捕获自然语言数据中的长期依赖关系而闻名[40]。这三种架构各有优点,并且已经在先前的研究中用于预测功能肽,取得了性能提升[13, 16, 41 - 43]。因此,实现这三种架构作为骨干架构,以基于编码器结构评估模型性能。编码器生成的特征向量被输入到线性头和投影仪中,用于计算交叉熵损失(CE)和对比损失。详细的模型信息见表S4。
模型训练
为了提高分类性能,交叉熵损失的计算如下:
其中,\(i\)是所选样本的索引,\({y}_{i,1}\)是真实类别标签,\({\widehat{y}}_{i,1}\)和 \({\widehat{y}}_{i,2}\) 分别表示使用不同分词器获得的线性头1和线性头2的预测结果。每个线性头的CE损失的权重比可以通过调整 \(\beta\) 系数来调节。当 \(\beta\) 为0时,仅使用线性头1的预测结果来计算CE损失。当 \(\beta\) 为0.5时,线性头1和2对CE损失计算的贡献相等。最后,\(\beta\) 值为1时仅考虑线性头2。
为了有效提取潜在表示,对比损失的推导如下:
其中,\(\tau\)是一个温度参数,\({\mathbf{z}}_{i,1}\)和 \({\mathbf{z}}_{i,2}\) 分别是线性头1和头2的输出。对比损失基于线性头1和2的投影向量计算,这两个线性头为模型接收不同的分词器输入,而不是增强样本。对比损失未用于预训练。相反,交叉熵损失和对比损失同时用于模型训练。最终损失由下式给出:
其中,\(\alpha\)系数在确定最终损失时控制交叉熵损失和对比损失之间的比例。
关于用于模型性能验证的数据集选择,在基准数据集中已经预先定义了一个独立测试集。对于模型训练期间的验证集,我们分配了训练数据集的10%,其余90%用于模型训练。训练 - 验证分割在每个训练过程中执行,分割过程由指定的随机种子输入控制。因此,数据分割在相同的输入随机种子下始终一致地复制,确保训练过程的可重复性和一致性。
模型训练使用网格搜索方法来优化随机种子、α、β和温度等几个参数。对于学习率,选择1e - 3,因为它在我们的训练案例中显示出稳定的训练效果。详细的训练信息见表S5。模型训练过程使用PyTorch [44]实现。实验在一台工作站上执行,该工作站具有AMD Ryzen Threadripper PRO 5955WX中央处理器、GeForce RTX 4090图形处理器和256GB随机存取存储器。
评估指标
基于准确率、灵敏度、特异性、马修斯相关系数(Matthews correlation coefficient,MCC)、接收器操作特征(receiver operating characteristic,ROC)和ROC曲线下面积(area under the ROC curve,AUC)评估模型性能。定义如下:
其中,TP是真阳性,TN是真阴性,FP是假阳性,FN是假阴性。
模型解释
应用可解释人工智能技术来识别模型中主要用于预测ACP的氨基酸残基。在各种技术中,使用Captum库[45, 46]应用了集成梯度方法(一种基于归因的方法)。在ACP - Mixed - 80测试数据集中,提供了正类数据并用于计算嵌入层的归因分数。然后通过计算每个氨基酸残基的平均值来汇总这些归因分数。
结果
基准数据集分析
为了有效地训练和评估分类模型,考虑数据集中样本的组成至关重要。已经开发了许多用于对ACP进行分类的模型和各种基准数据集。为了评估各个数据集对模型训练和评估的适用性,使用BLAST审查了序列冗余,BLAST可识别生物序列之间的序列相似性[38]。在训练集(训练集到训练集)、测试集(测试集到测试集)以及训练集和测试集之间(训练集到测试集以及反之亦然)进行了BLAST。在BLAST结果中,e值低于1E-5被认为是在序列之间表现出高相似性的同源序列。在上述比较组中验证了这些同源序列比率(表1)。
对于模型训练(训练集到训练集),ACP2.0替代方案显示出最高的冗余度,而ACP-Mixed-80在训练序列中表现出最低的冗余度。关于模型评估,在每个数据集中,超过20%的测试序列与训练数据集(测试集到训练集)呈现出显著的同源性,但ACP-Mixed-80除外。特别是,ACP2.0主数据集、ACP2.0替代数据集和LEE + 独立数据集的比率超过了40%。除了ACP-Mixed-80,超过10%的测试样本在相同的测试数据集中包含显著相似的序列(测试集到测试集)。测试数据集内或测试集与训练集之间的高样本重叠率可能会扭曲泛化性能的评估,可能导致对模型性能的过度乐观评估。因此,结果将主要集中在ACP-Mixed-80上,因为它在所有情况下始终表现出最低的序列冗余度。
对比学习的模型设计
为了提高ACP的预测性能,进行了对比学习,这有助于从有限的肽序列中提取潜在表示。在计算图像数据集中的对比损失时,对输入样本进行增强以提供正样本对和负样本对。然后计算对比损失以确保正样本对的特征向量相似而负样本对的特征向量不同。这个过程从数据中提取额外的特征。然而,与图像数据集不同,由于难以确定增强的氨基酸序列是否具有抗癌活性,基于氨基酸序列的数据集进行数据增强具有挑战性。
我们通过开发两个具有相同架构的不同模型来解决这个问题,以提供正数据集对和负数据集对,每个模型对相同的输入肽序列使用不同的分词器(图1)。第一个分词器使用单长度氨基酸词汇表处理氨基酸序列,而第二个分词器使用双长度氨基酸进行分词。每个分词器将相同的输入序列转换为索引,然后将其输入到每个编码器的各自嵌入层中。每个编码器层将嵌入层输出转换为表示向量,投影头将表示向量转换为特征向量。来自相同序列的特征向量用作正样本对,而来自不同序列的特征向量形成负样本对以计算对比损失。
为了训练用于对ACP进行分类的模型,同时进行了CE损失和对比损失的组合。在我们的方法中,两个模型的每个线性头都用于分类目的,从而产生两个不同的CE损失值。系数β对于确定使用单长度或双长度氨基酸令牌的任何一个模型的CE损失在训练过程中的考虑程度至关重要。具体而言,当系数β值为0或1时,模型训练分别仅依赖于模型1或模型2的CE损失。相反,当β设置为0.5时,在模型训练期间平等地使用两个模型的CE损失。此外,引入系数α来控制CE损失和对比损失分量之间的平衡。α系数用于在训练期间调整CE损失和对比损失的权重,从而可以灵活调整它们对学习过程的贡献。当α系数设置为0时,对比学习被排除在模型训练之外。此方法用作比较使用对比学习时模型性能的基线。为了识别具有最佳性能的模型,我们采用网格搜索策略,系统地将α调整为0.1、0.3、0.5、0.7和0.9的值,将β调整为0、0.5和1的值,同时调整这两个参数。这种方法使我们能够探索广泛的配置范围,以确定提高模型预测准确性的最有效组合。
表1:BLAST搜索结果中各数据集中显著同源肽序列的比例
| 数据集 | 训练到训练 | 训练到测试 | 测试到训练 | 测试到测试 |
| ACP混合-80 | 0.1983 | 0.1116 | 0.1803 | 0.0656 |
| ACP2.0主版本 | 0.3120 | 0.2351 | 0.4012 | 0.2384 |
| ACP2.0替代版本 | 0.3492 | 0.2448 | 0.4124 | 0.2577 |
| 李 + 独立 | 0.2275 | 0.2299 | 0.4733 | 0.1400 |
| ACP500 + ACP2710 | 0.2560 | 0.1560 | 0.2561 | 0.1707 |
| ACP500 + ACP164 | 0.2760 | 0.1640 | 0.2683 | 0.2073 |
加粗数字表示显著同源肽序列的最低比例。
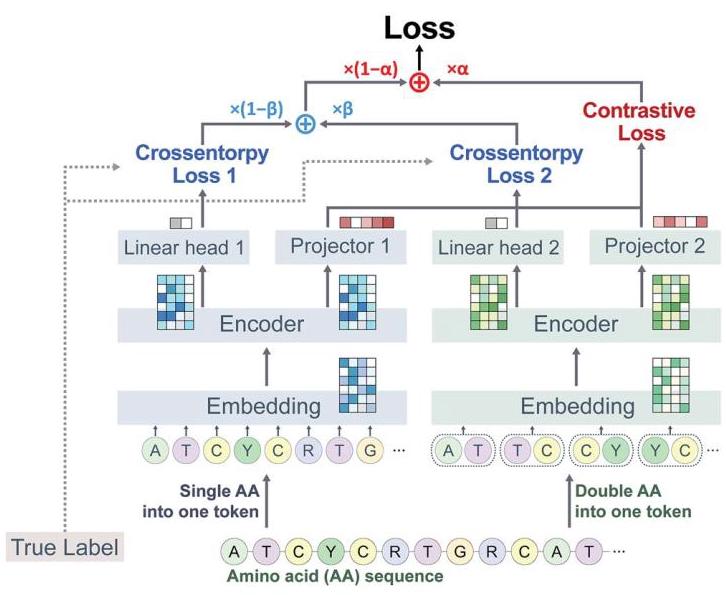
图1. 用于抗癌肽分类的对比学习概述。每个编码器接收由其相应的分词器转换的氨基酸序列,该分词器使用单氨基酸或双氨基酸词汇表。线性头1和2提供分类概率,而投影仪1和2为对比学习制定特征向量。系数α表示对比损失的反映程度;系数β表示每个编码器预测的分类中CE损失的比例。
对比学习增强预测性能
最初,我们评估了温度系数对预测性能的影响,温度系数是对比学习计算中的一个参数(图S1 - S6)。除了ACP500 + ACP2710和ACP - Mixed - 80数据集外,MCC指标的中位数随着温度系数的增加呈上升趋势。然而,对于ACP500 + ACP2710和ACP - Mixed - 80数据集,模型性能在温度系数为0.2时达到最佳,超过该值则未观察到进一步改善。
为了研究系数α对模型性能的影响,在ACP - Mixed - 80数据集上对各个系数β和预测分类器的预测结果进行了平均(图2)。误差条表示模型性能,是在七个随机种子和五个系数温度的训练会话中汇总得出的。当使用CNN实现编码器时,分类器1(线性头1的输出,使用单长度氨基酸词汇表的分词器1)在准确率、灵敏度和MCC方面表现优于分类器2(线性头2的输出,使用双长度氨基酸词汇表的分词器2)(图2A)。相反,分类器2始终表现出更高的特异性。在Transformer编码器架构中的结果与CNN架构的结果非常相似。相比之下,随着α的增加,分类器2的灵敏度下降与特异性增加呈负相关(图2B)。对于LSTM,随着α的增加,准确率、MCC和灵敏度下降,而分类器1和2的特异性增加(图2C)。在评估正样本和负样本时,平均而言,分类器1在准确率和MCC方面优于分类器2。在编码器架构顺序为CNN、Transformer编码器和LSTM的情况下,这些差异有所减小。对于其他数据集,在大多数性能指标上,分类器1始终与分类器2相当或优于分类器2,但在Transformer编码器架构下的LEE + 独立数据集除外(图S7 - S11)。总体而言,无论系数α如何,分类器1的表现都优于分类器2。
为了研究通过对比学习实现的性能提升,在ACP - Mixed - 80数据集上对对比学习和排除对比损失的基线模型进行了性能比较(图3)。散点图上的每个点表示在相同模型架构和初始模型权重的情况下,仅使用CE训练模型(基线,在x轴上表示)与对比学习(在y轴上表示)时的模型性能。散点主要位于红线之上,这表明对比学习通常比基线模型在性能指标上有更好的表现。在准确率、灵敏度、特异性和MCC的所有性能指标上都观察到了这种提升。
同样,其他数据集的结果在每个指标上的散点大多也位于线上方,但ACP500 + ACP2710数据集的灵敏度除外(图S12)。在保持相同模型架构和初始模型权重的情况下,对比学习相对于基线模型提高了性能(表S2)。在处理ACP - Mixed - 80数据集时,几乎所有模型通过对比学习在准确率和MCC方面相对于基线模型都有性能提升。此外,超过一半的模型在灵敏度和特异性方面表现出性能增强,无论编码器架构如何。在其他数据集中,ACP500 + ACP2710的灵敏度受到对比学习的不利影响;对于每种编码器架构,只有约30%的模型性能得到提升。然而,对比学习在其他指标上始终能提高模型性能。在所有其他情况下,无论模型编码器架构如何,对比学习在超过一半的模型中提高了性能。与基线相比,对比学习在所有数据集中的准确率和MCC(整体模型性能的主要指标)方面往往能提高模型性能。
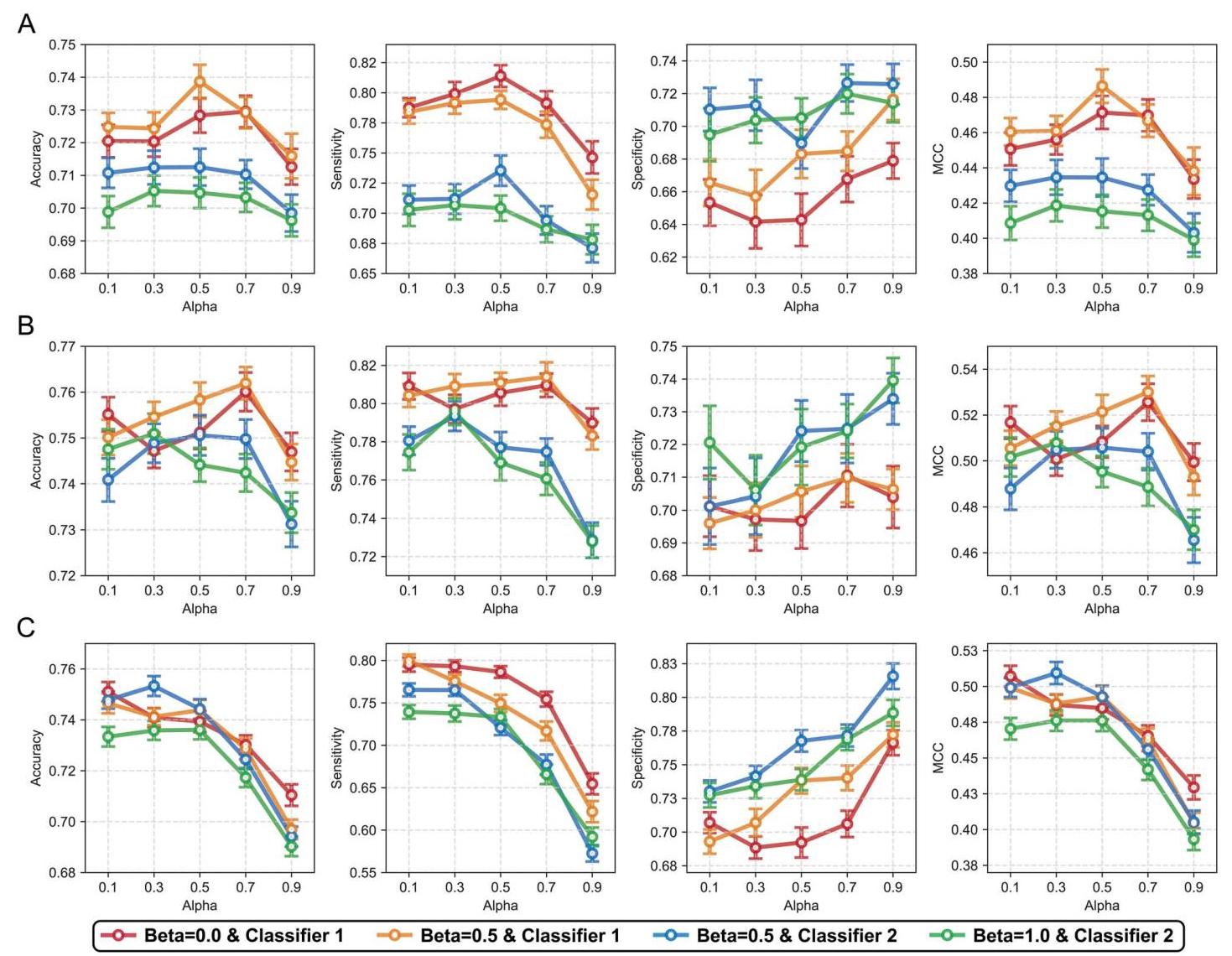
图2. 使用ACP - Mixed - 80数据集相对于对比学习超参数的性能比较。每个点表示相对于系数α通过对比学习训练的每个性能指标的平均值。结果绘制在与系数β和分类器类型对应的每条线上。误差条表示均值的标准误差。结果按(A)CNN、(B)Transformer编码器和(C)LSTM的编码器架构显示。
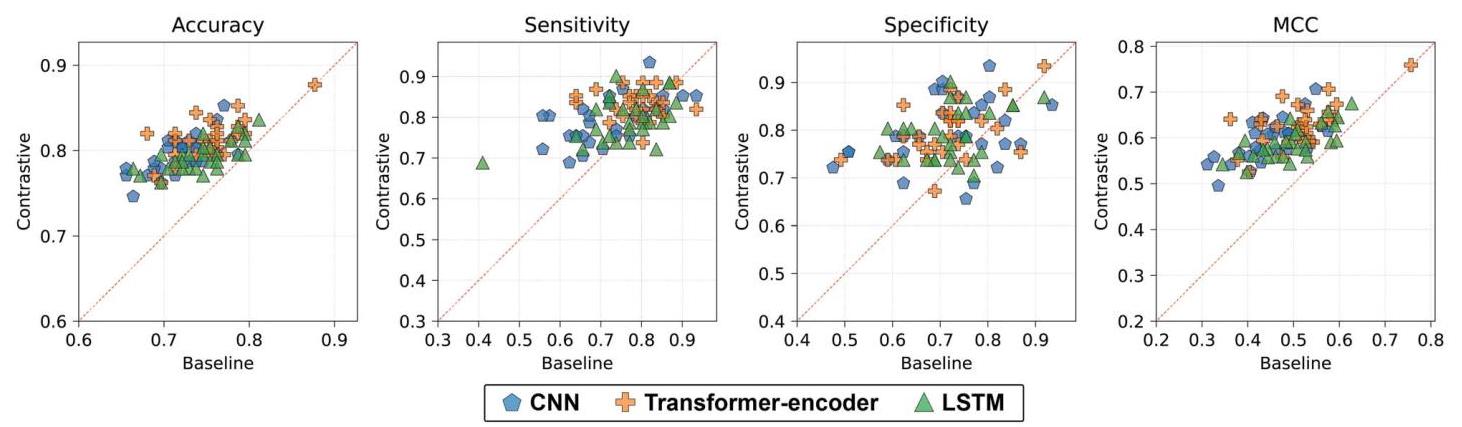
图3. 使用具有相同架构和初始权重的ACP-Mixed-80数据集,对不同训练方法的性能比较。每个散点表示通过交叉熵(CE,基线)损失和对比学习训练的模型性能,分别在各自的x轴和y轴上显示性能。虚线表示基线和对比学习之间的等效性能。对于相同的模型架构和初始模型权重,线以上的区域表示对比学习具有卓越性能,线以下的区域表示基线具有更好的性能。
与现有技术的性能比较
为了评估相对于现有技术(SOTA)模型的性能提升,我们根据alpha和beta系数,针对SOTA模型[14, 15, 17, 18, 23, 25, 27, 36, 37, 47 - 52]分析了模型性能。在每个编码器架构、alpha系数和beta系数内,选择具有最高马修斯相关系数(MCC)的模型,使用ACP-Mixed-80数据集的这些选定模型的性能如图4所示。当alpha设置为0.9、beta设置为0.5且使用分类器1时,使用卷积神经网络(CNN)编码器的模型在准确率、特异性和MCC方面超过了SOTA(图4A)。当仅使用交叉熵(CE)损失训练模型时,该模型在灵敏度方面优于SOTA,而SOTA在其他指标上表现出色。关于Transformer编码器,当参数设置为alpha为0.1、0.3、0.5、0.7和0.9;beta为0.5;并使用分类器2时,该模型在每个性能指标上均达到了SOTA(图4B)。虽然仅使用交叉熵(CE)时超过了SOTA性能,但采用对比学习在alpha为0.1、beta为0.5且使用分类器2的参数下,在准确率、MCC和特异性方面产生了更好的结果。图S13 - S17展示了使用其他数据集进行模型训练时的结果。虽然我们提出的模型在AntiCP2.0主数据集中的灵敏度方面超过了SOTA,但SOTA在准确率、特异性和MCC方面表现更好(图S13)。当使用其他数据集进行评估时,与SOTA基准相比,对比学习始终提高了模型指标(图S14 - S17)。
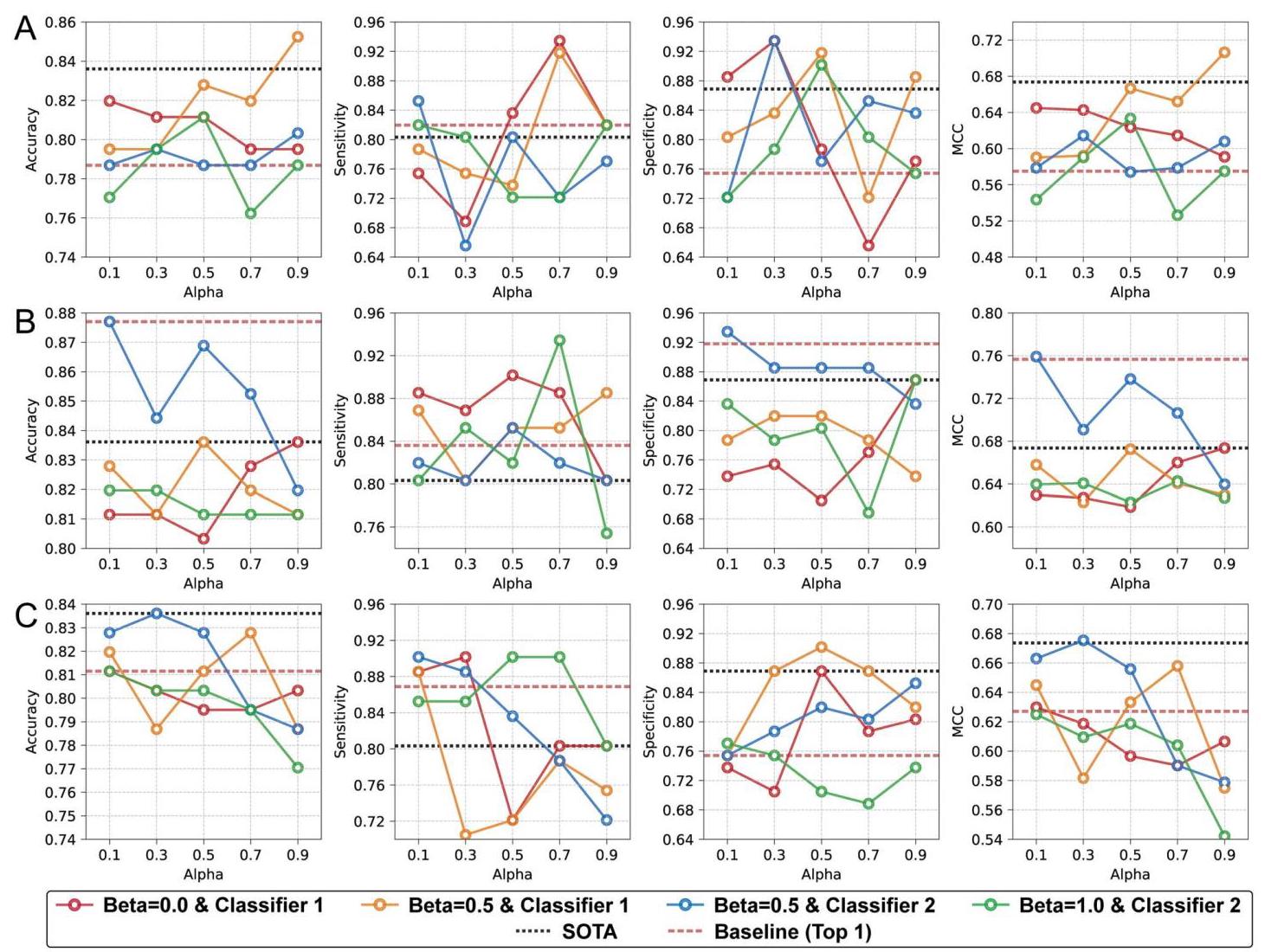
图4. 使用ACP-Mixed-80数据集,相对于每个系数alpha和beta的最佳模型性能。基于实现最高MCC指标值的alpha和beta系数选择最佳模型。针对每个beta系数和每个alpha系数的预测分类器绘制线图。虚线表示基线的SOTA和最佳性能。结果基于(A)CNN、(B)Transformer编码器和(C)长短期记忆网络(LSTM)的编码器架构显示。
为了选择优化模型,我们根据测试数据集上的最高MCC指标值,为每个编码器架构从对比学习和基线模型中进行选择(表2)。在ACP-Mixed-80的情况下,当通过对比学习训练时,Transformer编码器实现了最高的MCC指标值0.7591。alpha设置为0的基线设置紧随其后,MCC值为0.7566,排名第二。这两种配置在所有指标上均超过了SOTA,并且相对于其他编码器架构(不包括灵敏度)表现出卓越的性能。虽然长短期记忆网络(LSTM)表现出第一和第二高的灵敏度值,但其降低的特异性指标导致准确率和MCC指标值相对较低。对于其他数据集,与基线相比,使用对比学习训练的模型获得了最高的MCC指标值,超过了SOTA的MCC值,但不包括AntiCP2.0主数据集。总体而言,除了AntiCP2.0主数据集中的CNN外,对比学习在相同的编码器架构内提高了特异性,而这种权衡降低了灵敏度。表S3中有详细的比较结果。
最后,我们将表现出最高MCC指标值的模型视为数据集的优化模型。图S18展示了每个数据集优化模型的ROC曲线。这些模型在ACP-Mixed-80、AntiCP2.0主数据集、AntiCP2.0替代数据集、ACP500 + ACP164、ACP500 + ACP2710和LEE + 独立数据集上分别获得了曲线下面积值0.9366、0.8377、0.9678、0.9453、0.7941和0.9822。
模型解释
为了评估特定氨基酸残基在从输入数据中对酰基载体蛋白(ACP)进行分类时的重要性,我们通过集成梯度法进行了系统的模型解释。这种基于归因的可解释人工智能通过计算嵌入层内的归因来识别对预测有主要贡献的氨基酸。该方法提供了归因分数,表明单个氨基酸如何影响模型预测。我们使用ACP-Mixed-80测试数据集,通过优化模型来识别对ACP预测影响最大的氨基酸残基(图5A)。根据结果,赖氨酸(Lys,K)、色氨酸(Trp,W)和亮氨酸(Leu,L)对优化模型准确预测ACP有显著贡献;天冬氨酸(Asp,D)、丝氨酸(Ser,S)和酪氨酸(Tyr,Y)的归因分数较低。
表2:各模型架构、训练策略和预训练最优模型(pre-SOTA)的优化模型性能结果
| 数据集 | 编码器 | 阿尔法 | 贝塔 | AA令牌长度 | 准确率 | 灵敏度 | 特异性 | 马修斯相关系数 |
| ACP混合-80 | 当前最优方法 [36] | - | - | - | 0.8361 | 0.8033 | 0.8689 | 0.6736 |
| 卷积神经网络 | 0 | 0 | 1 | 0.7869 | 0.8197 | 0.7541 | 0.5750 | |
| 0.9 | 0.5 | 1 | 0.8525 | 0.8197 | 0.8852 | 0.7064 | ||
| Transformer- | 0 | 1 | 2 | 0.8770 | 0.8361 | 0.9180 | 0.7566 | |
| 编码器 | 0.1 | 0.5 | 2 | 0.8770 | 0.8197 | 0.9344 | 0.7591 | |
| 长短期记忆网络 | 0 | 1 | 2 | 0.8115 | 0.8689 | 0.7541 | 0.6271 | |
| 0.3 | 0.5 | 2 | 0.8361 | 0.8852 | 0.7869 | 0.6754 | ||
| 反CP 2.0主版本 | 当前最优方法 [50] | - | - | - | 0.8250 | 0.7260 | 0.9030 | 0.6460 |
| 卷积神经网络 | 0 | 0 | 1 | 0.7558 | 0.7326 | 0.7791 | 0.5122 | |
| 0.1 | 0.5 | 1 | 0.7791 | 0.8023 | 0.7558 | 0.5587 | ||
| Transformer- | 0 | 0 | 1 | 0.7645 | 0.8721 | 0.6570 | 0.5418 | |
| 编码器 | 0.1 | 0.5 | 2 | 0.8081 | 0.8023 | 0.8140 | 0.6163 | |
| 长短期记忆网络 | 0 | 1 | 2 | 0.7529 | 0.7209 | 0.7849 | 0.5069 | |
| 0.1 | 0.5 | 2 | 0.7587 | 0.6919 | 0.8256 | 0.5221 | ||
| 反CP 2.0替代版本 | 当前最优方法 [51] | - | - | - | 0.9430 | 0.9640 | 0.9210 | 0.8770 |
| 数据集 | 卷积神经网络 | 0 | 0 | 1 | 0.9356 | 0.9124 | 0.9588 | 0.8721 |
| 0.1 | 0 | 1 | 0.9381 | 0.8969 | 0.9794 | 0.8793 | ||
| Transformer- | 0 | 0 | 1 | 0.9253 | 0.9227 | 0.9278 | 0.8505 | |
| 编码器 | 0.7 | 0 | 1 | 0.9356 | 0.9227 | 0.9485 | 0.8714 | |
| 长短期记忆网络 | 0 | 0 | 1 | 0.9201 | 0.9124 | 0.9278 | 0.8403 | |
| 0.7 | 0 | 1 | 0.9330 | 0.9278 | 0.9381 | 0.8660 | ||
| ACP500 + ACP164 | 当前最优方法 [36] | - | - | - | 0.9146 | 0.8902 | 0.9390 | 0.8303 |
| 卷积神经网络 | 0 | 0 | 1 | 0.7866 | 0.7317 | 0.8415 | 0.5767 | |
| 0.7 | 0.5 | 1 | 0.8354 | 0.8049 | 0.8659 | 0.6720 | ||
| Transformer- | 0 | 0 | 1 | 0.8780 | 0.9146 | 0.8415 | 0.7581 | |
| 编码器 | 0.3 | 0 | 1 | 0.9146 | 0.8659 | 0.9634 | 0.8332 | |
| 长短期记忆网络 | 0 | 0 | 1 | 0.8110 | 0.7683 | 0.8537 | 0.6242 | |
| 0.3 | 0 | 1 | 0.8293 | 0.8049 | 0.8537 | 0.6593 | ||
| ACP500 + ACP2710 | 当前最优方法 [36] | - | - | - | 0.9358 | 0.5244 | 0.9468 | 0.3271 |
| 卷积神经网络 | 0 | 0 | 1 | 0.9236 | 0.5244 | 0.9361 | 0.2944 | |
| 0.9 | 0.5 | 1 | 0.9605 | 0.4878 | 0.9753 | 0.4110 | ||
| Transformer- | 0 | 0 | 1 | 0.8760 | 0.6951 | 0.8817 | 0.2884 | |
| 编码器 | 0.3 | 0.5 | 1 | 0.9708 | 0.2683 | 0.9928 | 0.3664 | |
| 长短期记忆网络 | 0 | 0 | 1 | 0.8952 | 0.5732 | 0.9053 | 0.2627 | |
| 0.3 | 0 | 1 | 0.9185 | 0.5732 | 0.9292 | 0.3070 | ||
| 李 + 独立 | 当前最优方法 [36] | - | - | - | 0.9667 | 0.9467 | 0.9867 | 0.9341 |
| 卷积神经网络 | 0 | 1 | 2 | 0.9733 | 0.9600 | 0.9867 | 0.9470 | |
| 0.1 | 0.5 | 1 | 0.9767 | 0.9533 | 1.0000 | 0.9544 | ||
| Transformer- | 0 | 1 | 2 | 0.9667 | 0.9467 | 0.9867 | 0.9341 | |
| 编码器 | 0.5 | 0 | 1 | 0.9767 | 0.9667 | 0.9867 | 0.9535 | |
| 长短期记忆网络 | 0 | 0 | 1 | 0.9633 | 0.9533 | 0.9733 | 0.9269 | |
| 0.1 | 0.5 | 1 | 0.9733 | 0.9667 | 0.9800 | 0.9468 |
最佳和次佳表现分别用加粗和下划线突出显示。
已知具有抗癌、抗菌和抗真菌活性的Stigmurin(Uniprot ID P0DQT1),被用于研究氨基酸残基的贡献,这些氨基酸残基包含在ACP-Mixed-80测试数据集中[53, 54]。Stigmurin呈现出α-螺旋构象,并且由于在α-螺旋区域的总体正归因分数,其被预测为ACP的概率为0.93(图5B和C)。由于α-螺旋形式的肽可以穿透癌细胞,在预测ACP时模型考虑了这一特性,特别强调α-螺旋部分[6]。此外,我们检查了在接收来自嵌入层输出的变压器编码器的第一个注意力层中,哪些氨基酸标记被主要考虑(图5D)。发现模型中的每个头捕捉到不同的自注意力模式,显著关注氨基酸对,特别是苯丙氨酸-赖氨酸(FK)、异亮氨酸-丝氨酸(IS)、苯丙氨酸-苯丙氨酸(FF)、甘氨酸-甘氨酸(GG)、苯丙氨酸-丝氨酸(FS)和脯氨酸-丝氨酸(PS),以及[CLS]标记。作为另一个例子,M-zodatoxic-Lt2a也被研究,因为它具有双α-螺旋结构并表现出抗癌和抗菌活性[55 - 57]。形成α-螺旋的氨基酸残基在优化模型中有较高的归因分数,将它们识别为ACP的概率为0.96(图S19A)。与先前结果一致,每个头捕捉到不同的自注意力模式(图S19B)。
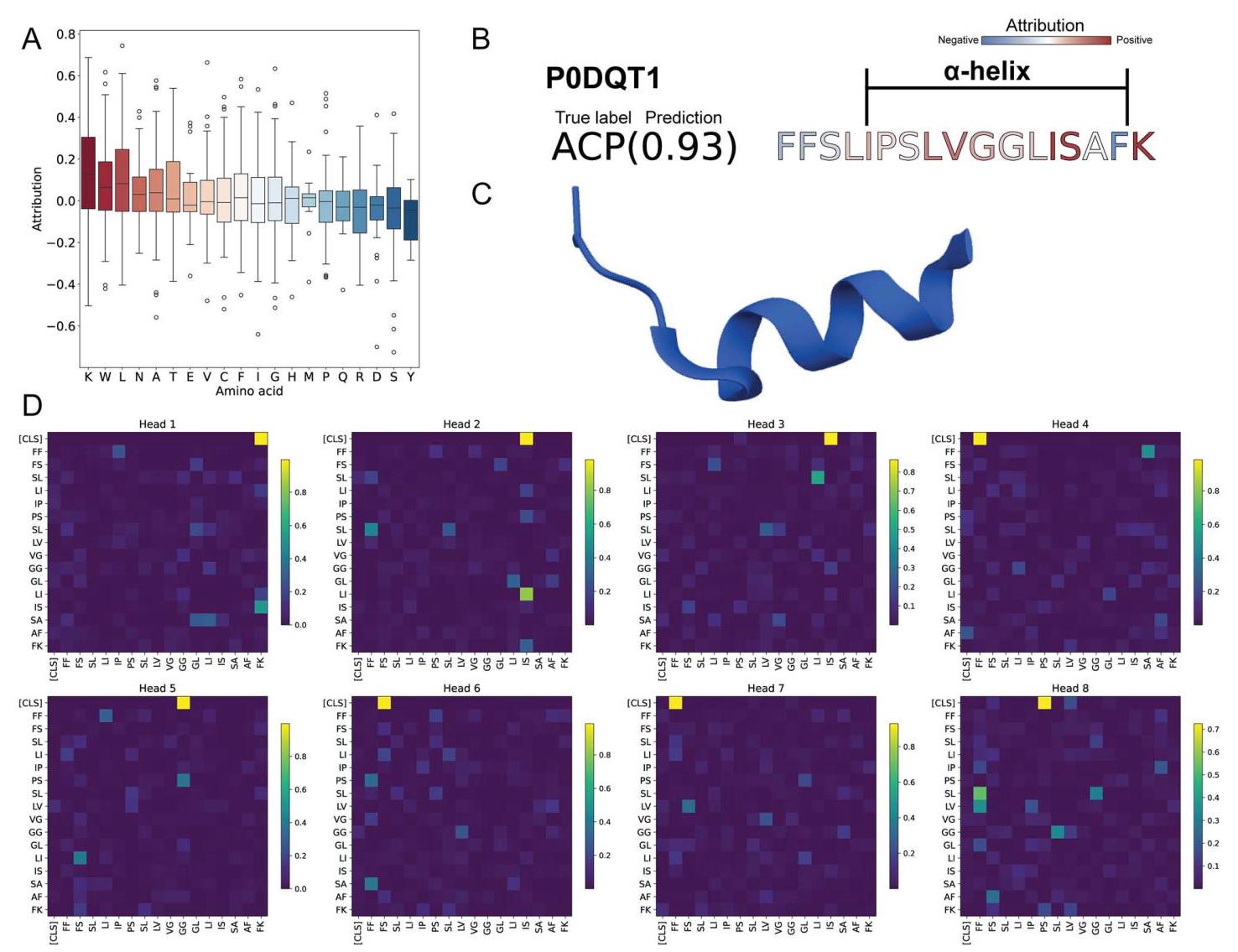
图5. 使用ACP-Mixed-80测试数据集的模型可解释性研究。(A) 一个箱线图,展示了用于分类抗癌肽(ACP)的单个氨基酸的归因分数。氨基酸根据其归因均值按降序排列。(B) 提供了Stigmurin(Uniprot ID: PODQT1)的归因示例。模型预测该肽为ACP的概率为0.93,并且α-螺旋氨基酸主要显示出高归因分数。(C) 描绘了从PDB数据库(PDB ID: 7KDQ)获得的Stigmurin的肽结构。(D) 展示了第一个变压器编码器层的各个注意力头的注意力分数。不同的头对某些氨基酸标记表现出不同的偏好,例如头2和头3关注“IS”,头5关注“GG”。
讨论
本研究提出了用于分类ACP的端到端深度学习模型,通过我们建议的对比学习方法展示了显著的性能提升。对比学习是一种自监督表示学习,通过将数据集配对来操作,以使正样本对的特征紧密对齐,同时拉开负样本对的特征距离,随后在数据中学习有意义的表示[30]。自监督对比学习通常需要数据增强;然而,由于肽的特性,数据增强过程不容易应用于肽。因此,构建了两个相同的模型,它们仅使用不同的嵌入层,每个嵌入层通过不同的分词器处理相同肽序列的不同索引。这种双编码器配置允许我们的模型整合肽序列的不同表示,通过为相同序列提供不同视角暗示了数据增强的效果。这些不同的表示对于我们的对比学习框架至关重要,在该框架中目标是学习一个能最大程度区分不同样本的特征空间。与不适合我们肽数据集的传统数据增强方法不同,这种使用双编码器的创新方法允许我们利用序列的固有变异性。通过为其提供更复杂的特征集,这种方法可能增强泛化能力和鲁棒性,类似于传统数据增强的目标。
为ACP建立了六个基准数据集,包含606 - 3210个样本,与其他深度学习应用相比相对较少(表S1)。其中,ACP500 + ACP2710在测试数据中的正样本和负样本之间存在不平衡,使得准确率指标不足以衡量模型性能。由于除了ACP-Mixed-80之外,在其他基准数据集中测试数据集中至少25%的样本与训练数据集样本显著相似,因此ACP-Mixed-80可以作为最无偏的性能指标呈现(表1)。
为衡量模型性能,主要建议使用准确率、灵敏度、特异性和马修斯相关系数(MCC)。选择这些指标是因为它们在先前的研究中经常被测量,从而为比较模型性能提供了一致的基础。此外,对于后续实验验证中被确定为抗癌肽(ACP)的预测,将误报率降至最低至关重要。通过提高所确定的抗癌肽的可靠性,可以减少与实验过程相关的成本损失。这是通过降低在实验验证期间对未表现出抗癌作用的肽投入资源的可能性来实现的,从而优化研究的效率和成本效益。从这个角度来看,由于精确率指标表明了性能,我们还在表S3中展示了召回率指标的性能。
为评估对比损失对模型训练的影响,我们根据系数α评估了模型性能。在使用抗癌肽混合80(ACP-Mixed-80)数据集的分析中,我们观察到随着系数α增加到0.7或0.9,反映出更高的对比损失率,灵敏度降低而特异性增加(图2)。在变压器编码器以及将长短期记忆网络(LSTM)应用于抗癌肽500 + 抗癌肽2710(ACP500+ ACP2710)数据集时也观察到了这种趋势。相比之下,在其他基准数据集中,高α值降低了灵敏度和特异性指标(图S7 - S11)。然而,在每种情况下,与其他α值相比,α值为0.9时观察到的马修斯相关系数(MCC)指标值更低,这表明高对比损失率对模型性能有负面影响。尽管对于抗癌肽500 + 抗癌肽2710数据集,在变压器编码器和长短期记忆网络中较高的α值会使特异性和准确率增加,但这种趋势可能因数据不平衡问题而产生偏差[58]。因此,我们使用MCC指标,因为当数据集不平衡时,它能提供更准确的度量,以便更全面地评估整体模型性能。相比之下,我们观察到在其余平衡数据集中,准确率指标与MCC指标呈现出平行趋势。
为评估对比学习对模型训练的有效性,我们将通过对比学习训练的模型性能与仅使用交叉熵(CE)损失训练模型的基线进行了比较。此外,为确保由于初始权重设置导致的模型性能一致,对对比学习和基线使用七个固定的随机种子值训练模型。这种方法允许在对比学习和基线之间进行直接的性能比较,且学习从相同的模型权重开始。在大多数情况下,无论编码器架构和基准数据集如何,对比学习与基线相比都提高了模型性能(表S2)。然而,在将对比学习与基线进行比较时,超过65%的模型在对抗癌肽500 + 抗癌肽2710进行分类时表现出灵敏度降低。对比学习增强了将负样本准确识别为真阴性的能力,同时可能降低了将正样本分类为真阳性的效果。
为将我们的模型与当前最优(SOTA)模型进行基准比较,基于最高的马修斯相关系数(MCC)指标值,在各自的编码器架构和训练方法中选择性能最佳的模型。在所有基准数据集中,除了抗癌肽2.0主数据集外,相对于SOTA模型都取得了卓越的性能(表2)。此外,在每个数据集中评估整体性能的MCC指标中表现最佳的情况,都始终与对比学习相关。在抗癌肽混合80(ACP-Mixed-80)和LEE + 独立(LEE + Independent)数据集中,实现最高MCC性能的优化模型在所有指标上均优于SOTA模型。然而,对于抗癌肽500+抗癌肽164(ACP500+ ACP164)、抗癌肽500+抗癌肽2710和抗癌肽2.0替代数据集,与SOTA模型相比,优化模型的灵敏度降低而特异性增加。此外,在所有数据集上基线与对比学习的比较中,在所有编码器架构中特异性始终保持或增加,而在某些情况下灵敏度会下降。这一结果可能归因于通过对比学习的表示学习过程,它有助于更深入地理解现实世界中更广泛、更多样化的负数据集所固有的特征。
为剖析该模型的预测过程,我们在嵌入层采用了可解释人工智能技术,从而能够了解每个输入氨基酸的贡献。在使用ACP-Mixed-80数据集时,赖氨酸、色氨酸和亮氨酸对ACP分类的贡献最为显著(图5A)。赖氨酸和亮氨酸因其参与细胞膜破坏而为人所知,而色氨酸与癌细胞代谢有关[59 - 62]。然而,由于这些氨基酸的存在,这一结果并不意味着优化后的模型会直接提高ACP的概率。该模型会考虑序列上下文,这意味着预测结果会受到周围氨基酸残基及其相互作用的影响。因此,由于氨基酸的顺序排列,氨基酸的箱线图结果显示出显著差异。对于特定的肽情况,我们将注意力集中在\(\alpha\) -螺旋二级结构上(图5B和图S19A)。在ACP-Mixed-80数据集中,当通过alphafold预测结构时,超过80.5%的ACP具有\(\alpha\) -螺旋结构,13.9%具有\(\beta\) -片层结构(补充数据)。这种显著出现的\(\alpha\) -螺旋结构与已有的研究一致,该研究强调了其在ACP中的重要性,支持了\(\alpha\) -螺旋结构在我们模型的归因分数中的重要性。
虽然该模型在癌症抗癌功能预测方面表现出一定能力,但在确定特定癌症类型时却遇到困难。这一局限性源于针对特定类型癌症的已知抗癌肽数量不足,无法满足有效学习的需求,而有效学习对于准确预测至关重要。预测出的ACP在抗癌活性方面可能具有特异性,有可能针对某些癌症亚型。因此,我们的模型可作为潜在ACP的筛选工具,这是治疗开发早期的关键一步,有助于快速识别有前景的肽类药物先导物。具体而言,鉴于动物毒液中存在大量有益肽,我们的模型能够加速对众多源自毒液的潜在ACP的筛选[64]。为了将这些肽推进到治疗应用阶段,筛选出的候选物必须经过进一步的实验验证和优化。我们的模型能够显著加快抗癌疗法中有前景的肽类药物先导物的发现,提高药物开发过程初始阶段的效率。
在当前的肽研究领域,人们对发现具有多种功能特性的肽,如抗癌、抗菌、抗真菌和抗炎活性,兴趣日益浓厚。随后,许多基于计算机模拟的方法正在被开发以推动这一努力。然而,由于生物领域的固有性质,阳性数据集只能从生物实验中获得,这对可用于模型训练的数据集大小施加了重大限制。本研究中开发的对比学习方法克服了这些限制,并且如我们在ACP预测中的结果所证实的,已证明有潜力产生比现有方法更精确的预测模型。为了增强对比学习在ACP中的应用,监督对比学习可能是一个可行的选择。虽然我们当前的方法在对比学习中将相同样本视为相似样本,而不考虑它们的类别,但监督对比学习可以将相似样本定义为属于同一类别的样本。此外,通过适当的数据增强,而不是我们的方法,它将能够在对比学习中使用更多样化的序列。这些方法可以促进提取特定于每个类别的通用特征,有可能提高预测性能。
要点
-
本文重点在于开发一种基于计算机模拟的工具来预测ACP,强调了有效癌症治疗的必要性。
-
本研究特别将对比学习应用于肽预测模型,在ACP预测方面取得了优于现有方法 的性能,这已通过多个评估指标得到验证。
-
本文利用可解释人工智能技术来理解特定氨基酸对ACP预测的影响,为模型的决策过程提供了见解。
补充数据
补充数据可在网上获取:http://bib.oxford journals.org/。
作者贡献
B.L.设计并实现了预测模型。D.S.监督了这项工作并为该项目组织了资源。
资金支持
本研究得到了韩国国家研究基金会(NRF)通过教育部资助的基础科学研究计划(RS - 2023 - 00272756)、韩国政府资助的韩国国家研究基金会(NRF)、科学和信息通信技术部(NRF - 2022R1A2C1010532)以及国家癌症中心资助(NCC - 2210510)的支持。
代码可用性
用PyTorch实现的代码可在GitHub上获取:(http:// github.com/bzlee-bio/con_ACP)。
参考文献
-
迪奥·S、夏尔马·J、库马尔·S。《2020年全球癌症负担报告:外科肿瘤学家面临的挑战与机遇》。《外科肿瘤学年鉴》2022年;29卷:6497 - 500页。
-
宋H、费雷J、西格尔·RL等。《2020年全球癌症统计数据:全球185个国家36种癌症的发病率和死亡率的全球癌症负担估计》。《CA:临床医师癌症杂志》2021年;71卷:209 - 49页。
-
陈·KB、朗·JJ、康普顿·K等。《2010 - 2019年归因于风险因素的全球癌症负担:全球疾病负担研究2019的系统分析》。《柳叶刀》2022年;400卷:563 - 91页。
-
格尔斯滕·O、威尔莫斯·JR。《1951年以来日本的癌症转变》。《人口研究》2002年;7卷:271 - 306页。
-
王L、王N、张W等。《治疗性肽:当前应用和未来方向》。《信号转导与靶向治疗》2022年;7卷:48页。
-
江宗W、楚提蓬塔内特·S、洪恩·S。《抗癌肽:物理化学性质、功能方面及临床应用趋势》。《国际肿瘤学杂志》2020年;57卷:678 - 96页。
-
博尔豪茨·C、昆茨·C、格罗纳·B。《基于肽的抗癌治疗药物开发的当前策略》。《肽科学杂志》2005年;11卷:713 - 26页。
-
钦纳杜赖·RK、汗·N、梅格万希·GK等。《抗癌肽的当前研究现状:作用机制、生产及临床应用》。《生物医学与药物治疗》2023年;164卷:114996页。
-
费茨·J、坎德尔·S、马马尼·U - F、程·K。《治疗性肽开发的最新进展》。《药理学趋势》202'3年;44卷:425 - 41页。
-
维什涅波尔斯基·B、格里戈拉娃·M、马纳加德泽·G等。《基于机器学习算法的微生物菌株特异性抗菌肽预测的比较分析》。《简短生物信息学》2022年;23卷:bbac233页。
-
严J、巴德拉·P、李A等。《深度抗菌肽预测模型30:利用深度学习改进短抗菌肽预测》。《分子治疗:核酸》2020年;20卷:882 - 94页。
-
李H、李S、李I、南H。《抗菌肽BERT模型:基于BERT模型的抗菌肽功能预测》。《蛋白质科学》2023年;32卷:e4529页。
-
李B、申·MK、柳·JS等。《通过深度多任务学习从星豹蛛毒腺中鉴定新型抗菌肽》。《微生物前沿》2022年;13卷:971503页。
-
吕Z、崔F、邹Q等。《基于深度表征学习特征的抗癌肽预测》。《简短生物信息学》2021年;22卷:bbab008页。
-
阿格拉瓦尔·P、巴加特·D、马哈瓦尔·M等。《抗癌肽预测模型2.0:预测抗癌肽的更新模型》。《简短生物信息学》2021年;22卷:bbaa153页。
-
李B、申·MK、金T等。《通过知识转移方法识别离子通道调节肽的预测模型》。《IEEE生物医学与健康信息学杂志》2022年;26卷:6150 - 60页。
-
蒂亚吉 A、卡普尔 P、库马尔 R 等。用于设计和发现新型抗癌肽的计算机模拟模型。《科学报告》2013 年;3:2984。
-
陈 W、丁 H、冯 P 等。iACP:一种基于序列的抗癌肽识别工具。《肿瘤靶向》2016 年;7:16895 - 909。
-
哈吉沙里菲 Z、皮尔亚伊 M、贝吉 MM 等。用周氏伪氨基酸组成预测抗癌肽并通过艾姆斯试验研究其致突变性。《理论生物学杂志》2014 年;341:34 - 40。
-
李 F - M、王 X - Q。使用改进的混合组成识别抗癌肽。《科学报告》2016 年;6:33910。
-
阿克巴尔 S、哈亚特 M、伊克巴尔 M、扬 MA。iACP - GAEnsC:利用混合特征空间基于进化遗传算法的抗癌肽集成分类。《人工智能医学》2017 年;79:62 - 70。
-
卡比尔 M、阿里夫 M、艾哈迈德 S 等。通过整合序列和进化谱信息鉴别抗癌肽的智能计算方法。《化学计量学与智能实验室系统》2018 年;182:158 - 65。
-
沙杜昂拉特 N、南塔塞纳马特 C、普拉查亚西蒂库尔 V、舒姆 - 布阿通 W。ACPred:一种用于预测和分析抗癌肽的计算工具。《分子》2019 年;24:1973。
-
马纳瓦兰 B、巴西思 S、申 TH 等。MLACP:基于机器学习的抗癌肽预测。《肿瘤靶向》2017 年;8:77121 - 36。
-
魏 L、周 C、苏 R、邹 Q。PEPred - suite:使用自适应特征表示学习改进并稳健预测治疗性肽。《生物信息学》2019 年;35:4272 - 80。
-
吴 C、高 R、张 Y、德马里尼斯 Y。PTPD:通过深度学习和 word2vec 预测治疗性肽。《BMC 生物信息学》2019 年;20:1 - 8。
-
易 H - C、游 Z - H、周 X 等。ACP - DL:一种使用高效特征表示预测抗癌肽的深度学习长短期记忆模型。《分子治疗:核酸》2019 年;17:1 - 9。
-
张 YP、邹 Q。PPTPP:一种使用物理化学性质编码和自适应特征表示学习的新型治疗性肽预测方法。《生物信息学》2020 年;36:3982 - 7。
-
贾斯瓦尔 A、巴布 AR、扎德 MZ 等。对比自监督学习综述。《牙科技术》2020 年;9:2。
-
勒 - 哈克 PH、希利 G、斯米顿 AF。对比表示学习:一个框架与综述。《IEEE 接入》2020 年;8:193907 - 34。
-
陈 T、科恩布利思 S、诺鲁齐 M 等。视觉表示对比学习的一个简单框架。见:《第 37 届国际机器学习会议论文集》,2020 年,1597 - 607。
-
考希克 D、霍维 E、利普顿 ZC。利用反事实增强数据学习有差异的差异。2020 年国际学习表征会议。
-
迟 J、尚德 W、于 Y 等。用于公平文本分类的条件监督对比学习。arXiv 预印本 arXiv:2205.11485 2022。
-
裴 S、金 J-W、赵 W-Y 等。基于音频频谱图变压器的补丁混合对比学习在呼吸音分类中的应用。arXiv 预印本 arXiv:2305.14032 2023。
-
海因辛格 M、利特曼 M、西利托 I 等。蛋白质嵌入的对比学习揭示了午夜区。《核酸研究:基因组学与生物信息学》2022 年;4:lqac043。
-
何 W、王 Y、崔 L 等。基于多感官缩放注意力架构学习嵌入特征以提高抗癌肽的预测性能。《生物信息学》2021 年;37: 4684 - 93。
-
魏 L、周 C、陈 H 等。ACPred - FL:一种基于序列的预测器,使用有效特征表示来改进抗癌肽的预测。《生物信息学》2018 年;34:4007 - 16。
-
卡马乔 C、库洛里斯 G、阿瓦吉安 V 等。BLAST +:架构与应用。《生物医学中心·生物信息学》2009 年;10:1 - 9。
-
闵 S、李 B、尹 S。生物信息学中的深度学习。《简短生物信息学》2017 年;18:851 - 69。
-
瓦斯瓦尼 A、沙泽尔 N、帕尔马尔 N 等。你只需要注意力。见:《第 31 届神经信息处理系统国际会议论文集》2017 年,5998 - 6008。
-
庞 Y、姚 L、徐 J 等。整合变压器和不平衡多标签学习以识别抗菌肽及其功能活性。《生物信息学》2022 年;38:5368 - 74。
-
肖 X、邵 Y - T、程 X、斯塔马托维奇 B。iAMP - CA2L:一种基于细胞自动机图像的新型 CNN - BiLSTM - SVM 分类器,用于识别抗菌肽及其功能类型。《简短生物信息学》2021 年;22:bbab209。
-
邢 W、张 J、李 C 等。iAMP - Attenpred:一种基于 BERT 特征提取方法和 CNN - BiLSTM - 注意力组合模型的新型抗菌肽预测器。《简短生物信息学》2024 年;25:bbad443。
-
帕斯克 A、格罗斯 S、马萨 F 等。PyTorch:一种命令式风格、高性能的深度学习库。《神经信息处理系统进展》2019 年;32。
-
科赫利基扬 N、米格兰尼 V、马丁 M 等。Captum:一个用于 PyTorch 的统一通用模型可解释性库。arXiv 预印本 arXiv:2009.07896 2020。
-
桑达拉扬 M、塔利 A、严 Q。深度网络的公理归因。见:《第 34 届机器学习国际会议论文集》2017 年;70:3319 - 28。
-
邓 H、丁 M、王 Y 等。ACP - MLC:一种用于识别抗癌肽及其功能类型多标签分类的两级预测引擎。《计算机生物学与医学》2023 年;158:106844。
-
艾哈迈德 S、穆罕默德 R、汗 ZH 等。ACP - MHCNN:一种用于预测抗癌肽的精确多头深度卷积神经网络。《科学报告》2021 年;11:23676。
-
袁Q,陈K,于Y等。基于深度学习和机器学习集成模型并使用有序位置编码的抗癌肽预测。《简短生物信息学》2023年;24:bbac630。
-
查罗恩坤P,蒋宗W,李VS等。使用新型灵活评分卡方法改进肽类抗癌活性的预测和表征。《科学报告》2021年;11:3017。
-
阿齐兹AZB,哈桑MAM,艾哈迈德S等。iACP - MultiCNN:基于多通道卷积神经网络的抗癌肽鉴定。《分析生物化学》2022年;650:114707。
-
饶B,周C,张G等。ACPred - fuse:融合多视图信息改进抗癌肽预测。《简短生物信息学》2020年;21:1846 - 55。
-
帕伦特AM,达尼埃莱 - 席尔瓦A,富尔塔多AA等。蝎毒肽stigmurin的类似物:结构评估、毒性及增强的抗菌活性。《毒素》2018年;10:161。
-
德梅洛ET,埃斯特雷拉AB,桑托斯ECG等。来自蝎子Tityus stigmurus毒腺的具有抗菌活性的新型肽stigmurin的结构表征。《肽》2015年;68:3 - 10。
-
瓦德瓦尼P,塞卡兰S,斯特兰德伯格E等。拉塔辛的膜相互作用:来自蜘蛛毒液的抗菌肽。《国际分子科学杂志》2021年;22:10156。
-
波利亚尼斯基AA,瓦西列夫斯基AA,沃利尼斯基PE等。N端两亲性螺旋作为抗菌肽溶血活性的触发因素:以拉塔辛为例的研究。《欧洲生物化学学会联合会快报》2009年;583:2425 - 8。
-
科兹洛夫SA,瓦西列夫斯基AA,费奥法诺夫AV等。拉塔辛,来自蜘蛛Lachesana tarabaevi( Zodariidae科)毒液的抗菌和细胞溶解肽,体现了生物分子多样性。《生物化学杂志》2006年;281:20983 - 92。
-
斋藤T,雷姆斯迈尔M。在不平衡数据集上评估二元分类器时,精确召回率图比ROC图更具信息性。《公共科学图书馆·综合》2015年;10:10。
-
戴Y,蔡X,施W等。富含赖氨酸或精氨酸的促凋亡阳离子宿主防御肽通过破坏肿瘤细胞膜逆转耐药性。《氨基酸》2017年;49:1601 - 10。
-
布尼亚D,蒙达尔P,达斯G等。空间位置调节色氨酸的作用:发现一种主要沟特异性核定位、细胞穿透四肽。《美国化学会志》2018年;140:1697 - 714。
-
哈里斯F,丹尼森SR,辛格J,菲尼克斯DA。关于防御肽对癌细胞的选择性和功效。《医学研究评论》2013年;33:190 - 234。
-
王C,董S,张L等。富含赖氨酸/亮氨酸的肽L - K6对人乳腺癌MCF - 7细胞的细胞表面结合、摄取及抗癌活性。《科学报告》2017年;7:8293。
-
朱珀J,埃文斯R,普里茨尔A等。使用AlphaFold进行高精度蛋白质结构预测。《自然》2021年;596:583 - 9。
-
穆滕塔勒M,金GF,亚当斯DJ,阿莱伍德PF。肽类药物发现的趋势。《自然综述:药物发现》2021年;20:309 - 25。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号