目前,在NCBI中下载SRA数据主要有三种方式:
- 利用Aspera工具下载。
- 利用SRA Toolkit下载。
- 利用wget命令直接下载
第三种最为方便。其中的关键是得到下载数据的链接,即ftp的地址
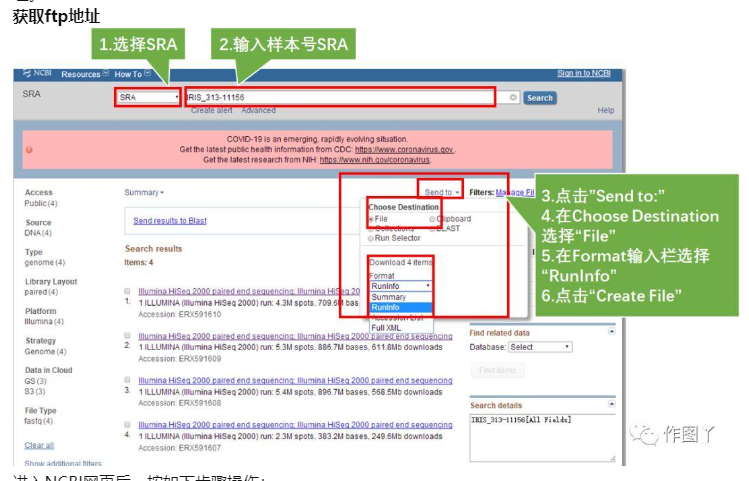
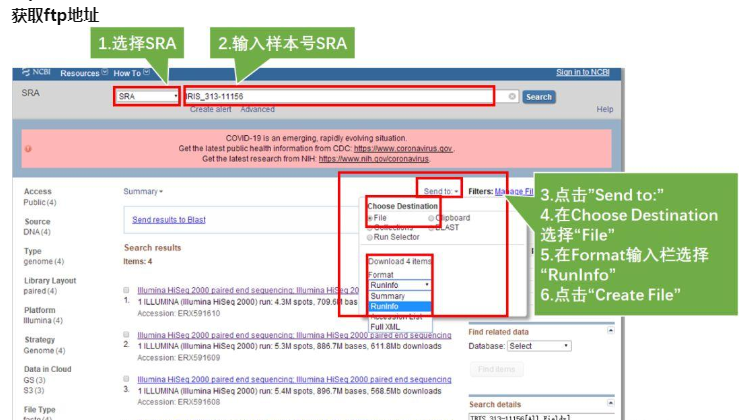
进入NCBI网页后,按如下步骤操作:
- Step1.设置NCBI的分类为:SRA
- Step2.输入感兴趣的样本号:IRIS_313-11156,点击Search,弹出四条item,说明该样本分四次run上级,我们需要全部下载
- Step3.点击右上角的Send to
- Step4.在Choose Destination中选择File
- Step5.在Format输入栏选择RunInfo
- Step6.点击Create File,此刻会生成一个名为SraRunInfo.csv的文件,图中标黄的一列即为不同次run数据的ftp地址。
wget -c 50 下载地址
若想批量下载则把下载地址放到一个list里面,然后运行下面的代码:
wget -c 50 -i list.txt
下面这个网址里面也有一些内容可供参考:
https://www.jianshu.com/p/0694fcb77157
https://www.cnblogs.com/zdwu/p/8473986.html
下载好的数据是sra压缩格式,这个格式是ncbi特有的一种格式,需要将此格式的文件转换成fastq文件的格式
sra是NCBI 推出的存储高通量数据的格式,而平常我们工作用得多是fastq格式。如果需要把sra 转成fastq,从
http://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?cmd=show&f=software&m=software&s=software
下载相应的软件。
或者下载最新的source code,在服务器上用make 编译。
然后使用如下命令行:
sra_sdk-2.0.0rc1/linux/rel/gcc/x86_64/bin/fastq-dump -A SRR034580 -D SRR034580.sra
这样就可以很简单的把sra格式转成fastq格式了。
转换 .sra 文件成 .fastq/fasta 文件
#single-end 单端测序
.../fastq-dump DRR000003.sra # 结果生成DRR000003.fastq
.../fastq-dump --fasta DRR000003.sra # 结果生成DRR000003.fastq
#pair-end 双端测序
.../fastq-dump --split-3 DRR002018.sra # 结果生成 DRR002018_1.fastq,DRR002018_2.fastq
REF:
http://blog.sina.com.cn/s/blog_4055a5940100o1mg.html
http://hi.baidu.com/wuyu466/item/152006eb4363eac3baf37d29
http://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software
http://blog.sina.com.cn/s/blog_70b2b6020100liee.html



 浙公网安备 33010602011771号
浙公网安备 33010602011771号