Reconstruction of neocortex: Organelles, compartments, cells, circuits, and activity
郑重声明:原文参见标题,如有侵权,请联系作者,将会撤销发布!

Cell, (2022)
SUMMARY
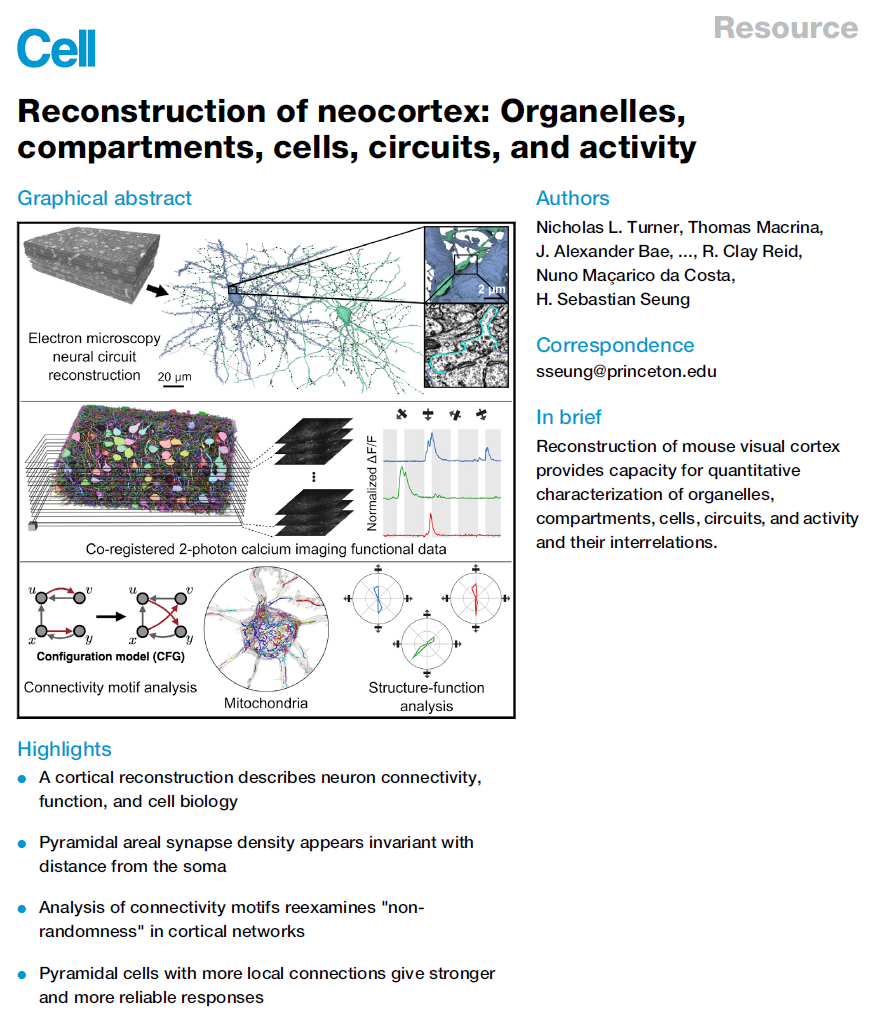
我们从~250 × 140 × 90 μm3的电子显微镜图像中组装了L2/3小鼠初级视觉皮层的半自动重建,包括锥体和非锥体神经元、星形胶质细胞、小胶质细胞、少突胶质细胞和前体、周细胞、脉管系统、细胞核、线粒体和突触。包括锥体细胞子集的视觉反应。这些数据以及用于程序化和三维交互式访问的工具是公开的。简短的小插曲说明了在皮层回路和神经元细胞生物学中将结构与功能相关的潜在应用的广度。线粒体和突触组织的特征是从胞体的路径长度的函数。使用随机图的配置模型准确预测金字塔连接基序频率。从附近细胞接收更多连接的锥体细胞表现出更强和更可靠的视觉反应。示例代码显示了数据访问和分析。
INTRODUCTION
借助图像采集(Kornfeld and Denk, 2018)和分析(Lee et al., 2019)的自动化,从无脊椎动物和脊椎动物物种的电子显微镜(EM)图像重建脑组织的规模和复杂性都在增加。在一些研究中,体内钙成像先于EM重建(Briggman et al., 2011; Vishwanathan et al., 2017; Bae et al., 2018; Wanner and Friedrich,2020)。这种组合的结构-功能方法已应用于小鼠初级视觉(V1)皮层(Bock et al., 2011; Lee et al., 2016),但以前仅限于数十个细胞。在此,我们展示了小鼠V1的重建,包括来自100多个锥体细胞(PyC)的神经元和非神经元细胞、线粒体、细胞核、突触和钙成像。这些数据是公开的(microns-explorer.org),以及用于编程和交互式访问的工具。我们还提供了四个小插曲,探索线粒体、突触、电路基序以及连通性与功能的关系,选择这些小插曲来展示这种多方面重建可以揭示的定量信息的多样性。
RESULTS
Reconstruction of mouse visual cortex
Spatial organization of mitochondria
Spatial organization of synapses onto excitatory and inhibitory cells
Predicting motif frequencies from degree sequence
Relation of in-connection density to visual function
DISCUSSION
我们对250 × 140 × 90 μm3小鼠视觉皮层的EM重建的多模态性质和规模使其可用于定量表征细胞器、隔室、细胞、电路和活动,以及它们的相互关系。这些小插曲和随附的Jupyter笔记本表明,我们的资源将广泛用于调查有关皮质结构和功能的广泛问题。
我们研究了PyCs不同隔室中线粒体的空间组织,并评估了远端树突中线粒体体积、突触数和神经突直径之间的相关性。总体而言,我们发现线粒体的大小分布与之前关于其运输和动力学限制的假设一致(Lewis et al., 2018)。线粒体体积与突触数量呈正相关,这与树突状线粒体通过ATP产生和钙缓冲支持突触功能的观点一致,尽管这种关系很难与树突状直径分开。
我们的突触分析发现,平均区域突触密度与与基部树突远端区域的体细胞的距离大致保持不变,并且在距离胞体的任何距离处波动小于线性突触密度。观察到的不变性表明0.33突触/μm2是基底树突远端区域的稳态设定点。树突生物物理学的一个结果是突触电导可以随树突表面积线性缩放。假定离子通道密度是恒定的,本征电导也与表面积成线性关系。突触和内在电导的常见缩放对树突的计算建模有影响。
皮质发育的计算模型假设突触表现得好像它们在争夺固定资源(Miller, 1996),并且有实验证据支持这一想法(Kasthuri and Lichtman, 2003)。一个开创性的模型(von der Malsburg, 1973)提出突触竞争树突区域,这可能导致每单位面积的突触数量近似不变。根据基于轴突和树突的随机几何形状的理论预测,树突段接收的突触数量平均应与包含刺的圆柱体的表面积成正比(Stepanyants et al., 2002)。这种比例类似于我们观察到的面积突触密度不变性。
我们的连通性主题检查使用随机图的CFG模型分析我们的PyC图,正如在秀丽隐杆线虫中所做的那样(Milo et al., 2002; Reigl et al., 2004; Varshney et al., 2011),这由于重建的规模是可能的。与秀丽隐杆线虫连接组不同,我们的PyC接线图低估了连接性,因为细胞被EM体积的边界截断。对于轴突长度至少为100 μm的 、111个PyC的子集,相隔50 mm或更短的胞体的连接概率为8.1%(±1.1%, 95%CI, n = 2530)。这低于脑切片生理学的类似值,在年轻小鼠中的范围从14%到22%(Ko et al., 2011, 2013; Cossell et al., 2015)。我们对连通性的低估并没有阻止我们复制众所周知的发现(Markram et al., 1997; Song et al., 2005; Le Bé and Markram, 2006; Wang et al., 2006),相对于ER随机图模型,双向连接被过度表示。低估减少了双向连接的数量,但也减少了ER预测。同样,截断并没有阻止我们发现双向连接相对于CFG的过度表示。
由于我们距离完整的哺乳动物连接组还有很长的路要走(Abbott et al., 2020),因此对于哺乳动物接线图来说,截断仍将是一个现实。脑切片实验也受到轴截断的影响,因为记录的神经元通常距离切片表面只有几十微米(Stepanyants et al., 2009; Levy and Reyes, 2012)。更大的皮质体积的重建将告诉我们我们的研究结果受到截断的影响有多大。就目前而言,我们应该小心那些声称连接基序在皮层中高度"非随机"的说法。
如果分析仅限于强连接,则图案的过度表现可能会变得更加引人注目(Song et al., 2005)。连接概率被认为在成年后会降低(Jiang et al., 2016; Campagnola et al., 2021),这与连通性细化的概念一致。目前的研究是在年轻的成年(P36)小鼠中进行的,之前的研究是在幼年大鼠中进行的(Song et al., 2005; Le Bé and Markram, 2006; Perin et al., 2011)。在成年动物中寻找过度代表的图案会很有趣。
最后,在检查视觉功能的连接密度时,我们发现连接密度与间歇性呈负相关,与视觉反应幅度呈正相关。我们的发现与之前对小鼠V1中"合唱团"与"独奏者"PyC的研究有关(Okun et al., 2015)。合唱团比独奏者更有可能接收到来自相邻单元的连接,并且往往对视觉刺激做出更强烈的反应。这两个结果都与我们的分析一致,表明连接密度与细胞对其首选刺激的反应强度相关。我们的分析包括具有广泛入度的单元(图S7D和S7E),而之前的工作仅限于几个单元的小电路,并使用随机图模型外推到更大的入度。先前的工作也没有考虑视觉反应的间歇性。
除了这些小插曲之外,三篇文章还提供了深入分析,展示了如何使用该资源来研究PyC突触大小分布和多突触连接(Dorkenwald et al., 2019)、枝形吊灯细胞(Schneider-Mizell et al., 2021)和OPCs (Buchanan et al., 2021)。另外两篇文章描述了我们对立方毫米尺度体积的重建(MICrONS Consortium et al., 2021; Macrina et al., 2021),这也是公开的。
Limitations of the study
每个分析都假设我们样本中的PyCs是一个统一的群体。未来在细胞分型方面的努力可能会揭示这些结果的新维度或提供研究抑制细胞的新方法。同样,我们经常假设同一神经突隔室的分支是统一的,其中细胞器和突触的位置可能在各个分支内相互作用以影响细胞功能。对这些重建进行更细粒度的分析可能是未来工作的另一个有价值的途径。我们希望这项工作能够激发我们的读者发现新的应用程序并充分发挥资源的发现潜力。
STAR METHODS
KEY RESOURCES TABLE
RESOURCE AVAILABILITY
EXPERIMENTAL MODEL AND SUBJECT DETAILS
Mouse
METHOD DETAILS
Cranial window surgery
Widefield imaging
Two-photon imaging
Functional imaging scans
High-resolution structural stack
Behavioral monitoring
Visual stimulus
EM & two-photon co-registration
Trace extraction and spike inference
Tissue preparation and staining
Sectioning and collection
Transmission electron microscopy
Alignment in two blocks
Alignment, block one
Stitching
Rough alignment
Alignment, block two
Stitching
Rough alignment
Coarse alignment
Fine alignment
Alignment, whole dataset
Coarse alignment
Fine alignment
Image volume estimation
Voxel resolution
Image defect handling
Affinity prediction
Initial oversegmentation
Mean affinity agglomeration
Synaptic cleft detection
Synaptic partner assignment
Mitochondria detection and assignment
Cell-type assignment
Tissue quality assessment
PyC proofreading
PyC-PyC synapse proofreading
Nucleus detection
Skeletonization
Neuronal compartment labeling
QUANTIFICATION AND STATISTICAL ANALYSIS
Neurite length calculation
Synapse detection performance estimation
PyC dendritic segment analysis
Mitochondrion analysis
PyC and interneuron cell input analysis
Random network models
Random network model comparison
Whole-cell degree and connection density
Direction and orientation selectivity
Preferred orientation
Mean response
Intermittency
Location-restricted permutation test
Statistical reporting
Figure rendering information
ADDITIONAL RESOURCES
Supplemental figures




 浙公网安备 33010602011771号
浙公网安备 33010602011771号