安装并使用SnpEff注释
SnpEff is a variant annotation and effect prediction tool. It annotates and predicts the effects of genetic variants (such as amino acid changes).
一、安装
# Go to home dir
cd
# Download latest version
wget https://snpeff.blob.core.windows.net/versions/snpEff_latest_core.zip
# Unzip file
unzip snpEff_latest_core.zip二、测试
#进入到snpEff目录下测试能否正常使用,日后在此目录下使用相关程序
cd /home/liuxin/snpEff
#查询数据库,并列出musculus相关的行或链接,正常显示则安装成功
java -jar snpEff.jar databases | grep -i musculus三、配置
#配置文件
vim snpEff.config任意行插入如下新物种信息如下
# Lychee genome
Lychee.genome : feizixiao创建对应的data文件夹存储基因组文件
#此处的Lychee文件名与前处添加的相对应
mkdir -p ~/snpEff/data/Lychee
#进入目录下
cd ~/snpEff/data/Lychee在Lychee文件夹下面,获取建库所需文件的两个文件
#参考基因组序列
sequences.fa.gz
#gff3注释文件
genes.gff.gz四、建库
使用build命令建库
java -jar snpEff.jar build -gff3 -v Lychee五、注释
以vcf文件作为输入调用自建库Lychee开始注释获取注释后的snpeff.vcf
#input.vcf.gz作为输入VCF文件
java -jar snpEff.jar ann Lychee input.vcf.gz > snpeff.vcf若只关注CDS中的注释信息,不考虑上游、下游、UTR、基因间区等信息,可使用如下参数简化输出集
java -jar snpEff.jar ann Lychee \
-no-utr -no-downstream -no-upstream -no-intergenic \
input.vcf.gz > snpeff.vcfPS:若数据库中有参考也可以直接下载,无须建库。
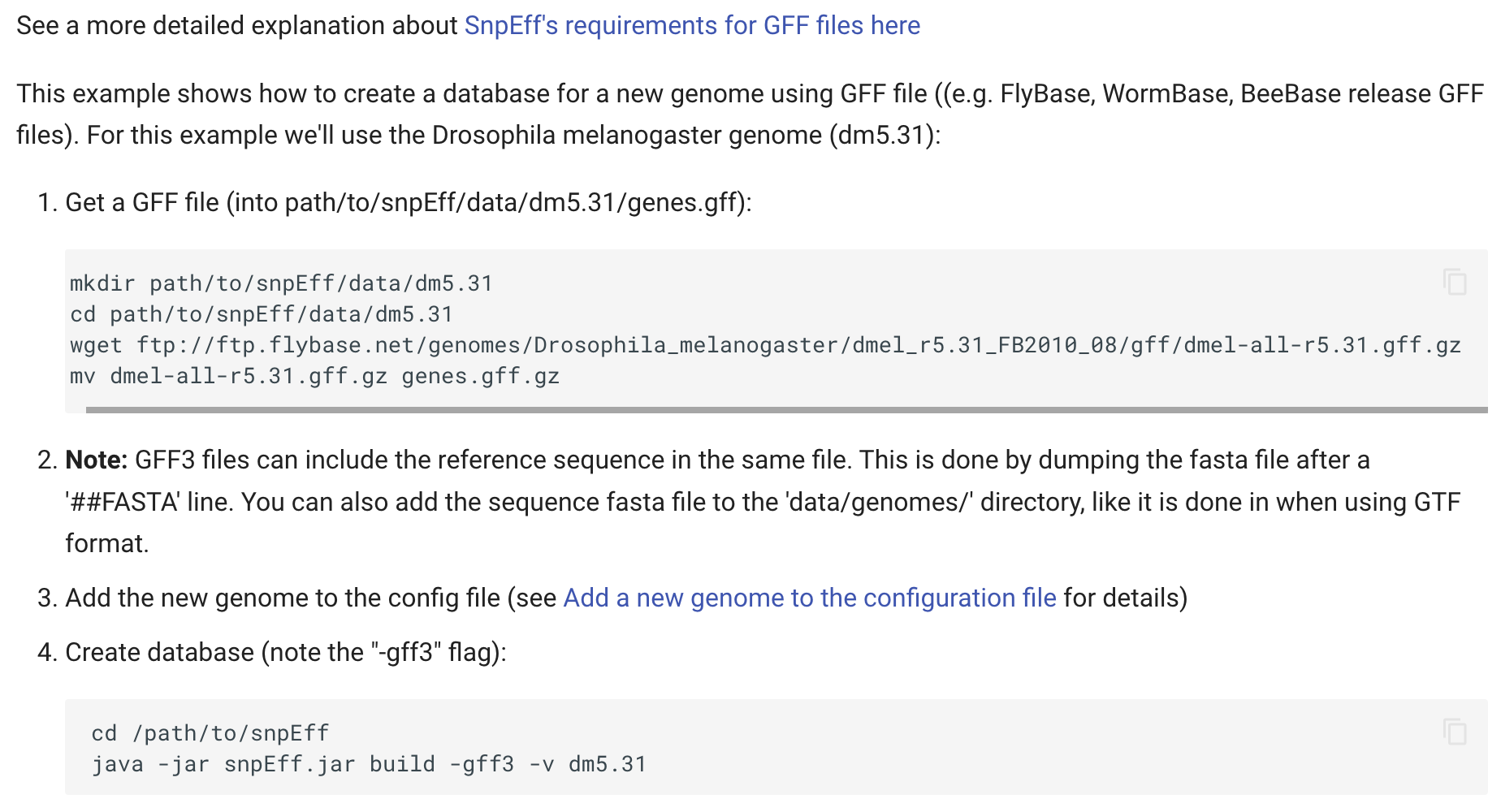
PS:建库过程参考图(配置config文件建议准备好内容然后粘贴插入)
参考自:



 浙公网安备 33010602011771号
浙公网安备 33010602011771号