Linux 中统计每条染色体上gap的数目
001、将fasta碱基转换为一行
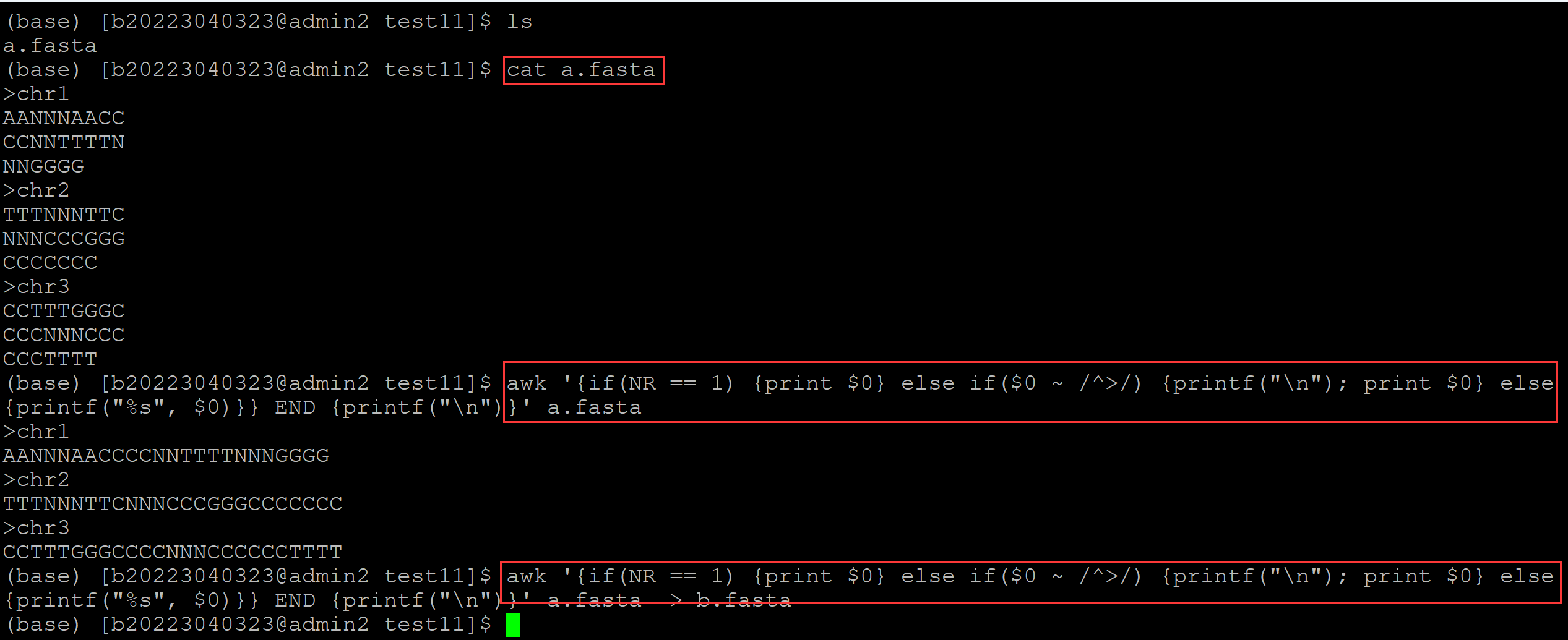
(base) [b20223040323@admin2 test11]$ ls a.fasta (base) [b20223040323@admin2 test11]$ cat a.fasta >chr1 AANNNAACC CCNNTTTTN NNGGGG >chr2 TTTNNNTTC NNNCCCGGG CCCCCCC >chr3 CCTTTGGGC CCCNNNCCC CCCTTTT (base) [b20223040323@admin2 test11]$ awk '{if(NR == 1) {print $0} else if($0 ~ /^>/) {printf("\n"); print $0} else {printf("%s", $0)}} END {printf("\n")}' a.fasta >chr1 AANNNAACCCCNNTTTTNNNGGGG >chr2 TTTNNNTTCNNNCCCGGGCCCCCCC >chr3 CCTTTGGGCCCCNNNCCCCCCTTTT (base) [b20223040323@admin2 test11]$ awk '{if(NR == 1) {print $0} else if($0 ~ /^>/) {printf("\n"); print $0} else {printf("%s", $0)}} END {printf("\n")}' a.fasta > b.fasta
。
002、统计每条染色体上的gap数据
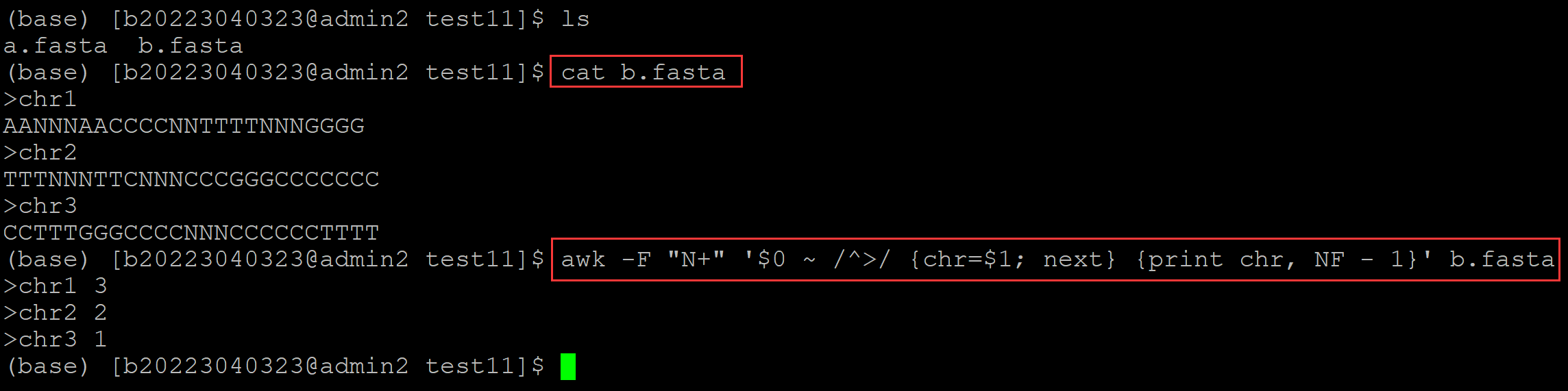
(base) [b20223040323@admin2 test11]$ ls a.fasta b.fasta (base) [b20223040323@admin2 test11]$ cat b.fasta ## 上一步转换出来的单行碱基fasta文件 >chr1 AANNNAACCCCNNTTTTNNNGGGG >chr2 TTTNNNTTCNNNCCCGGGCCCCCCC >chr3 CCTTTGGGCCCCNNNCCCCCCTTTT (base) [b20223040323@admin2 test11]$ awk -F "N+" '$0 ~ /^>/ {chr=$1; next} {print chr, NF - 1}' b.fasta ## 统计每条染色体上的gap数目 >chr1 3 >chr2 2 >chr3 1
。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号