使用PAUP软件基于最大简约发构建系统进化树
使用PAUP软件基于最大简约发构建系统进化树
001、将vcf格式转换为fasta格式:
(base) [b20223040323@admin2 test5]$ ls outcome.vcf (base) [b20223040323@admin2 test5]$ vcf2phylip.py --input outcome.vcf --fasta

002、将fasta格式转换nex格式
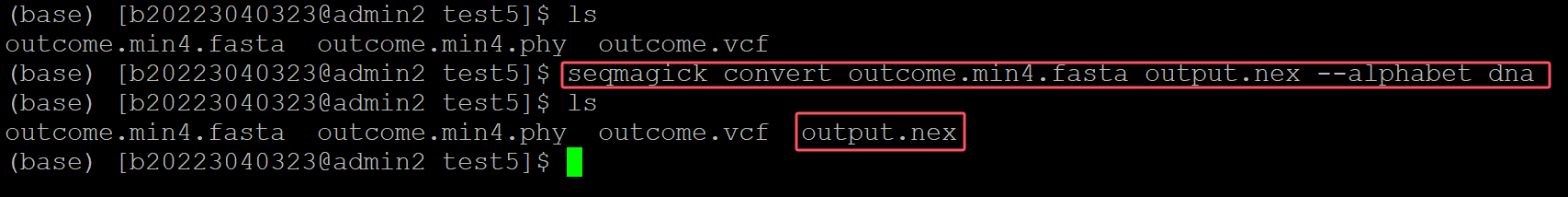
(base) [b20223040323@admin2 test5]$ ls outcome.min4.fasta outcome.min4.phy outcome.vcf (base) [b20223040323@admin2 test5]$ seqmagick convert outcome.min4.fasta output.nex --alphabet dna (base) [b20223040323@admin2 test5]$ ls outcome.min4.fasta outcome.min4.phy outcome.vcf output.nex
。
003、修改nex文件的格式,增加如何内容到文件的末尾
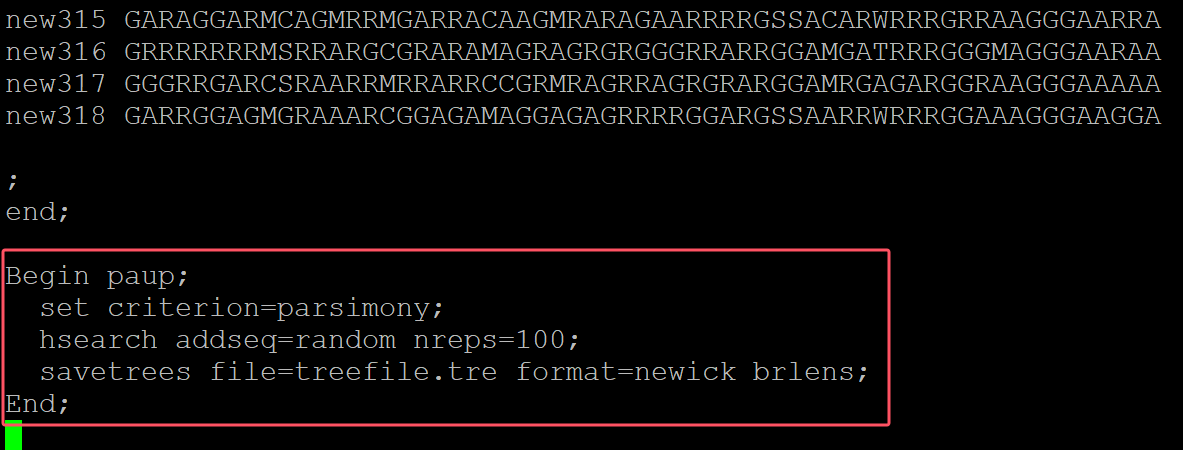
Begin paup; set criterion=parsimony; hsearch addseq=random nreps=100; savetrees file=treefile.tre format=newick brlens; End;
。
或者:
Begin paup; set criterion=parsimony; bootstrap nreps=500; hsearch addseq=random nreps=100; savetrees file=treefile.tre format=newick brlens; End;
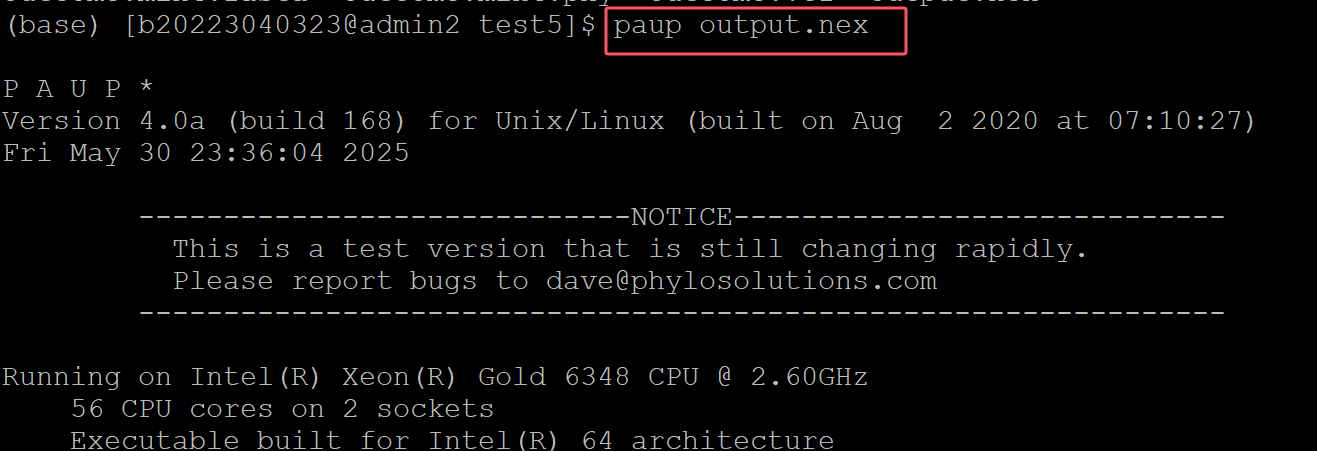
004、paup output.nex 执行运算
。
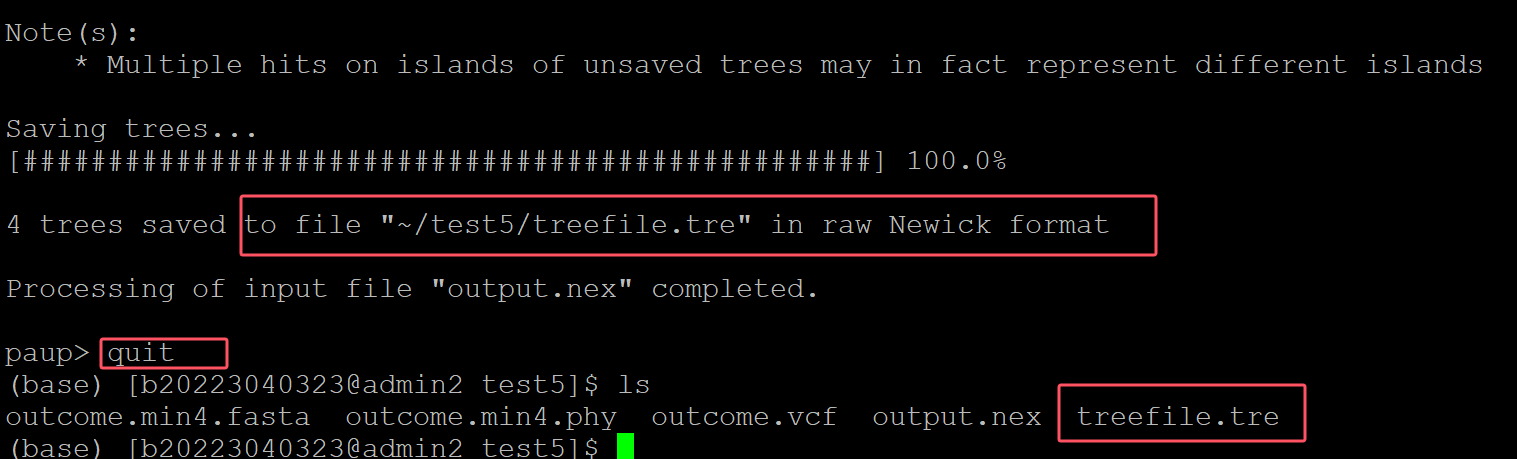
005、 运算结束
。




 浙公网安备 33010602011771号
浙公网安备 33010602011771号