shell中实现DNA序列一致性计算
001、
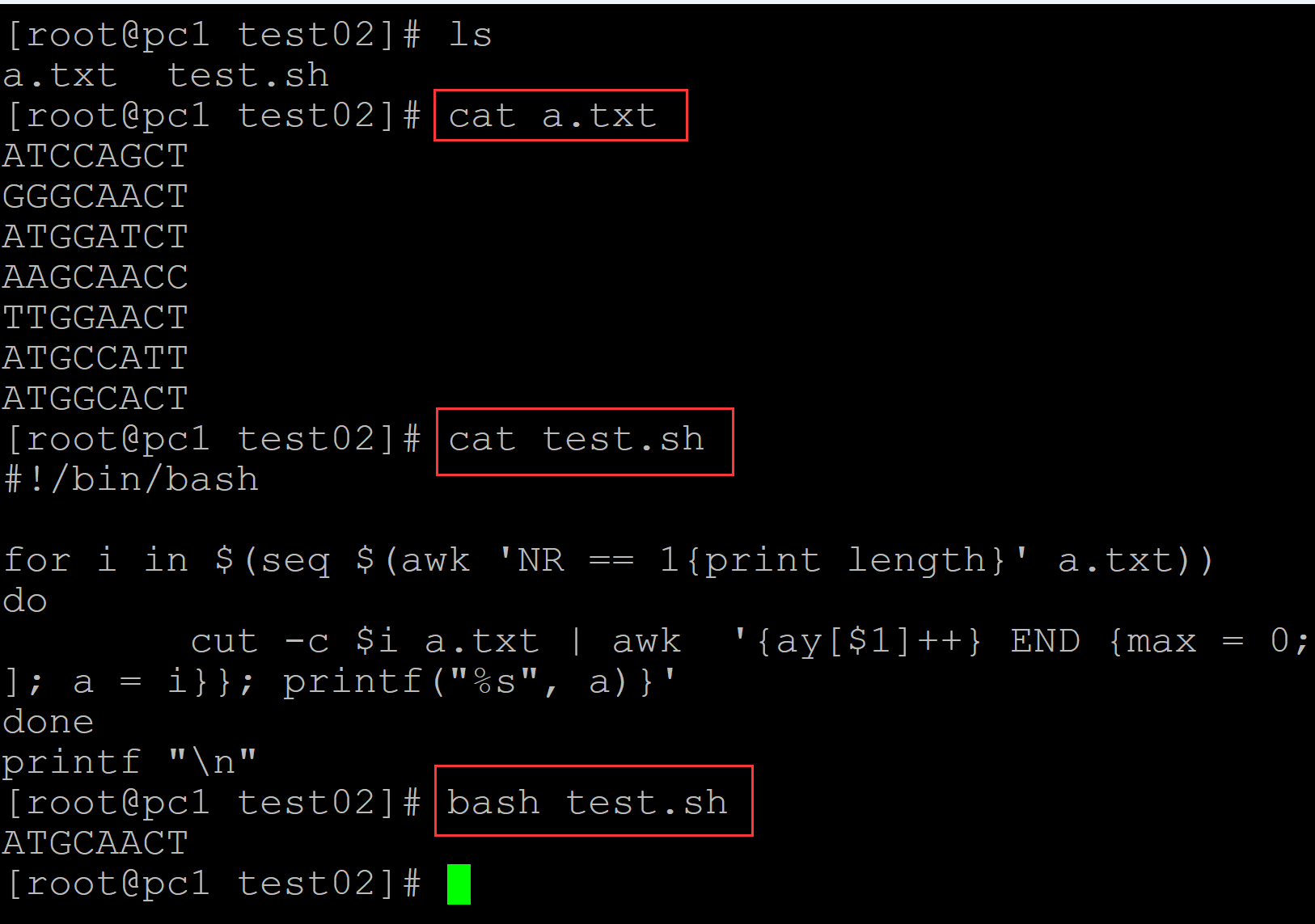
[root@pc1 test02]# ls a.txt test.sh [root@pc1 test02]# cat a.txt ## 测试数据 ATCCAGCT GGGCAACT ATGGATCT AAGCAACC TTGGAACT ATGCCATT ATGGCACT [root@pc1 test02]# cat test.sh ## 测试脚本 #!/bin/bash for i in $(seq $(awk 'NR == 1{print length}' a.txt)) do cut -c $i a.txt | awk '{ay[$1]++} END {max = 0; for (i in ay) {if(ay[i] > max){max = ay[i]; a = i}}; printf("%s", a)}' done printf "\n" [root@pc1 test02]# bash test.sh ## 计算结果 ATGCAACT
002、
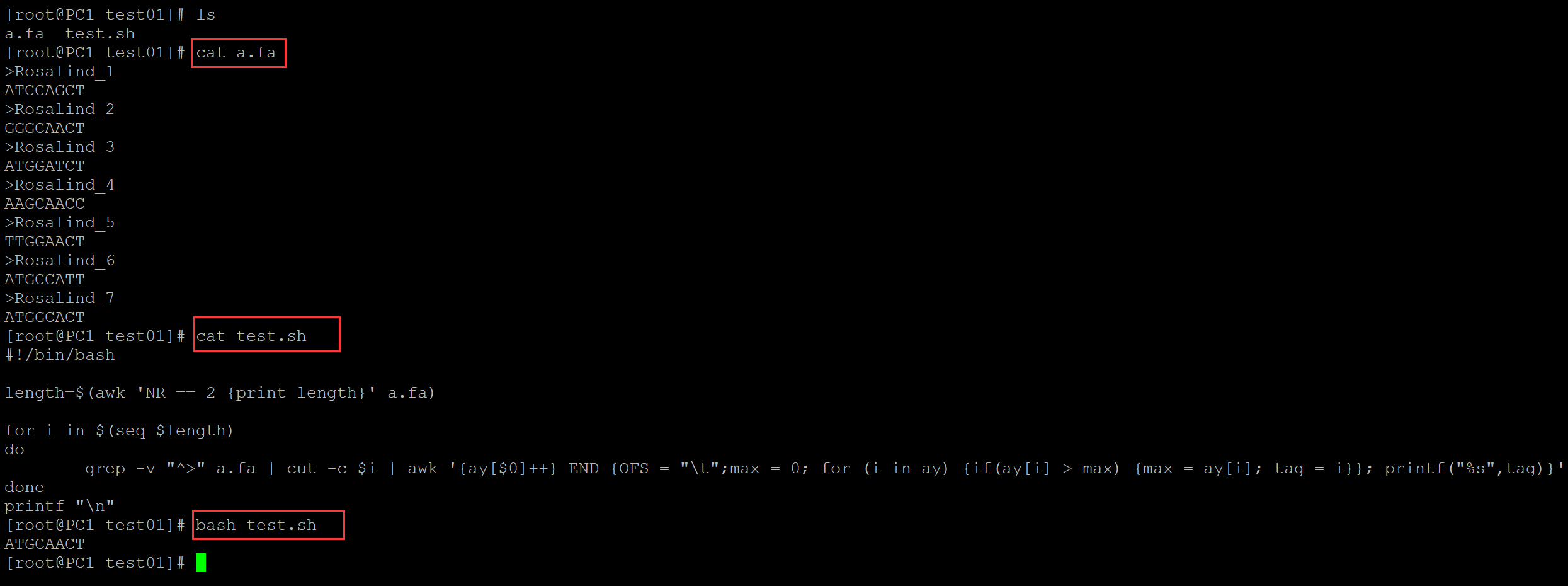
[root@PC1 test01]# ls a.fa test.sh [root@PC1 test01]# cat a.fa ## 测试数据 >Rosalind_1 ATCCAGCT >Rosalind_2 GGGCAACT >Rosalind_3 ATGGATCT >Rosalind_4 AAGCAACC >Rosalind_5 TTGGAACT >Rosalind_6 ATGCCATT >Rosalind_7 ATGGCACT [root@PC1 test01]# cat test.sh ## 测试程序 #!/bin/bash length=$(awk 'NR == 2 {print length}' a.fa) for i in $(seq $length) do grep -v "^>" a.fa | cut -c $i | awk '{ay[$0]++} END {OFS = "\t";max = 0; for (i in ay) {if(ay[i] > max) {max = ay[i]; tag = i}}; printf("%s",tag)}' done printf "\n" [root@PC1 test01]# bash test.sh ## 执行程序 ATGCAACT
。




 浙公网安备 33010602011771号
浙公网安备 33010602011771号