python中计算点突变的数目
001、直接比较计算
[root@PC1 test01]# ls a.fa b.fa test.py [root@PC1 test01]# cat a.fa ## 测试dna序列 GAGCCTACTAACGGGAT [root@PC1 test01]# cat b.fa ## 测试dna序列 CATCGTAATGACGGCCT [root@PC1 test01]# cat test.py ## 计算程序 #!/usr/bin/env python # -*- coding: utf-8 -*- in_file1 = open("a.fa", "r") in_file2 = open("b.fa", "r") file1 = in_file1.read().strip() file2 = in_file2.read().strip() in_file1.close() in_file2.close() count = 0 for i in range(len(file1)): if file1[i] != file2[i]: count += 1 print(count) [root@PC1 test01]# python3 test.py ## 计算结果 7

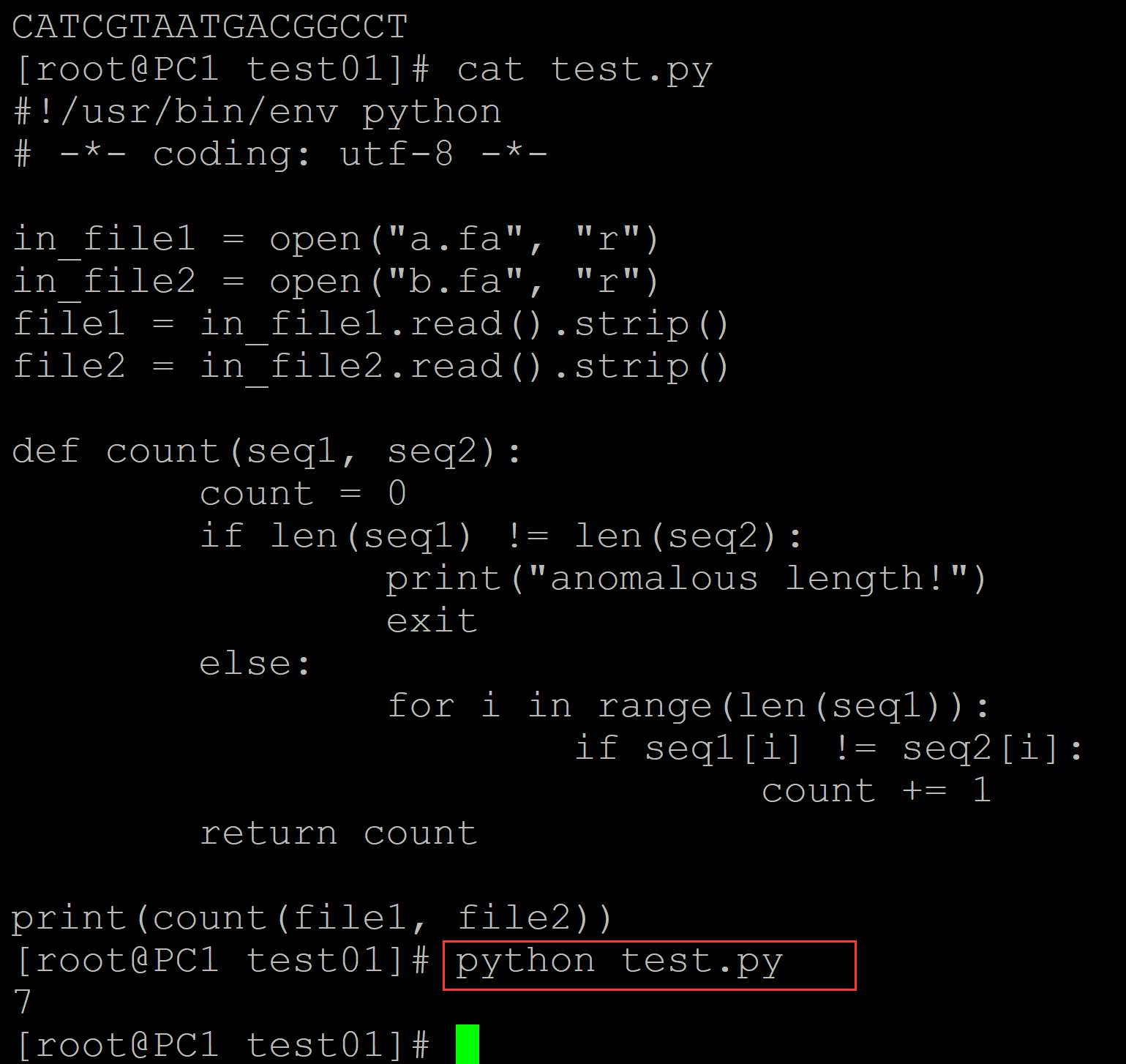
002、利用函数结构计算
[root@PC1 test01]# ls a.fa b.fa test.py [root@PC1 test01]# cat a.fa ## 测试dna序列 GAGCCTACTAACGGGAT [root@PC1 test01]# cat b.fa ## 测试dna序列 CATCGTAATGACGGCCT [root@PC1 test01]# cat test.py ## 计算程序 #!/usr/bin/env python # -*- coding: utf-8 -*- in_file1 = open("a.fa", "r") in_file2 = open("b.fa", "r") file1 = in_file1.read().strip() file2 = in_file2.read().strip() def count(seq1, seq2): count = 0 if len(seq1) != len(seq2): print("anomalous length!") exit else: for i in range(len(seq1)): if seq1[i] != seq2[i]: count += 1 return count print(count(file1, file2)) [root@PC1 test01]# python test.py ## 计算结果 7
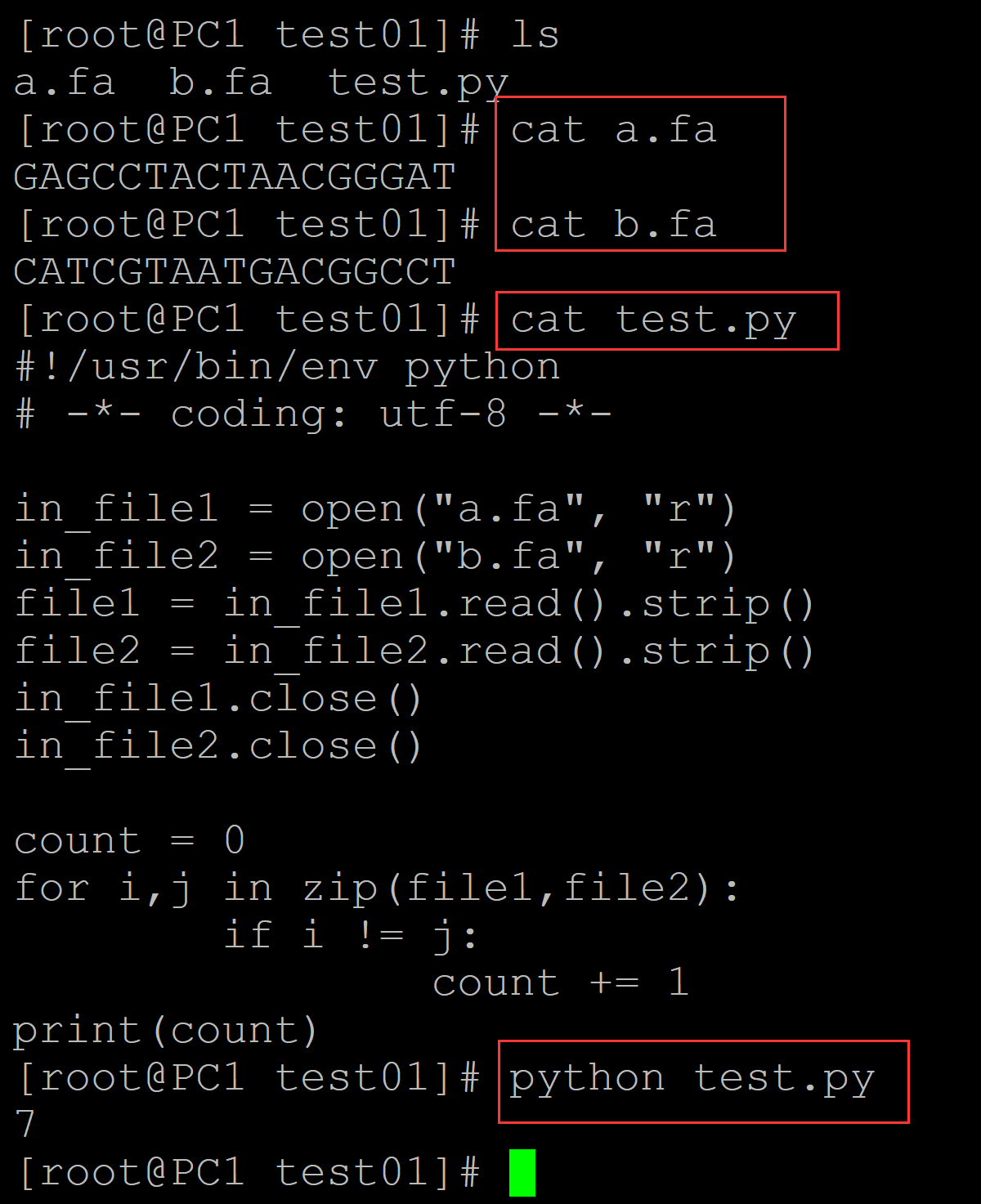
003、借助zip实现
[root@PC1 test01]# ls a.fa b.fa test.py [root@PC1 test01]# cat a.fa GAGCCTACTAACGGGAT [root@PC1 test01]# cat b.fa CATCGTAATGACGGCCT [root@PC1 test01]# cat test.py ## 计数程序 #!/usr/bin/env python # -*- coding: utf-8 -*- in_file1 = open("a.fa", "r") in_file2 = open("b.fa", "r") file1 = in_file1.read().strip() file2 = in_file2.read().strip() in_file1.close() in_file2.close() count = 0 for i,j in zip(file1,file2): if i != j: count += 1 print(count) [root@PC1 test01]# python test.py ## 点突变结果 7
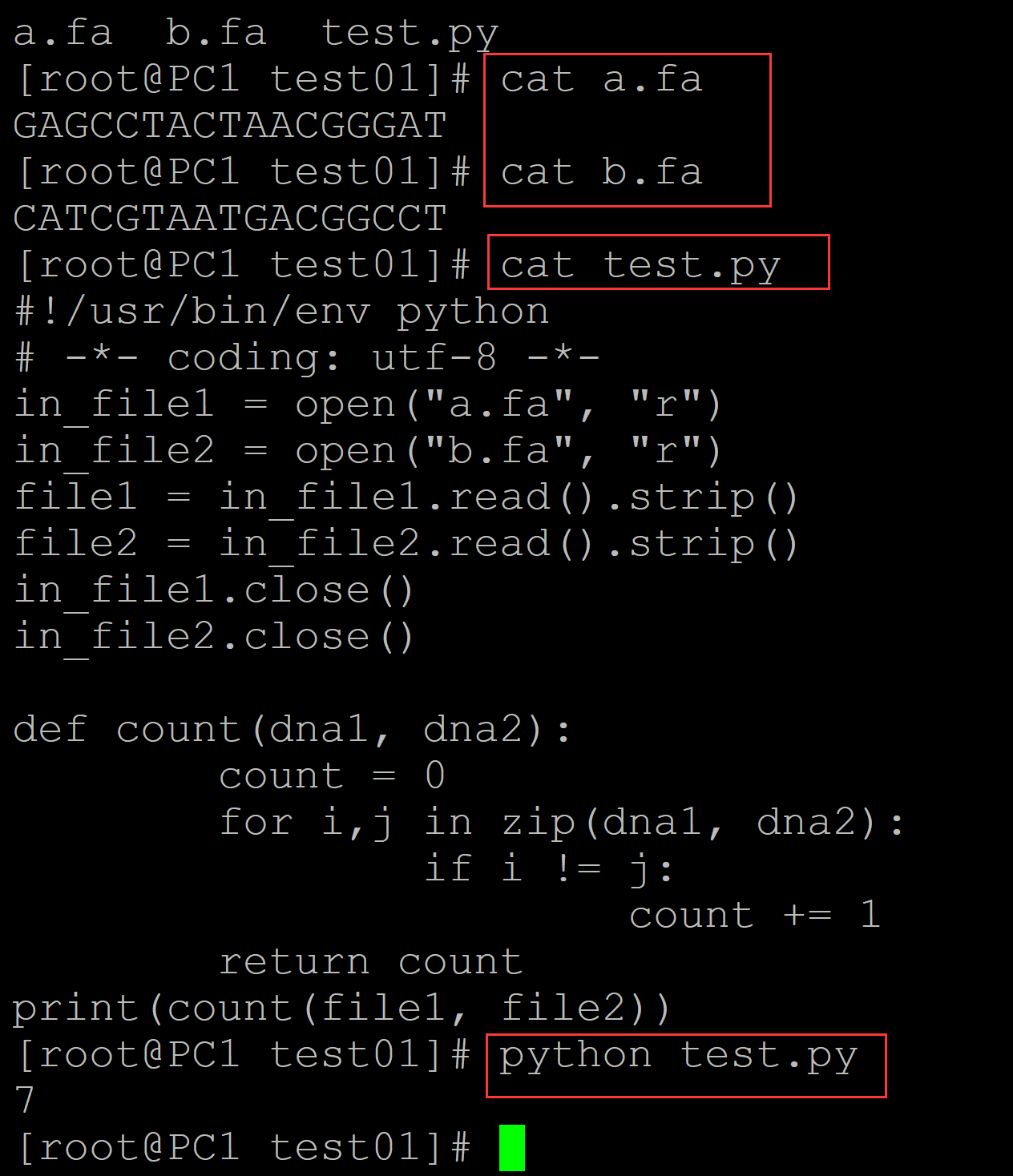
004、函数实现
[root@PC1 test01]# ls a.fa b.fa test.py [root@PC1 test01]# cat a.fa GAGCCTACTAACGGGAT [root@PC1 test01]# cat b.fa CATCGTAATGACGGCCT [root@PC1 test01]# cat test.py ## 计数程序 #!/usr/bin/env python # -*- coding: utf-8 -*- in_file1 = open("a.fa", "r") in_file2 = open("b.fa", "r") file1 = in_file1.read().strip() file2 = in_file2.read().strip() in_file1.close() in_file2.close() def count(dna1, dna2): count = 0 for i,j in zip(dna1, dna2): if i != j: count += 1 return count print(count(file1, file2)) [root@PC1 test01]# python test.py ## 计数结果 7
。




 浙公网安备 33010602011771号
浙公网安备 33010602011771号