R语言中利用prcomp函数 对基因型数据进行PCA分析
001、plink软件生成raw文件
root@PC1:/home/test# ls outcome.map outcome.ped ## maf过滤,并将基因型数据转换为0、1、2的形式 root@PC1:/home/test# plink --file outcome --sheep --maf 0.01 --recode A 1> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls outcome.map outcome.ped plink.raw root@PC1:/home/test# sed 1d plink.raw | cut -d " " -f 7- > use.raw ## 修改数据格式 root@PC1:/home/test# ls outcome.map outcome.ped plink.raw use.raw
002、R读入,并使用prcomp函数计算, 然后绘图
dat <- read.table("use.raw") ## 读入数据 pca <- prcomp(dat,center = TRUE,scale. = TRUE) ## prcomp函数进行pca分析 plot(pca$x[,1], pca$x[,2], pch = 19) ## 绘图
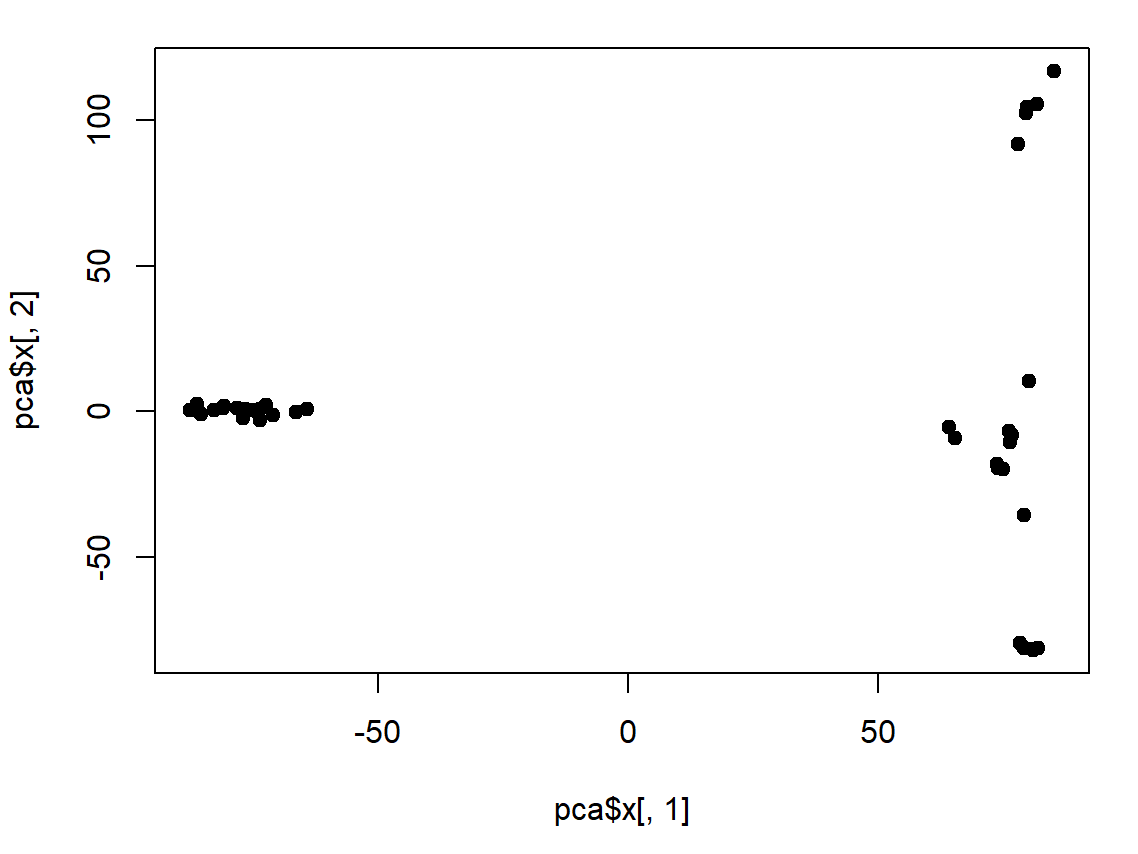
002、plink软件pca验证
root@PC1:/home/test# ls outcome.map outcome.ped ## plink软件计算pca root@PC1:/home/test# plink --file outcome --sheep --pca 3 header tabs --out pca 1> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls ## 结果文件 outcome.map outcome.ped pca.eigenval pca.eigenvec
R绘图:
par(mfrow = c(1,2)) ##分割画布 dat <- read.table("use.raw") pca <- prcomp(dat,center = TRUE,scale. = TRUE) plot(pca$x[,1], pca$x[,2], pch = 19, main = "R") ## R中生成的结果 dat2 <- read.table("pca.eigenvec", header = T) plot(dat2$PC1, dat2$PC2, pch = 19, main = "plink") ## plink中生成的结果
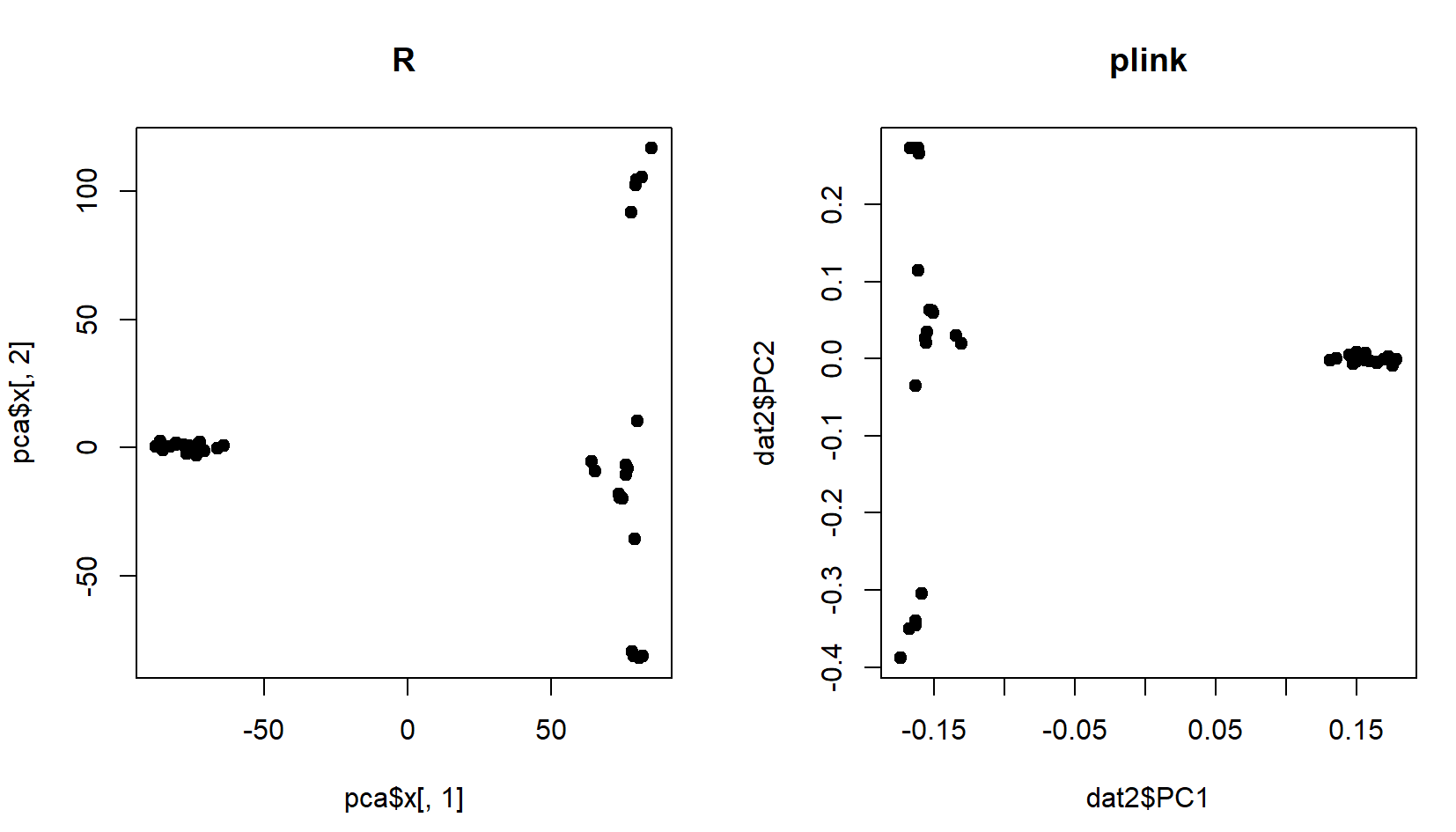
为什么结果时反着呢?




 浙公网安备 33010602011771号
浙公网安备 33010602011771号