plink软件maf过滤24号染色体异常
001、
root@PC1:/home/test# ls outcome.map outcome.ped root@PC1:/home/test# cat outcome.map ## 测试数据,均为24号染色体数据 24 snp1 0 260996 24 snp2 0 363750 24 snp3 0 299533 root@PC1:/home/test# cat outcome.ped DOR 1 0 0 0 -9 A A A G A A DOR 2 0 0 0 -9 A A G G A A DOR 3 0 0 0 -9 A A A A A A DOR 4 0 0 0 -9 A A G G A A DOR 5 0 0 0 -9 A A G G A A DOR 6 0 0 0 -9 A A A G A A
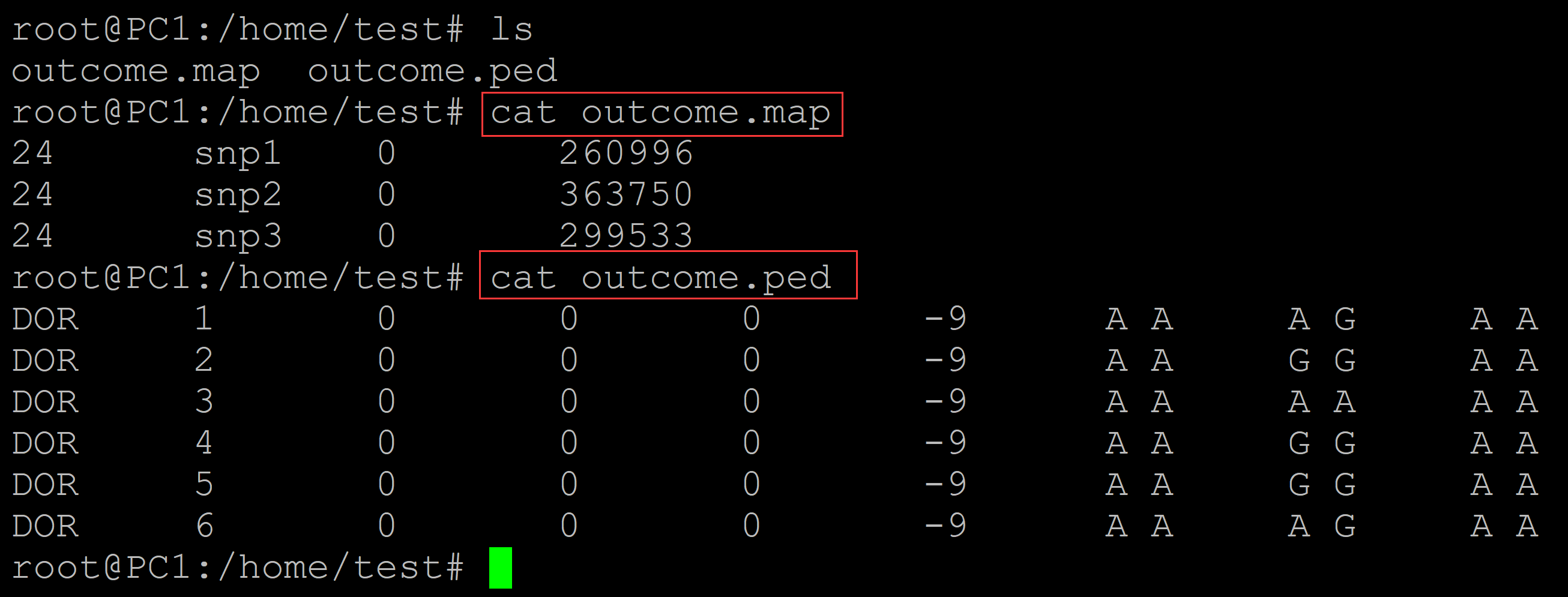
002、利用maf参数过滤
root@PC1:/home/test# plink --file outcome --maf 0.01 --recode tab --out test &>/dev/null; rm *.log *.nosex root@PC1:/home/test# ls ## 过滤结果, test*文件 outcome.map outcome.ped test.map test.ped root@PC1:/home/test# cat test.map ## maf命令并没有起作用,而且snp顺序发生了变化 24 snp1 0 260996 24 snp3 0 299533 24 snp2 0 363750 root@PC1:/home/test# cat test.ped ## 仍然有没有多态性的位点 DOR 1 0 0 0 -9 A A A A A G DOR 2 0 0 0 -9 A A A A G G DOR 3 0 0 0 -9 A A A A A A DOR 4 0 0 0 -9 A A A A G G DOR 5 0 0 0 -9 A A A A G G DOR 6 0 0 0 -9 A A A A A G
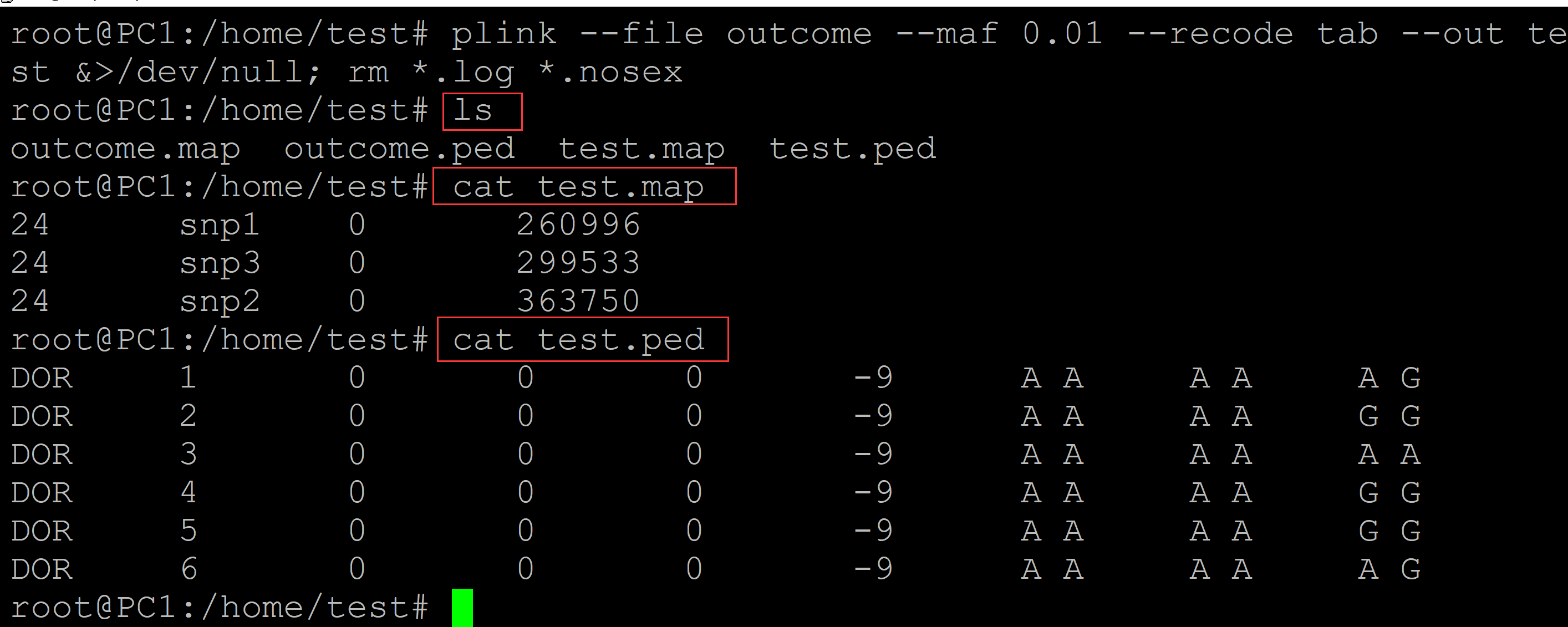
出现如下警告:

003、设定染色体的情况
root@PC1:/home/test# ls outcome.map outcome.ped root@PC1:/home/test# plink --file outcome --chr-set 40 --maf 0.01 --recode tab --out test &>/dev/null; rm *.log *.nosex root@PC1:/home/test# ls ## 过滤结果, test*文件 outcome.map outcome.ped test.map test.ped root@PC1:/home/test# cat test.map ## 当指定染色体时, maf参数起作用 24 snp2 0 363750 root@PC1:/home/test# cat test.ped DOR 1 0 0 0 -9 A G DOR 2 0 0 0 -9 G G DOR 3 0 0 0 -9 A A DOR 4 0 0 0 -9 G G DOR 5 0 0 0 -9 G G DOR 6 0 0 0 -9 A G

001、可能原因时plink将24号染色体作为了Y染色体
002、在过滤包含24号染色体的数据时, 应注意避免设置染色体。
补充:利用绵羊芯片数据测试
root@PC1:/home/test# ls sheep.map sheep.ped root@PC1:/home/test# cut -f 1 sheep.map |uniq -c ## 查看当前数据的染色体, 包含24号 998 20 768 21 984 22 1005 23 662 24 892 25 805 26 ## 在不加染色体信息情况下过滤 root@PC1:/home/test# plink --file sheep --maf 0.01 --recode --out test &> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls ## 过滤结果, test文件 sheep.map sheep.ped test.map test.ped ## 指定染色体(--sheep), 然后计算24号基因频率 root@PC1:/home/test# plink --file test --sheep --freq --chr 24 --out verify &> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls sheep.map sheep.ped test.map test.ped verify.frq root@PC1:/home/test# head verify.frq ## 查看基因频率结果文件, 发现有基因频率为0的, 说明第一步的maf参数并没有对24号染色体起作用 CHR SNP A1 A2 MAF NCHROBS 24 s75016.1 0 C 0 880 24 s72739.1 G A 0.217 880 24 s29575.1 A G 0.4852 880 24 s23759.1 0 C 0 880 24 OAR24_862318.1 G A 0.2716 880 24 s01400.1 A T 0.1284 880 24 OAR24_885120.1 T A 0.2568 880 24 s65907.1 C A 0.4989 880 24 s56532.1 A G 0.4818 880
001、因此,在使用maf过滤染色体包含24的数据时,应当指定染色体数目




 浙公网安备 33010602011771号
浙公网安备 33010602011771号