Nature Communications | 单细胞表观图谱破解颅神经发育疾病非编码变异之谜
摘要总结
这篇文章是2024年9月发表在《Nature Communications》杂志上的一篇研究,标题为“A cell type-aware framework for nominating non-coding variants in Mendelian regulatory disorders”。这篇文章通过整合小鼠胚胎颅运动神经元的单细胞染色质可及性、组蛋白修饰和基因表达数据,构建了细胞类型特异性的调控元件图谱,并应用于270个未解决的先天性颅神经失调症(CCDDs)家系的全基因组分析,发现了多个致病候选非编码变异。这对于解析孟德尔疾病中非编码变异的致病机制、提升罕见病诊断率具有重要意义。
研究背景
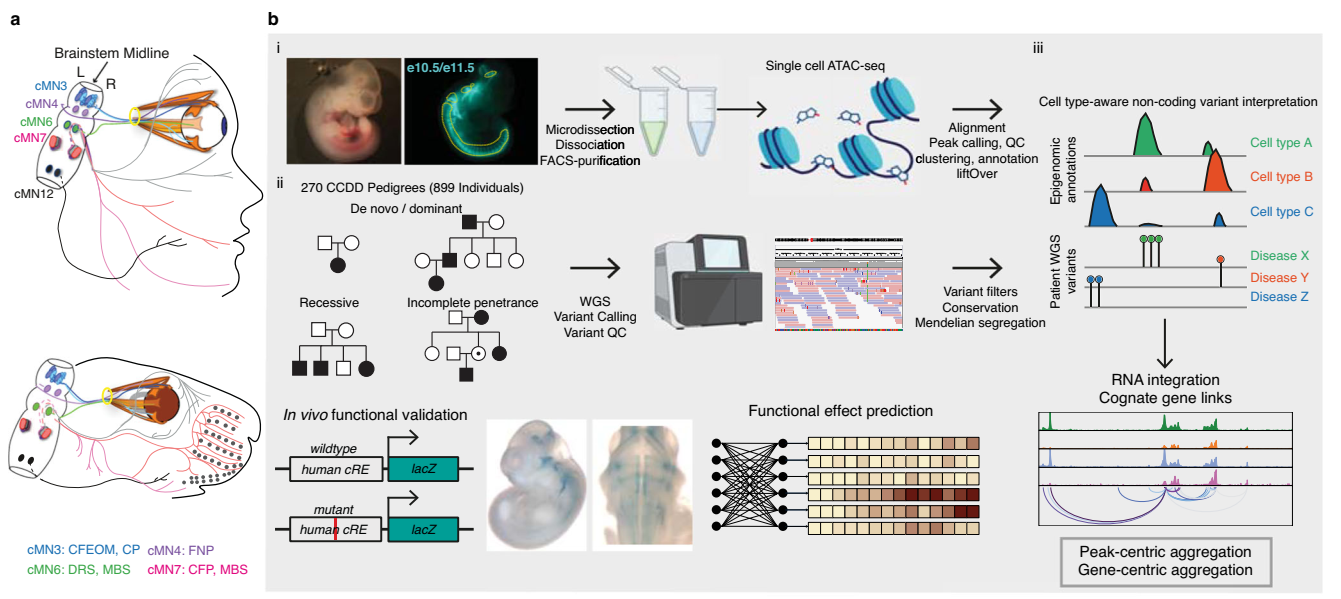
孟德尔疾病中约95%的病例由编码区变异解释,但仍有大量未解决病例可能与非编码调控变异相关。传统方法因非编码区功能注释不足、细胞类型特异性数据缺乏而受限,尤其发育关键期细胞难以获取。先天性颅神经失调症(CCDDs)由颅运动神经元(cMN)发育异常导致,其细胞类型明确且小鼠模型发育保守,为研究非编码变异提供了理想体系。
研究分析
研究团队通过以下步骤展开:
-
单细胞表观图谱构建:对小鼠胚胎发育关键期(e10.5-e11.5)的颅运动神经元及周边细胞进行单细胞ATAC-seq,获得86,089个细胞的高分辨率染色质可及性图谱,鉴定25.5万个调控元件,并通实验验证了75%的候选增强子活性。
-
跨物种功能关联:整合组蛋白修饰(H3K27ac)和单细胞RNA数据,建立调控元件与靶基因的关联,发现小鼠与人类增强子功能高度保守。
-
机器学习预测:训练卷积神经网络预测变异对染色质可及性的影响,成功验证已知致病位点(如GATA2非编码变异),并发现多个新候选(如EBF3、PHOX2A相关调控元件)。
研究结果
-
关键发现:
-
鉴定145,073个增强子-基因关联,发现44个增强子通过体内实验验证具有调控活性。
-
在CCDDs家系中提名MAFB、PHOX2A等已知疾病基因的非编码变异,以及MN1基因上游保守增强子缺失等新候选。
-
机器学习模型预测的变异效应与实验数据高度一致(Pearson’s r=0.84),且可解析发育阶段特异性影响(如cMN7在e10.5与e11.5的变异效应差异)。
-
-
临床意义:
-
发现EBF3基因非编码变异与多种眼肌表型相关,扩展了其表型谱。
-
在孤立性CCDDs病例中,多峰候选变异富集显著(OR=5.9),提示非编码变异的组织特异性效应。
-
亮点与局限
亮点:
-
首次构建颅运动神经元发育期高分辨率表观图谱,填补了早期神经发育调控数据的空白。
-
跨物种整合实验与计算模型,提升非编码变异注释的可靠性。
-
强调了非编码变异在孟德尔疾病中的重要性,改变了以往主要关注编码区变异的研究模式。
局限:
-
小鼠与人类发育时间窗差异可能影响调控元件保守性推断。
-
部分候选变异的功能验证仍需体内模型进一步确认。
-
当前框架对复杂结构变异和非保守元件的解析能力有限。
本文来自博客园,作者:橙子牛奶糖(陈文燕),转载请注明原文链接:https://www.cnblogs.com/chenwenyan/p/18788085



 浙公网安备 33010602011771号
浙公网安备 33010602011771号