遗传算法
遗传算法学习笔记
本学习笔记介绍了一种最优化函数值的一种方法 ---- 遗传算法
在本学习笔记中会先介绍遗传算法的用处及思想 然后分部分介绍其代码实现和具体做法 最后会给出完整代码
本学习笔记有借鉴Python手把手构建遗传算法(GA)实现最优化搜索 - FINTHON的部分 谢谢大佬的博客
欢迎各位评判指正
遗传算法
用处
遗传算法广泛应用于数学建模当中 ,用于寻找最优化函数值的问题(不仅仅是局限于寻找最大值问题)
在下文中使用样例:寻找 \( y = x_1^2 + x_2^2 + x_3^2 + x_4^2 \) 的最大值 \( \forall i , x_i \in \left [ 1,30 \right ] \)
显然 当 全部值为30时 函数值取到最大值
思想
遗传算法模仿了遗传算法中的各个过程,在一开始我们在区间内随机出一个size的种群,然后通过交叉和变异的方式产生下一代,评估每一代的个体的适应度函数,我们选择更适应的那些个体进入下一代(亦或者是那些适应度函数更优的个体拥有更大的概率进入下一代) ,在若干代的进化之下 , 就有大的概率找到最优解 / 偏优解
一些基本概念
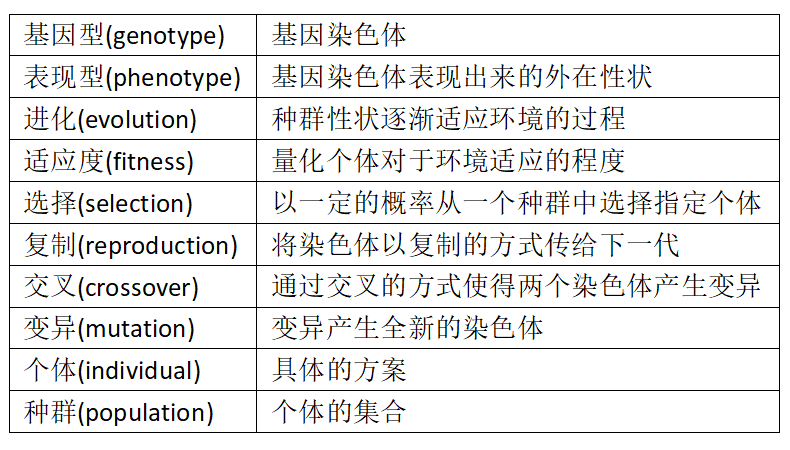
遗传算法的各个组成部分详解
初始化方法
在初始化方法当中 随机生成一个含popsize个个体的种群并获取初始种群中的各个信息
def __init__(self,parameter): #这里的parameter是一个list变量 #parameter = [cp,mp,max_gen,popsize,low,up] #cp ----> cross_possbility 交叉率 #mp ----> mutiply_possbility 变异率 #max_gen --> max_generation 最大繁殖代数 self.parameter = parameter low = self.parameter[4] up = self.parameter[5] self.bound = [] self.bound.append(low) self.bound.append(up) pop = [] #初始化一个种群 for i in range(self.parameter[3]): geneinfo = [] for pos in range(len(low)): geneinfo.append(random.randint(self.bound[0][pos], self.bound[1][pos])) fitness = self.evaluate(geneinfo) #每个初始的染色体都是一个字典 #字典里封装着一个基因的Gene类 和 适应度函数值 pop.append({'Gene':Gene(data=geneinfo), 'fitness': fitness}) self.pop = pop self.bestindividual = self.selectBest(self.pop)
评估适应度函数
也就是最终要最优化的函数 在本样例当中 fitness = \( y = x_1^2 + x_2^2 + x_3^2 + x_4^2 \)
def evaluate(self,geneinfo): x1 = geneinfo[0] x2 = geneinfo[1] x3 = geneinfo[2] x4 = geneinfo[3] func = x1 ** 2 +x2 ** 2 +x3 ** 2 +x4 ** 2 return func
选择最优的个体
选择一个种群当中适应度函数最高的个体
def selectBest(self,pop): #将种群按照适应度函数fitness函数从大到小排序 #并将最大适应度函数的个体返回 s_inds = sorted(pop, key=itemgetter("fitness"), reverse=True) return s_inds[0]
选择个体
按照轮盘赌的方式来选择个体
使用random.random()生成一个0-1之间的数字
random.random() * fit_sum 计算出该值位于某个个体的概率区间
可以看出个体适应度越大的个体被选中的概率也就越大
def selection(self,individuals,k): s_inds = sorted(individuals, key=itemgetter("fitness"), reverse=True) sum_fits = sum(ind['fitness'] for ind in individuals) chosen = [] for i in range(k): #random.random()返回一个0-1之间的值 #我们使用轮盘赌选择的方式来进行选择 #即u在哪个区间则选择那个区间内的个体 #由于random.random()是均匀产生 #可以知道fitness更大的值拥有更大的区间 #则拥有更大的被选择的概率 u = random.random() * sum_fits sum_ = 0 for ind in s_inds: sum_ += ind['fitness'] if sum_ >= u: chosen.append(ind) break #按照fitness从小到大的方式排序chosen的种群 chosen = sorted(chosen, key=itemgetter("fitness"), reverse = False) return chosen
交叉变异
可以选择单点位交叉变异亦或是双点位交叉变异的方式
可以看见单点位变异与双点位交叉变异均可以有效的完成变异

def crossoperate(self,offspring): #使用双点位交叉的方式进行交叉变异 #在实际中双点位交叉和单点位交叉的变异程度都是可以的 dim = len(offspring[0]['Gene'].data) geneinfo1 = offspring[0]['Gene'].data geneinfo2 = offspring[1]['Gene'].data #如果基因长度只有1 进行特判 if dim == 1: pos1 = 1 pos2 = 1 else: pos1 = random.randrange(1,dim) pos2 = random.randrange(1,dim) newoff1 = Gene(data = []) newoff2 = Gene(data = []) temp1 = [] temp2 = [] for i in range(dim): if min(pos1 , pos2) <= i <= max(pos1 , pos2): temp1.append(geneinfo1[i]) temp2.append(geneinfo2[i]) else: temp1.append(geneinfo2[i]) temp2.append(geneinfo1[i]) newoff1.data = temp1 newoff2.data = temp2 return newoff1,newoff2
变异
本文提供两种变异方式 : 逆位变异 和 基本位变异
逆位变异:将单条染色体中的两个基本位进行互换
基本位变异:将某个染色体中的某个位置进行随机变换
其解决了仅交叉变异时变异数不足导致的基因型丰富度不足的问题
def mutation(self,crossoff,bound): #在这里使用基本位单点变异 dim = len(crossoff.data) if dim == 1: pos = 0 else: pos = random.randrange(0,dim) crossoff.data[pos] = random.randint(bound[0][pos], bound[1][pos]) return crossoff ''' def reversal_mutation(self,crossoff): #提供逆位变异的方法 dim = len(crossoff.data) if dim == 1: pos1 = 0 pos2 = 0 else: pos1 = random.randrange(0,dim) pos2 = random.randrange(0,dim) #交换基因位 bridge = crossoff.data[pos2] crossoff.data[pos2] = crossoff.data[pos1] crossoff.data[pos1] = bridge return crossoff '''
主函数部分
在写好各部分的函数之后 编写主函数部分
def GA_main(self): popsize = self.parameter[3] print("Evolution start") for g in range(self.parameter[2]): print("######## Generation {} #######".format(g)) selectpop = self.selection(self.pop, popsize) nextoff = [] while len(nextoff) != popsize: offspring = [selectpop.pop() for _ in range(2)] #判定是否需要交叉 if random.random() < cp: crossoff1 , crossoff2 = self.crossoperate(offspring) #判定是否需要变异 if random.random() < mp: muteoff1 = self.mutation(crossoff1, self.bound) muteoff2 = self.mutation(crossoff2, self.bound) fit_muteoff1 = self.evaluate(muteoff1.data) fit_muteoff2 = self.evaluate(muteoff2.data) nextoff.append({'Gene':muteoff1, 'fitness':fit_muteoff1}) nextoff.append({'Gene':muteoff2, 'fitness':fit_muteoff2}) else: fit_crossoff1 = self.evaluate(crossoff1.data) fit_crossoff2 = self.evaluate(crossoff2.data) nextoff.append({'Gene':crossoff1, 'fitness':fit_crossoff1}) nextoff.append({'Gene':crossoff2, 'fitness':fit_crossoff2}) else: nextoff.extend(offspring) self.pop = nextoff fits = [ind['fitness'] for ind in self.pop] best_ind = self.selectBest(self.pop) if best_ind['fitness'] > self.bestindividual['fitness']: self.bestindividual = best_ind print("Best individual found is {}, {}".format(self.bestindividual['Gene'].data, self.bestindividual['fitness'])) print("Max fitness of current pop: {}".format(max(fits))) print("------ End of (successful) evolution ------")
最终代码
# -*- coding: utf-8 -*- """ Created on Wed Jan 19 20:47:18 2022 @author: Rbrq """ import random from operator import itemgetter class Gene: def __init__(self, **data): self.__dict__.update(data) self.size = len(data['data']) class GA: def __init__(self,parameter): #这里的parameter是一个list变量 #parameter = [cp,mp,max_gen,popsize,low,up] #cp ----> cross_possbility 交叉率 #mp ----> mutiply_possbility 变异率 #max_gen --> max_generation 最大繁殖代数 self.parameter = parameter low = self.parameter[4] up = self.parameter[5] self.bound = [] self.bound.append(low) self.bound.append(up) pop = [] #初始化一个种群 for i in range(self.parameter[3]): geneinfo = [] for pos in range(len(low)): geneinfo.append(random.randint(self.bound[0][pos], self.bound[1][pos])) fitness = self.evaluate(geneinfo) #每个初始的染色体都是一个字典 #字典里封装着一个基因的Gene类 和 适应度函数值 pop.append({'Gene':Gene(data=geneinfo), 'fitness': fitness}) self.pop = pop self.bestindividual = self.selectBest(self.pop) def evaluate(self,geneinfo): x1 = geneinfo[0] x2 = geneinfo[1] x3 = geneinfo[2] x4 = geneinfo[3] func = x1 ** 2 +x2 ** 2 +x3 ** 2 +x4 ** 2 return func def selectBest(self,pop): #将种群按照适应度函数fitness函数从大到小排序 #并将最大适应度函数的个体返回 s_inds = sorted(pop, key=itemgetter("fitness"), reverse=True) return s_inds[0] def selection(self,individuals,k): s_inds = sorted(individuals, key=itemgetter("fitness"), reverse=True) sum_fits = sum(ind['fitness'] for ind in individuals) chosen = [] for i in range(k): #random.random()返回一个0-1之间的值 #我们使用轮盘赌选择的方式来进行选择 #即u在哪个区间则选择那个区间内的个体 #由于random.random()是均匀产生 #可以知道fitness更大的值拥有更大的区间 #则拥有更大的被选择的概率 u = random.random() * sum_fits sum_ = 0 for ind in s_inds: sum_ += ind['fitness'] if sum_ >= u: chosen.append(ind) break #按照fitness从小到大的方式排序chosen的种群 chosen = sorted(chosen, key=itemgetter("fitness"), reverse = False) return chosen def crossoperate(self,offspring): #使用双点位交叉的方式进行交叉变异 #在实际中双点位交叉和单点位交叉的变异程度都是可以的 dim = len(offspring[0]['Gene'].data) geneinfo1 = offspring[0]['Gene'].data geneinfo2 = offspring[1]['Gene'].data #如果基因长度只有1 进行特判 if dim == 1: pos1 = 1 pos2 = 1 else: pos1 = random.randrange(1,dim) pos2 = random.randrange(1,dim) newoff1 = Gene(data = []) newoff2 = Gene(data = []) temp1 = [] temp2 = [] for i in range(dim): if min(pos1 , pos2) <= i <= max(pos1 , pos2): temp1.append(geneinfo1[i]) temp2.append(geneinfo2[i]) else: temp1.append(geneinfo2[i]) temp2.append(geneinfo1[i]) newoff1.data = temp1 newoff2.data = temp2 return newoff1,newoff2 def mutation(self,crossoff,bound): #在这里使用基本位单点变异 dim = len(crossoff.data) if dim == 1: pos = 0 else: pos = random.randrange(0,dim) crossoff.data[pos] = random.randint(bound[0][pos], bound[1][pos]) return crossoff ''' def reversal_mutation(self,crossoff): #提供逆位变异的方法 dim = len(crossoff.data) if dim == 1: pos1 = 0 pos2 = 0 else: pos1 = random.randrange(0,dim) pos2 = random.randrange(0,dim) #交换基因位 bridge = crossoff.data[pos2] crossoff.data[pos2] = crossoff.data[pos1] crossoff.data[pos1] = bridge return crossoff ''' def GA_main(self): popsize = self.parameter[3] print("Evolution start") for g in range(self.parameter[2]): print("######## Generation {} #######".format(g)) selectpop = self.selection(self.pop, popsize) nextoff = [] while len(nextoff) != popsize: offspring = [selectpop.pop() for _ in range(2)] #判定是否需要交叉 if random.random() < cp: crossoff1 , crossoff2 = self.crossoperate(offspring) #判定是否需要变异 if random.random() < mp: muteoff1 = self.mutation(crossoff1, self.bound) muteoff2 = self.mutation(crossoff2, self.bound) fit_muteoff1 = self.evaluate(muteoff1.data) fit_muteoff2 = self.evaluate(muteoff2.data) nextoff.append({'Gene':muteoff1, 'fitness':fit_muteoff1}) nextoff.append({'Gene':muteoff2, 'fitness':fit_muteoff2}) else: fit_crossoff1 = self.evaluate(crossoff1.data) fit_crossoff2 = self.evaluate(crossoff2.data) nextoff.append({'Gene':crossoff1, 'fitness':fit_crossoff1}) nextoff.append({'Gene':crossoff2, 'fitness':fit_crossoff2}) else: nextoff.extend(offspring) self.pop = nextoff fits = [ind['fitness'] for ind in self.pop] best_ind = self.selectBest(self.pop) if best_ind['fitness'] > self.bestindividual['fitness']: self.bestindividual = best_ind print("Best individual found is {}, {}".format(self.bestindividual['Gene'].data, self.bestindividual['fitness'])) print("Max fitness of current pop: {}".format(max(fits))) print("------ End of (successful) evolution ------") if __name__ == "__main__": #在主函数中确定各个参数的值 cp , mp, max_gen , popsize = 0.8 , 0.1 , 1000 , 100 up = [30, 30, 30, 30] low = [1, 1, 1, 1] parameter = [cp,mp,max_gen,popsize,low,up] max_func = GA(parameter) max_func.GA_main()



 浙公网安备 33010602011771号
浙公网安备 33010602011771号