GEOquery
GEOquery 是 bioconductor 项目下的一个R包
官网:http://www.bioconductor.org/packages/release/bioc/html/GEOquery.html
下载安装:
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("GEOquery")
GEO 是由NCBI负责维护的一个数据库,期初是有芯片数据,后来增加了甲基化芯片和高通量测序数据。
只要看到文献里面有提到 GEO 数据库,都可以通过这个R包来进行批量下载,其实就是网页版的一个API调用而已。
GEO 数据库基础知识(有另一篇博文专门介绍GEO数据库)
GEO Platform (GPL) 芯片平台
GEO Sample (GSM) 样本ID号
GEO Series (GSE) study 的ID号
GEO Dataset (GDS) 数据集的ID号
GEOquery 用法
常用函数,以及每个函数返回的对象该如何处理即可。
getGEO() 从GEO数据库下载数据或者打开本地数据
返回值:GDS / GSE / GSM / GPL 具体返回值取决于GEO的参数

GEO参数 决定下载的数据种类,GDS / GSE / GSM / GPL
filename参数 如果已经下载好了文件,直接读取
destdir参数 下载文件存放的地址,默认的是工作目录
GSEMatrix参数 若为TRUE,则下载Matrix文件;若为FALSE,则下载SOFT文件。默认为TRUE。
getGPL参数 若为TRUE,则下载GPL注释文件;若为FALSE,则不下载。默认为TRUE。
getGEOSuppFiles() 下载 supplementary data,包含 raw data
返回值:supplementary data

GEO参数 下载GSE的supplemental files
makeDirectory参数 若为TRUE,则默认新建文件夹并下载到里面;若为FALSE,则直接下载到工作目录。默认为TRUE。
baseDir参数 指定下载目录,默认为工作目录
getGEOfile()
gunzip() 解压gz格式的文件
返回值:从gz压缩文件中提起的文件。
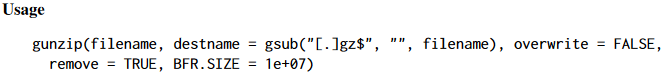
filename参数 需要解压缩的文件名,可以为 list
destname参数 解压缩之后的文件名,默认去除 .gz$
overwrite参数 是否覆盖重名文件,默认为FALSE
remove参数 解压完是否删除原文件,默认为TRUE
这三个函数根据上面的四种ID下载数据的时候,返回的对象是不同的。
首先介绍 getGEO() 函数
-- gse1009 <- getGEO("GSE1009", destdir = ".") # 根据GSE号下载数据,下载 ***_series_matrix.txt.gz 文件
-- gds858 <- getGEO("GDS858", destdir = ".") # 根据GDS号下载数据,下载 soft 文件。 如果使用了 GSEMatrix = TRUE 这个参数,那么除了下载soft文件还会下载表达量矩阵文件,可以直接用read.table()函数读取
-- gpl96 <- getGEO("GPL96", destdir = ".") # 根据GPL号下载的是芯片设计的信息
下载的文件都会保存在本地,destdir 参数用来指定下载地址。
getGEO()的比较重要的参数有:
GSEmatrix = TRUE
AnnotGPL = FALSE
getGPL = TRUE
根据返回对象的不同,针对返回对象的方法也不一样
下载GSE返回对象的处理函数
即直接根据GSE号返回的对象:gse1009
相应的处理函数有:
geneNames
sampleNames
pData
exprs
下面的操作得到一个数值矩阵
expr <- read.table("GSE26253_series_matrix.txt", comment.char = "!", stringAsFactors = F)
下载GDS返回对象的处理函数
gds858返回的对象很复杂
用 Table(gds858) 可以得到表达矩阵
用 Meta(gds858) 可以得到描述信息
用 GDS2eSet() 函数把它转变为 expression set 对象
library(GEOquery) gds858 <- getGEO("GDS858", destdir = ".") names(Meta(gds858)) Table(gds858)[1:5, 1:5]
eset <- GDS2eSet(gds858, do.log2 = TRUE)
g4100 <- GDS2eSet(getGEO("GDS4100"))
e4100 <- exprs(g4100) # 这样e4100就是一个数值矩阵了,可以进行下游分析。
下载GPL返回对象的处理函数
根据GPL号下载返回的对象也用 Table() meta()处理
library(GEOquery) gpl <- getGEO("GPL96", destdir = ".") names(Meta(gpl96)) Table(gpl96)[1:10, 1:4] ## 下面这个就是芯片ID的基因注释信息 Table(gpl96)[1:10, c("ID", "GB_LIST", "Gene.Symbol", "Entres.Gene")]
getGEO() 函数除了可以下载数据,也可以打开本地数据,使用参数 filename 指定本地数据文件
gds858 <- getGEO(filename = "GDS858.soft.gz")


 浙公网安备 33010602011771号
浙公网安备 33010602011771号