miRAN 分析以及mRNA分析
一些参考资料
http://www.360doc.com/content/17/0528/22/19913717_658086490.shtml
https://www.cnblogs.com/triple-y/p/9338890.html
一、对miRNA进行分析
1、bowtie比对
"bowtie -q -v 2 -l 10 -k 15 /data/pub/shehb/Spinach_genome/spinach_genome_v1.fa "+fq+" -S "+fq+".sam >mapping.info"
2、HTSeq 计算reads数量 (详细:http://www.chenlianfu.com/?p=2438)
(1)HTSeq是对有参考基因组的转录组测序数据进行表达量分析的,其输入文件必须有SAM和GTF文件。
(2)一般情况下HTSeq得到的Counts结果会用于下一步不同样品间的基因表达量差异分析,而不是一个样品内部基因的表达量比较。因此,HTSeq设置了-a参数的默认值10,来忽略掉比对到多个位置的reads信息,其结果有利于后续的差异分析。
(3)输入的GTF文件中不能包含可变剪接信息,否则HTSeq会认为每个可变剪接都是单独的基因,导致能比对到多个可变剪接转录本上的reads的计算结果是ambiguous,从而不能计算到基因的count中。即使设置-i参数的值为transcript_id,其结果一样是不准确的,只是得到transcripts的表达量。
run = "htseq-count -s no -t mRNA -i ID "+sam+" ./Spinach_genome/spinach_gene_v1miRNA.gff3 > "+sam+"count_out.txt"
参数:
-f | --format default: sam 设置输入文件的格式,该参数的值可以是sam或bam
-r | --order default: name 设置sam或bam文件的排序方式,该参数的值可以是name或pos。,但HTSeq推荐使用name排序,且一般比对软件的默认输出结果也是按name进行排序的。
-s | --stranded default: yes 设置是否是链特异性测序。该参数的值可以是yes,no或reverse。no表示非链特异性测序;若是单端测序,yes表示read比对到了基因的正义链上;若是双末端测序,yes表示read1比对到了基因正义链上,read2比对到基因负义链上;reverse表示双末端测序情况下与yes值相反的结果。根据说明文件的理解,一般选择no, yes是成对的reads数量-a | --a default: 10 忽略比对质量低于此值的比对结果。在0.5.4版本以前该参数默认值是0。 -t | --type default: exon 程序会对该指定的feature(gtf/gff文件第三列)进行表达量计算,而gtf/gff文件中其它的feature都会被忽略。 比如:在mRNA中,选择exon,表示只计算exon的reads数量-i | --idattr default: gene_id 设置feature ID是由gtf/gff文件第9列那个标签决定的;若gtf/gff文件多行具有相同的feature ID,则它们来自同一个feature,程序会计算这些features的表达量之和赋给相应的feature ID。 看基因前面的标志是什么-m | --mode default: union 设置表达量计算模式。该参数的值可以有union, intersection-strict and intersection-nonempty。这三种模式的选择请见上面对这3种模式的示意图。从图中可知,对于原核生物,推荐使用intersection-strict模式;对于真核生物,推荐使用union模式。 -o | --samout 输出一个sam文件,该sam文件的比对结果中多了一个XF标签,表示该read比对到了某个feature上。 -q | --quiet 不输出程序运行的状态信息和警告信息。 -h | --help 输出帮助信息。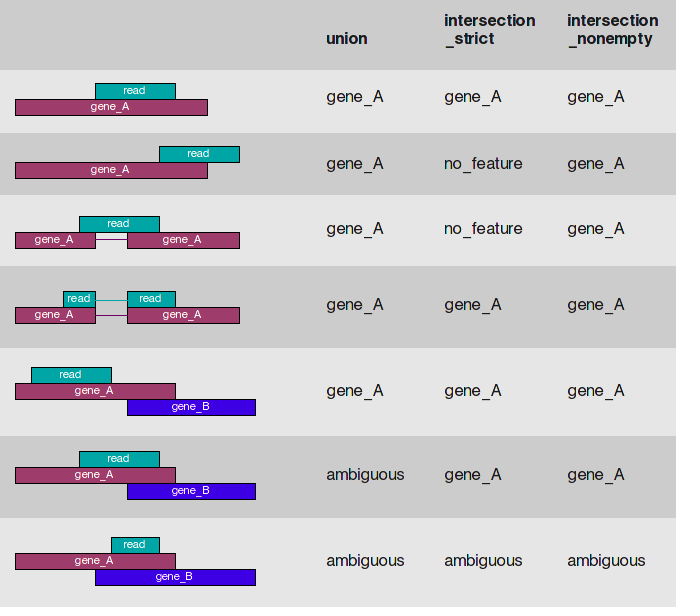
1 paste *.txt |awk '{printf $1"\t";for(i=2;i<=18;i+=2)printf $i"\t";printf $i"\n"}' |less -S 2 3 ##18 为总共有多少列
3、对于有重复的样本用DESeq2进行分析
自认为感觉不是很好安装包 ,我用下前面方法安装上去的
若R安装包出现package ‘xx’ is not available (for R version 3.4.2)的问题时:
用以下方法安装包
source("http://bioconductor.org/biocLite.R")
biocLite("包名")
DESeq2对于输入数据的要求
1.DEseq2要求输入数据是由整数组成的矩阵。
2.DESeq2要求矩阵是没有标准化的。
DESeq2进行差异表达分析
DESeq2包分析差异表达基因简单来说只有三步:构建dds矩阵,标准化,以及进行差异分析。
1、构建dds矩阵
需要:(1)countData, 是readscount计算后,合并的形成的矩阵。行为基因名字,列为样本
(2)colData, 样本信息,标出哪些是control,哪些是treat。第一列样品名称(其实就是countData第一行的名称),第二列是样品处理情况(对照还是处理等),condition的类型是一个factor。
(3)差异比较矩阵 即上述代码中的design。 差异比较矩阵就是告诉差异分析函数是要从要分析哪些变量间的差异,简单说就是说明哪些是对照哪些是处理。
library(S4Vectors)
library(stats4)
library(BiocGenerics)
library(parallel)
library(IRanges)
library(GenomicRanges)
library(GenomeInfoDb)
library(SummarizedExperiment)
library(Biobase)
library(DelayedArray)
library(matrixStats)
library(DelayedArray)
library(DESeq2)
library(ggplot2)
setwd("/Users/hongbingshe/Desktop")
condition <- factor(c("XX","XX","XX","XY","XY","XY"))
countData <- read.table("XX_XYmiRNA.count_out",header = T,sep = "\t",row.names = "X") ##open file
colData <- data.frame(row.names = colnames(countData),condition)
dds <- DESeqDataSetFromMatrix(countData,DataFrame(condition),design = ~ condition)
head(dds) ##
dds2 <- DESeq(dds) ###normalize rlogTransformation(dds2). ???
resultsNames(dds2) ###objective name
res <- results(dds2)
summary(res) ###infomation
table(res$padj<0.05) ##number of true 小于0.05 的基因个数
res <- res[order(res$padj),]
diff_gene_deseq2 <- subset(res,padj <0.05 & (log2FoldChange >1 |log2FoldChange < -1)) ###FC >1, P <0.05
differ_gene_deseq2 <- row.names(diff_gene_deseq2)
resdata <- merge(as.data.frame(res),as.data.frame(counts(dds2,normalize=TRUE)),by="row.names",sort=FALSE)
write.csv(resdata,file = "/Users/hongbingshe/Desktop/XX_XYmiRNA.cvs",row.names = F) ##output file
resdata[which(resdata$padj < 0.05 & resdata$log2FoldChange > 1 ),'significant'] <-'up'
resdata[which(resdata$padj < 0.05 & resdata$log2FoldChange < -1 ),'significant'] <-'down'
resdata[!resdata$significant%in%c('up','down'),'significant'] <- 'no'
p <- ggplot(resdata,aes(resdata$log2FoldChange,-log10(resdata$padj))) + geom_point(aes(color=significant),size=0.5) +scale_color_manual(limits=c('up','down','no'),values = c('blue','red','gray40')) +labs(x="log2(Fold Change)",y="-log10(P-value)")
p <- p + theme(panel.grid.major = element_line(color="gray",size=0.2), panel.background = element_rect(color = "black",fill = "transparent")) +geom_vline(xintercept = c(-1,1),color="gray",linetype=2,size=0.5)+geom_hline(yintercept = -log10(1),color='gray',linetype=2,size=0.5)
p <- p + theme(legend.title = element_blank(),legend.key = element_rect(fill='transparent'),legend.background = element_rect(fill = 'transparent'))
p <- p + xlim(-4,4) +ylim(0,40) ##X scale
p
ggsave("XY_XXmiRNAvalcano_plot.png",p,width = 4.5,height=5.5)
标记出你期望的基因名字
(1)首先需要做一个数据框. 假如为test
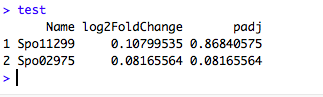
(2)导入 ggrepel 包
library(ggrepel)
(3)添加图层
p + geom_text_repel(data =test,aes(test$log2FoldChange,-log10(test$padj),label=test$Name))
或者用p +geom_label_repel(data = test,aes(test$log2FoldChange,-log10(test$padj),label=test$Name))
二、mRNA分析
选用hisat2+HTseq+DEseq2
1、hisat2:
run = "hisat2 -x ./Spinach_genome/spinach_genome_v1.fa -p 4 -1 "+i+" -2 "+j+" -S "+Out
2、HTSeq2
htseq-count -f bam -r name -s no -a 10 -t exon -i ID -m intersection-nonempty yourfile_name.bam ~/reference/hisat2_reference/Homo_sapiens.GRCh38.86.chr_patch_hapl_scaff.gtf > counts.txt
3、DEseq2
同上
关注下方公众号可获得更多精彩





 浙公网安备 33010602011771号
浙公网安备 33010602011771号