扩增子数据分析工具安装
一、配置conda环境(进入cutadapt环境)
1 conda activate cutadapt
不会安装conda和用conda安装cutadapt可以参考一下我前两篇随笔:
https://www.cnblogs.com/zengy-bioinfostudy/p/14914828.html
https://www.cnblogs.com/zengy-bioinfostudy/p/14929008.html
或者其它博客或者直接找官网文档。
二、安装blast工具
进入ncbi下载blast本地工具的下载站:https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
选择linux版本的.tar.gz文件,下载到本地,然后用xftp或其它你喜欢用的工具上传到你的服务器上
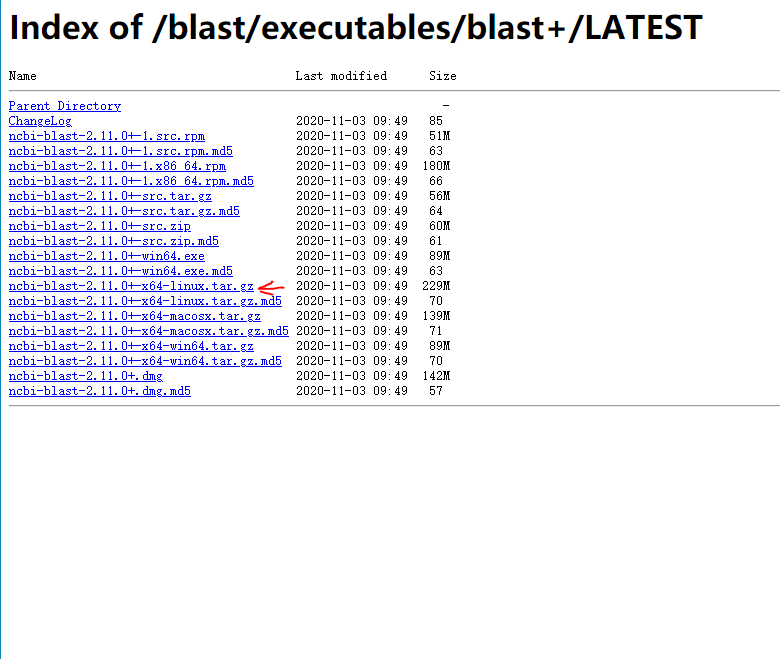
进入linux服务器,cd到你上传文件的目录
给文件加个可执行权限,然后tar解压
1 sudo chmod +x ncbi-blast-2.11.0+-x64-linux.tar.gz 2 3 tar -zxvf ncbi-blast-2.11.0+-x64-linux.tar.gz
把安装目录添加进$PATH下:
1 vim ~/.bashrc
在最后一行添加:
1 export PATH=/解压后的文件夹的路径/ncbi-blast-2.11.0+/bin:$PATH
我这里是放在了自己家目录下,毕竟我用的不是root用户安装的(狗头)
检查blast命令是否正常:
命令行键入blast,如果弹出如下界面,说明工作正常:
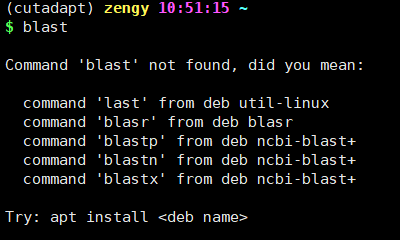
详细使用说明可以参考这位大佬的知乎:
https://zhuanlan.zhihu.com/p/141644734
三、安装biom工具
刘永鑫老师我觉得是讲的很详细了,这里我就不再赘述了:
https://blog.csdn.net/woodcorpse/article/details/84678543
四、获取vsearch工具
其实这个应该用官方给的方法安装的,但是我的系统老是在make的时候出错,找了半天找不到解决方法,于是就直接用了可执行文件,安装的方法请参考官方github网站:
https://github.com/torognes/vsearch
我的获取方法是在github的发行版本那下载对应版本的可执行文件然后上传到服务器上,最后添加进$PATH下(添加方法上面安装blast的时候有提到,这里不提了)
https://github.com/torognes/vsearch/releases
OK,到这里,所有我们需要的工具就全部安装完成了!!芜湖!!!
接下来,就可以愉快的开始分析扩增子数据喽!!!(虽然我还没学会各个工具的使用方法)



 浙公网安备 33010602011771号
浙公网安备 33010602011771号