nnUNet使用指南(三):nnUNet对数据的预处理
数据预处理步骤
根据nnUNet框架,三维医学图像分割的通用预处理可以分为四步,分别是数据格式的转换,裁剪crop,重采样resample以及标准化normalization。
1.数据格式的转化
常见的医学图像格式有DICOM(后缀名为.dcm),MHD(后缀名为.mhd和.raw)以及NIFTY(后缀名为.nii或.nii.gz)。
这几种格式都不太方便直接进行操作,一般都使用对应的Python库将数据进行读取后,转换成numpy数组后再进行后续处理。
nnUNet中给出了一种建议的目标数据格式,将每一个病例的数据,都存成一个四维numpy数组(npz)以及与之对应的pickle文件(pkl)。
numpy的文件存储.npy .npz 文件详解
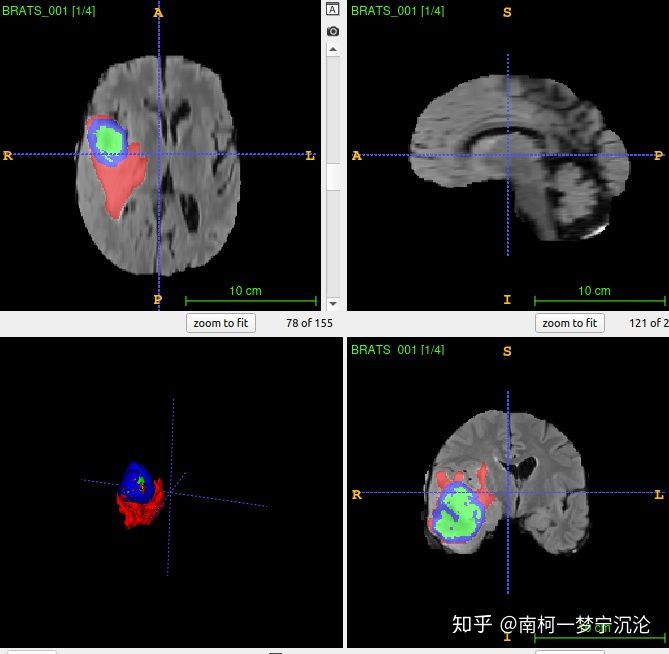
四维数组array(C、X、Y、Z)中,C维度的最后一个array[-1,:,:,:]存储的是分割标注结果。
而C维度的前面存储不同模态的数据,如MRI数据中有FLAIR, T1w, t1gd, T2w等四种模态,
array[0,:,:,:]表示FLAIR序列成像的强度数据,array[1,:,:,:]表示T1加权的强度数据,以此类推。
如果仅单模态,则四维数组第一维度长度仅为2,分别表示影像数据以及标注数据。
四维数组array的后三个维度代表x,y,z三个坐标表示的三维数据,对于原始影像数据,值大小代表强度,
而对于标注结果,后三个维度的三维数据值分别为0,1,2……表示不同的标注类别。
在后续的代码中,为了简便,将不同模态的原始图像与分割标注分开,使用data(CXYZ)代表四维图像数据,使用seg(XYZ)代表三维标注数据。
而pickle文件中存储该医学影像中其它的重要信息,是对numpy数组提供信息的补充。包含spacing,direction,origin等信息。
2.图像裁剪Crop
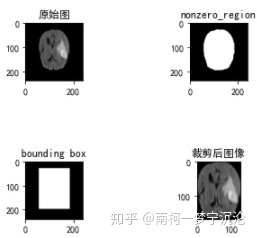
图像裁剪就是将三维的医学图像裁剪到它的非零区域,具体方法就是在图像中寻找一个最小的三维bounding box,
该bounding box区域以外的值为0,使用这个bounding box对图像进行裁剪。
相比裁剪前,裁剪后的图像对于最后的分割结果没有影响,但是却可以减小图像尺寸,避免无用的计算,提高计算效率。
这个操作对于部分数据集如大脑数据集比较有效,这些数据集中外围全黑的背景相对较多。
裁剪在nnUNet的实现中可以分为3步。
第一步
根据四维图像数据data(C,X,Y,Z)生成三维的非零模板nonzero_mask,标示图像中哪些区域是非零的 。
不同的模态都有对应的三维数据,产生不同的三维nonzero_mask,而整个四维图像的非零模板为各个模态非零模板的并集。
最后调用scipy库的binary_fill_holes函数对生成的nonzero_mask进行填充。
from scipy.ndimage import binary_fill_holes
# data.shape[1:]取x,y,z三维,bool型,初始全为false的三维框nonzero_mask
nonzero_mask = np.zeros(data.shape[1:], dtype=bool)
# 取data中每一个channel
for c in range(data.shape[0]):
# this mask 表示当前这个channel的三维图像框,将data中该channel的图像
# 不等于0的地方标记为True,等于0的地方标记为False,并赋给this mask
this_mask = data[c] != 0
# 对三维框和当前框取并集,只要有True的地方都标记为True
# nparray的并集(|):True + True = True,True + False = True,False + False = False
# nparray的交集(&):True + True = True,True + False = False,False + False = False
nonzero_mask = nonzero_mask | this_mask
# 最后得到的总的三维框包括了在所有channel中有true的地方,就可以包括所有非0区域
# 用binary_fill_holes对该非零区域去洞填充
nonzero_mask = binary_fill_holes(nonzero_mask)
第二步
根据生成的非零模板,确定用于裁剪的bounding_box大小和位置,在代码中就是要找到nonzero_mask在x,y,z三个坐标轴上值为1的最小坐标值以及最大坐标值。
def get_bbox_from_mask(nonzero_mask, outside_value=0):
mask_voxel_coords = np.where(nonzero_mask != outside_value)
minzidx = int(np.min(mask_voxel_coords[0]))
maxzidx = int(np.max(mask_voxel_coords[0])) + 1
minxidx = int(np.min(mask_voxel_coords[1]))
maxxidx = int(np.max(mask_voxel_coords[1])) + 1
minyidx = int(np.min(mask_voxel_coords[2]))
maxyidx = int(np.max(mask_voxel_coords[2])) + 1
return [[minzidx, maxzidx], [minxidx, maxxidx], [minyidx, maxyidx]]
第三步
根据bounding_box对该张图像的每个模态依次进行裁剪,然后重新组合在一起。
bbox = [[minzidx, maxzidx], [minxidx, maxxidx], [minyidx, maxyidx]]
resizer = (slice(bbox[0][0], bbox[0][1]),
slice(bbox[1][0], bbox[1][1]),
slice(bbox[2][0], bbox[2][1]))
cropped_data = []
for c in range(data.shape[0]):
cropped = data[c][resizer]
cropped_data.append(cropped[None])
data = np.vstack(cropped_data)
在对原始数据裁剪完毕之后,使用同样的bounding box对分割标注seg进行裁剪,具体步骤与上述代码的第三步一致。
注意到,nnUNet在对标注图像seg进行裁剪之后,还额外利用了nonzero_mask的信息,将nonzero_mask区域以外的背景标签值,从0修改为-1。
non_zero_label = -1
seg[(seg == 0) & (nonzero_mask == 0)] = nonzero_label
这样一来, seg中值为0和-1的都代表背景, 只是值为0的代表图像中值不为0的背景, 值为-1的代表图像中值为0的背景.
这样做可在后续的处理中,用seg<0迅速指示图像中的nonzero region.
在normalization中, 如果图像尺寸减小了1/4以上,就需要利用这个信息,仅在nonzero region进行normalization。
重采样Resample
未完待续



 浙公网安备 33010602011771号
浙公网安备 33010602011771号