聚类-分层聚类
1.分层聚类的介绍
分层聚类法(hierarchical cluster method)一译“系统聚类法”。聚类分析的一种方法。其做法是开始时把每个样品作为一类,然后把最靠近的样品(即距离最小的群品)首先聚为小类,再将已聚合的小类按其类间距离再合并,不断继续下去,最后把一切子类都聚合到一个大类。
一般来说,当考虑聚类效率时,我们选择平面聚类,当平面聚类的潜在问题(不够结构化,预定数量的聚类,非确定性)成为关注点时,我们选择层次聚类。 此外,许多研究人员认为,层次聚类比平面聚类产生更好的聚类.
层次聚类(Hierarchical Clustering)是聚类算法的一种,通过计算不同类别数据点间的相似度来创建一棵有层次的嵌套聚类树。在聚类树中,不同类别的原始数据点是树的最低层,树的顶层是一个聚类的根节点。创建聚类树有自下而上合并和自上而下分裂两种方法。
层次聚类方法对给定的数据集进行层次的分解,知道某种条件满足为止,具体又可分为:
凝聚的层次聚类:AGNES算法
一种自底向上的策略,首先将每一个对象作为一个簇,然后合并这些原子簇为越来越大的簇,直到某个终止条件被满足.
分层的层次聚类:DIANA算法
采用自顶向下的策略,他首先将所有对象置于一个簇中,然后逐渐细分为越来越小的簇,直到达到某个终止条件.
举例:
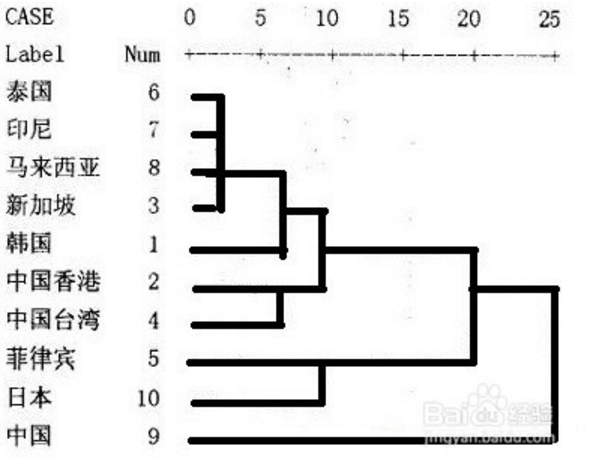
由上图可知,给定不同的距离,可以得到不同的分类,比如,23,则分为两类,中国和其他国家和地区;17,则可分成三类,中国单独一类,菲律宾和日本一类,其余国家和地区为一类。
自底向上的合并算法:

2.两个组合数据点之间的距离:
(1)Single Linkage 最小距离
方法是将两个组合数据点中距离最近的两个数据点间的距离作为这两个组合数据点的距离。
这种方法容易受到极端值的影响。两个很相似的组合数据点可能由于其中的某个极端的数据点距离较近而组合在一起。
(2)Complete Linkage 最远距离
complete Linkage的计算方法与Single Linkage相反,将两个组合数据点中距离最远的两个数据点间的距离作为这两个组合数据点的距离。
Complete Linkage的问题也与Single Linkage相反,两个相似的组合数据点可能由于其中的极端值距离较远而无法组合在一起。
(3)Average Linkage 平均距离
Average Linkage的计算方法是计算两个组合数据点中的每个数据点与其他所有数据点的距离。
将所有距离的均值作为两个组合数据点间的距离。
这种方法计算量比较大,但结果比前两种方法更合理。
我们使用Average Linkage计算组合数据点间的距离。下面是计算组合数据点(A,F)到(B,C)的距离,这里分别计算了(A,F)和(B,C)两两间距离的均值。

(4)ward linkage 离差平方和
其实这个也算是平均距离中的一种
两个集合中样本间两两距离的平方和
参考以下文章:
http://blog.sciencenet.cn/blog-2827057-921772.html
3.分层聚类的案例
瑞士卷案例
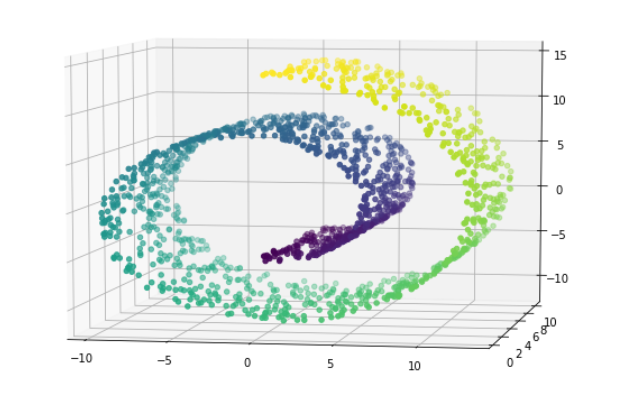
1 import warnings 2 warnings.filterwarnings('ignore') 3 import numpy as np 4 import matplotlib.pyplot as plt 5 %matplotlib inline 6 from sklearn import datasets 7 # 凝聚聚类:自下而上聚类 8 from sklearn.cluster import AgglomerativeClustering,KMeans 9 from mpl_toolkits.mplot3d.axes3d import Axes3D 10 11 # 加载数据 12 X,t = datasets.make_swiss_roll(n_samples=1500,noise = 0.05) 13 # 纵坐标变‘薄’,离的更近 14 X[:,1]*=0.5 15 # (1500, 3) (1500,)三维的 16 display(X.shape,t.shape) 17 18 # 画图 19 fig = plt.figure(figsize=(9,6)) 20 axes3D = Axes3D(fig) 21 axes3D.scatter(X[:,0],X[:,1],X[:,2],c =t) 22 # 调整角度 23 axes3D.view_init(7,-80)
1 # 使用kmeans 2 kmeans = KMeans(6) 3 kmeans.fit(X) 4 y_ = kmeans.predict(X) 5 6 fig = plt.figure(figsize=(9,6)) 7 axes3D = Axes3D(fig) 8 axes3D.scatter(X[:,0],X[:,1],X[:,2],c = y_) 9 axes3D.view_init(7,-80)

kmeans效果不好,和上图比较一下就好
1 from sklearn.neighbors import kneighbors_graph 2 3 # linkage : {"ward", "complete", "average", "single"} 4 conn = kneighbors_graph(X,5) 5 agg = AgglomerativeClustering(n_clusters=6,linkage='ward',connectivity=conn) 6 7 agg.fit(X) 8 9 y_ = agg.labels_ 10 11 fig = plt.figure(figsize=(9,6)) 12 axes3D = Axes3D(fig) 13 14 axes3D.scatter(X[:,0],X[:,1],X[:,2],c = y_) 15 16 axes3D.view_init(7,-80)
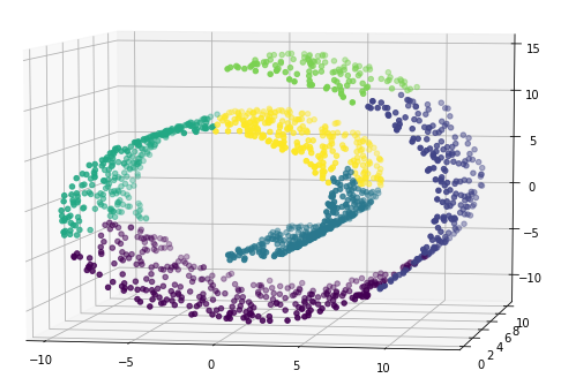
分层聚类,也需要选择合适的参数,距离方法
没有连接性约束的忽视其数据本身的结构,会形成了跨越流形的不同褶皱
添加connectivity便可以得到很好的结果
该图为不加connectivity的结果



 浙公网安备 33010602011771号
浙公网安备 33010602011771号