Python机器学习笔记:线性判别分析(LDA)算法
完整代码及其数据,请移步小编的GitHub
传送门:请点击我
如果点击有误:https://github.com/LeBron-Jian/MachineLearningNote
预备知识
首先学习两个概念:
线性分类:指存在一个线性方程可以把待分类数据分开,或者说用一个超平面能将正负样本区分开,表达式为y=wx,这里先说一下超平面,对于二维的情况,可以理解为一条直线,如一次函数。它的分类算法是基于一个线性的预测函数,决策的边界是平的,比如直线和平面。一般的方法有感知器,最小二乘法。
非线性分类:指不存在一个线性分类方程把数据分开,它的分类界面没有限制,可以是一个曲面,或者是多个超平面的组合。
LDA在模式识别领域(比如人脸识别,舰艇识别等图形图像识别领域)中有非常广泛的应用,因此我们有必要了解一下它的算法原理。不过在学习LDA之前,我们有必要将其与自然语言处理领域中的LDA区分开,在自然语言处理领域,LDA是隐含狄利克雷分布(Latent DIrichlet Allocation,简称LDA),它是一种处理文档的主题模型,我们本文讨论的是线性判别分析,因此后面所说的LDA均为线性判别分析。
那么为什么要用LDA呢?
1,为什么要学习LDA算法?
前面博客学了PCA降维,博客地址如下:
Python机器学习笔记:主成分分析(PCA)算法
PCA是一种无监督的数据降维方法,与之不同的是:LDA是一种有监督的数据降维方法。我们知道即使在训练样本上,我们提供了类别标签,在使用PCA模型的时候,我们是不利于类别标签的,而LDA在进行数据降维的时候是利用数据的类别标签提供的信息的。
从几何的角度来看,PCA和LDA都是将数据投影到新的相互正交的坐标轴上。只不过在投影的过程中他们使用的约束是不同的,也可以说目标是不同的。PCA是将数据投影到方差最大的几个相互正交的方向上,以期待保留最多的样本信息。样本的方差越大表示样本的多样性越好,在训练模型的时候,我们当然希望数据的差别越大越好。否则即使样本很多但是他们彼此相似或者相同,提供的样本信息将相同,相当于只有很少的样本提供信息是有用的。样本信息不足将导致模型性能不够理想。这就是PCA降维的目的:将数据投影到方差最大的几个相互正交的方向上。这种约束有时候很有用,比如在下面这个例子:
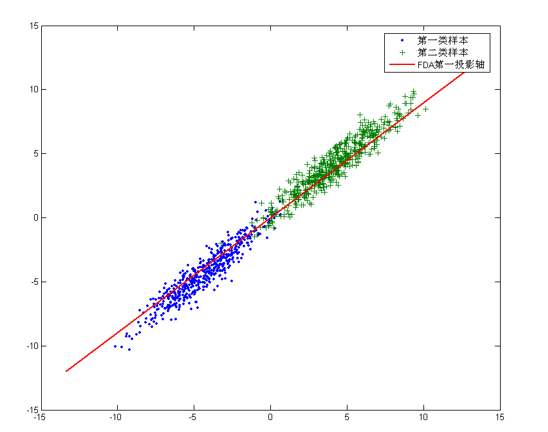
对于这个样本集我们可以将数据投影到 x 轴或者 y 轴,但这都不是最佳的投影方向,因为这两个方向都不能最好的反映数据的分布。很明显还存在最佳的方向可以描述数据的分布趋势,那就是图中红色直线所在的方向。也是数据样本作投影,方差最大的方向。向这个方向做投影,投影后数据的方差最大,数据保留的信息最多。
但是,对于另外的一些不同分布的数据集,PCA的这个投影后方差最大的目标就不太适合了。比如对于下面图片中的数据集:
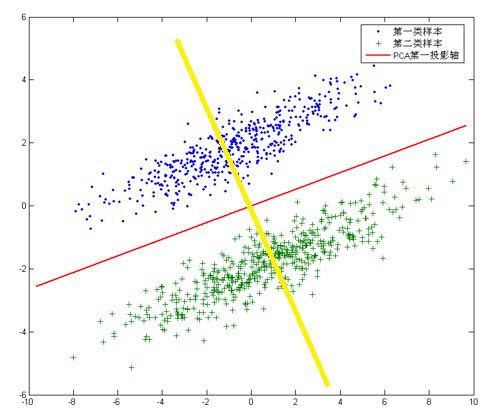
针对这个数据集,如果同样选择使用PCA,选择方差最大的方向作为投影方向,来对数据进行降维。那么PCA选出的最佳投影方向,将是图中红色直线所示的方向。这样做投影确实方差最大,但是是不是有其他问题。聪明的你发现了,这样做投影之后两类数据样本将混合在一起,将不再线性可分,甚至是不可分的。这对我们来说简直是地狱,本来线性可分的样本被我们亲手变得不再可分。而我们发现,如果使用图中黄色直线,向这条直线做投影即能使数据降维,同时还能保证两类数据仍然是线性可分的。上面的这个数据集如果使用LDA降维,找出的投影方向就是黄色直线所在的方向。
这其实就是LDA的思想,或者说LDA降维的目标:将带有标签的数据降维,投影到低维空间同时满足三个条件:
- 1,尽可能多的保留数据样本的信息(即选择最大的特征是对应的特征向量所代表的方向)。
- 2,寻找使样本尽可能好分的最佳投影方向。
- 3,投影后使得同类样本尽可能近,不同类样本尽可能远。


2,LDA的思想
LDA的核心思想:类内小,类间大
线性判别分析(Linear Discriminant Analysis, 以下简称LDA)是一种监督学习的降维技术,也就是说它的数据集的每个样本是有类别输出的。这点和PCA不同,PCA是不考虑样本类别输出的无监督降维技术。LDA的思想可以用一句话概述,就是“投影后类内方差最小,类间方差最大”,什么意思呢?我们要将数据在低维度上进行投影,投影后希望每一种类别数据的投影点尽可能的接近,而不同类别的数据的类别中心之间的距离尽可能的大。
可能这句话有点抽象,那我们先看看最简单的情况。假设我们有两类数据分别是红色和蓝色,如下图所示,这些数据特征是二维的,我们希望将这些数据投影到一维的一条直线,让每一种类别数据的投影点尽可能的接近,而红色和蓝色数据中心之间的距离尽可能的大。
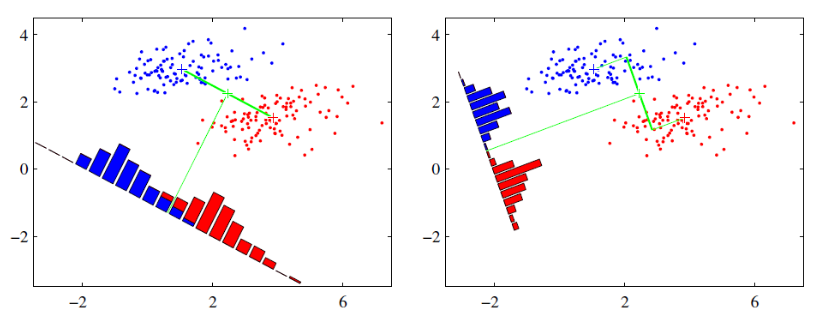
上图中提供了两种投影方式,哪一种能更好的满足我们的标准呢?从直观上看出,右边要比左边图的投影效果好,因为右边图的红色数据进而蓝色数据各个较为集中,且类别之间的距离明显,左边的则在边界处数据混杂。以上就是LDA的主要思想了,当然在实际应用中,我们的数据是多个类别的,我们的原始数据一般也是超过二维的,投影后的也一般不是直线,而是一个低维的超平面。
周志华老师的《机器学习》一书中,已有简述线性判别分析的中心思想,其实这可以联想到方差分析中的组内偏差SSE和组间偏差SSA,毕竟Fisher线性判别分析和方差分析的发明者都是R.A.Fisher。
Fisher判别分析的思想非常朴素:给定训练样本例集,设法将样例投影到一条直线上,使得同类样例的投影点尽可能接近,不同类样例的投影点尽可能远离。在对新样本进行分类时,将其投影到同样的这条直线上,再根据新样本投影点的位置来确定它的类别。
3,LDA算法的优化目标(周志华老师的推导过程)
LDA的原理:投影到维度更低的空间中,使得投影后的点,会形成按类别区分,一簇一簇的情况,相同类别的点,将会在投影后的空间中更接近方法。
下图来自周志华《机器学习》,给出了一个二维示意图:
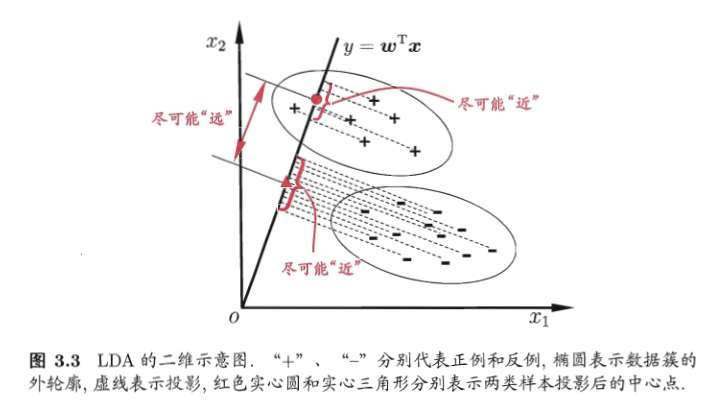
什么是线性判别分析呢?所谓的线性就是,我们要将数据点投影到直线上(可能是多条直线),直线的函数解析式又称为线性函数,通常直线的表达式为:

其实这里的 x 就是样本向量(列向量),如果投影到一条直线上 w 就是一个特征向量(列向量形式)或者多个特征向量构成的矩阵。至于 w 为什么是特征向量,后面我们就能推导出来。 y 为投影后的样本点(列向量)。
我们首先使用两类样本来说明,然后再推广到多类问题。这里简单的学习一下(后面会学习详细推导过程)。
将数据投影到直线 w 上,则两类样本的中心在直线上的投影分别为 WTμ0,WTμ1,若将所有的样本点都投影到直线上,则两类样本的协方差分别为 WT∑0 W 和 WT∑1W。
投影后同类样本协方差矩阵的求法:
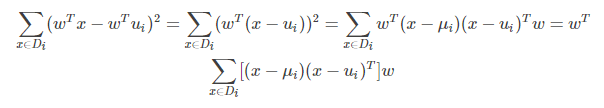
上式的中间部分(即第二行的式子)就是同类样本投影前的协方差矩阵。还可以看出同类样本投影前后协方差矩阵之间的关系。如果投影前的协方差矩阵为 ∑ 则投影后的为 WT∑ W。
上式的推导需要用到如下公式,a,b都是列向量:
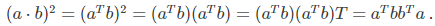
欲使同类样例的投影点尽可能接近,可以让同类样本点的协方差矩阵尽可能小,即下面式子尽可能小:
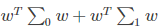
欲使异类样例的投影点尽可能远离,可以让类中心之间的距离尽可能大,即下面式子尽可能大:
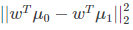
同时考虑二者,则可得到最大化的目标
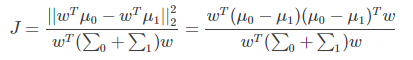
上式 ||*|| 表示欧几里得范数,其中:
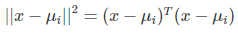
3.1 对上面推导关键点的理解
上面说的同类样本点的协方差矩阵尽可能小,同类样例的投影点就尽可能接近,这句话如何理解呢?
我们在最初接触协方差是用来表示两个变量的相关性,我们来看一下协方差和方差的公式:
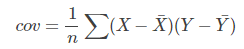
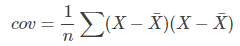
可以看到协方差的公式和方差十分相近,甚至可以说方差是协方差的一种特例。我们知道方差可以用来度量数据的离散程度,(X - Xhat)越大,表示数据距离样本中心越远,数据越离散,数据的方差越大。同样我们观察,协方差的公式,(X - Xhat)和(Y - Yhat)越大,表示数据距离样本中心越远,数据分布越分散,协方差越大。相反他们越小表示数据距离样本中心越近,数据分布越集中,协方差越小。
所以协方差不仅是反映了变量之间的相关性,同样反映了多维样本分布的离散程度(一维样本使用方差),协方差越大(对于负相关来说是绝对值越大),表示数据的分布越分散。所以上面的“欲使同类样例的投影点尽可能接近,可以让同类样本点的协方差矩阵尽可能小”就可以理解了。
要是还不能理解,我们这里再分析一波。
首先LDA分类的一个目标是使得不同类别之间的距离越远越好,同一类别之中的距离越近越好。那么不同类别之间的距离越远越好,我们是可以理解的,就是越远越好区分。
比如每类样例的均值为:
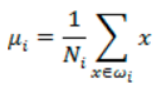
而投影后的均值为:
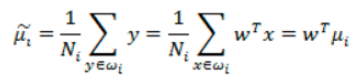
那投影后的两类样本中心点尽量分离:
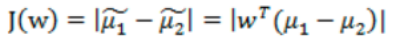
那是不是说我们只最大化J(w) 就可以了?
实际情况当然不是,比如下面情况:

我们发现,对上图来说,假设我们只有两种分类方向,一种是X1方向,一种是 X2方向。
我们看到X1的方向可以最大化J(w)的值,但是却分的不好,μ1 和 μ2 两个数据集之间还有交叉。但是X2方向,虽然不会最大化J(w)的值,但是却分的好。。。。
基于这个问题,我们提出了散列值(也就是样本点的密集程度),当散列值越大,越分散,反之越集中。
我们肯定是希望同类之间应该更密集些,表示如下:
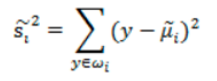
所以,总结来说,就是我们希望不同类样本中心值尽量越分散越好,也就是J(w) 越大越好,而同类之间距离越小越好,也就是散列值之和越小越好。我们最终还是希望J(w)越大越好,所以我们可以将公式写成下面方式,基于此,我们得到目标函数为:
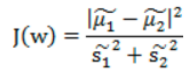
分子展示如下(其中SB称为类间散布矩阵):
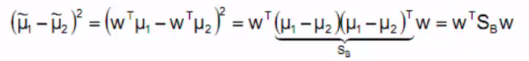
散列值公式展示:

散列矩阵(scatter matrices):

类间散布矩阵:

其中:
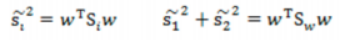
最终目标函数为:
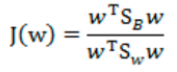
4,瑞利商(Rayleigh quotient)与广义瑞利商(genralized Rayleigh quotient)
我们首先来看看瑞利商的定义。瑞利商是指这样的函数R(A, x):
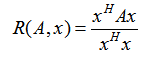
其中 x 为非零向量,而A为 n*n 的Hermitan矩阵。所谓的 Hermitan矩阵就是满足共轭转置矩阵和自己相等的矩阵,即AH=A。如果我们的矩阵A是实矩阵,则满足 AH=A 的矩阵即为Hermitan矩阵。
瑞利商R(A, x) 有一个非常重要的性质,即它的最大值等于矩阵A最大的特征值,而最小值等于矩阵A的最小的特征值,也就是满足:
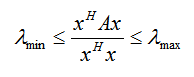
具体的证明这里不给出了,当向量 x 是标准正交基时,即满足xHx = 1时,瑞利商退化为:R(A, x) =xHAx,这个形式在谱聚类和PCA中都有出现。
以上就是瑞利商的内容,现在我们看看广义瑞利商。广义瑞利商是指这样的函数 R(A, B, x):
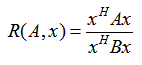
其中 x 为非零向量,而A, B为 n*n 的Hermitan矩阵。B为正定矩阵。它的最大值和最小值是什么呢?其实我们只要通过标准化就可以转化为瑞利商的格式。我们令 x = B-1/2x',则分母转化为:

而分子转化为:

此时我们的 R(A, B, x) 转化为 R(A, B, x'):
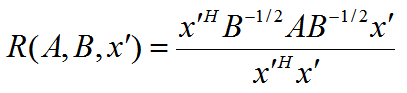
利用前面的瑞利商的性质,我们可以很快的知道,R(A, B, x')的最大值为矩阵 B-1/2AB-1/2的最大特征值,或者说矩阵 B-1A 的最大特征值,而最小值为矩阵 B-1A 的最小特征值(这里使用了一个技巧,即对矩阵进行标准化)。
5,二类LDA原理
现在我们回到LDA的原理上,我们在上面说到了LDA希望投影后希望同一种类别数据的投影点尽可能的接近,而不同类别的数据的类别中心之间的距离尽可能的大,但是这只是一个感官的度量。现在我们首先从比较简单的二类LDA入手,严谨的分析LDA的原理。
LDA的原理:投影到维度更低的空间中,使得投影后的点,会形成按类别区分,一簇一簇的情况,相同类别的点,将会在投影后的空间中更接近方法。

假设我们的数据集 D={(x1, y1), (x2, y2), .... (xm, ym)},其中任意样本 xi 为n维向量,yi属于{0, 1}。我们定义 Nj(j=0, 1)为第 j 类样本的个数,Xj(j = 0, 1)为第 j 类 样本的集合,而 μj(j=0, 1)为第 j 类样本的均值向量,定义 Σj(j=0, 1) 为第 j 类样本的协方差矩阵(严格说是缺少分母部分的协方差矩阵)。
μj 的表达式为:
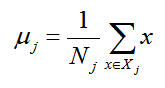
其中 j = 0, 1
Σj的表达式为:
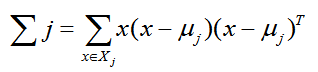
由于是两类数据,因此我们只需要将数据投影到一条直线上即可。假设我们的投影直线是向量 w,则对任意一个样本 xi,它在直线 w 上的投影为 wTxi,对于我们的两个类别的中心点 μ0, μ1,在直线 w 的投影为 wTμ0,wTμ1。由于LDA需要让不同类别的数据的类别中心之间的距离尽可能的大,也就是我们要最大化 ||wTμ0 - wTμ1||22 ,同时我们希望同一种类别数据的投影点尽可能的接近,也就是要同类样本投影点的协方差 wT Σ0 w 和 wT Σ1w 尽可能的小,即最小化 wT Σ0 w + wT Σ1w 。综上所述,我们的优化目标为:

类内散度矩阵
我们一般定义类内散度矩阵 Sw为:
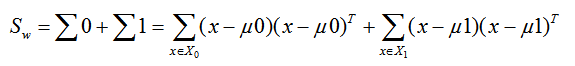
类间散度矩阵
类间散度矩阵其实就是协方差矩阵乘以样本数目,即散度矩阵与协方差矩阵只是相差一个系数。
同时定义类间散度矩阵 Sb为:
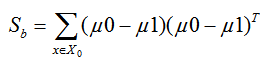
定义过类内散度矩阵和类间散度矩阵后,我们就可以将上述的优化目标重写为:

仔细一看上式,就是我们的广义瑞利商嘛!利用我们提到的广义瑞利商的性质,我们知道我们的 J(w') 最大值为矩阵 Sw-1/2SbSw-1/2 的最大特征值,而对应的w' 为 Sw-1/2SbSw-1/2 的最大特征值对应的特征向量!,而Sw-1/2Sb 的最大特征值对应的特征向量!,而Sw-1Sb 的特征值和 Sw-1/2SbSw-1/2 的特征值相同, Sw-1Sb 的特征向量 w' 满足 w=Sw-1/2w'的关系。
注意到对于二类的时候,Sbw 的方向恒平行于 μ0 - μ1,不妨令 Sbw = λ( μ0 - μ1),将其代入:(Sw-1Sb)w = λ w,可以得到 w = Sw-1( μ0 - μ1),也就是我们只要求出原始二类样本的均值和方差就可以确定最佳的投影方向 w 了。
所以如何确定 w?上面已经简单的说了,这里使用公式更加显而易见吧。
注意上面的分子和分母都是关于 w 的二次项,因此上面的解与 w 的长度无关,只与其方向有关,不失一般性,我们令:

(我们对分母进行归一化:因为如果分子,分母都是可以取任意值的,那就会使得有无穷解,我们将分母限制为长度为1)
则优化目标等价于:


使用拉格朗日乘子法,上式等价于:
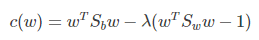
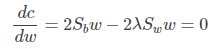
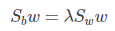
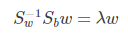
可以看到上式就有转换为一个求解特征值和特征向量的问题了。W就是我们矩阵SW-1SB要求解的特征向量,这就验证了我们之前所说的式子:

中的 w 就是特征向量构成的矩阵。但是在这里我们仍然有个问题要解决,就是Sw是否可逆,遗憾的是在实际的应用中,它通常是不可逆的,我们通常有两个办法解决它。
方法1:令 Sw = Sw + γ I ,其中 γ 是一个特别小的数,这样 Sw一定可逆。
方法2:先用PCA对数据进行降维,使得在降维后的数据上 Sw可逆,再使用LDA。
6,多类 LDA 原理
有了二类LDA的基础,我们再来看看多类别 LDA的原理。
假设我们的数据集 D={(x1, y1), (x2, y2), .... (xm, ym)},其中任意样本 xi 为n维向量,yi属于{C1, C2,... ,Ck}。我们定义 Nj(j=1, 2, ... k)为第 j 类样本的个数,Xj(j = 1, 2, ....k)为第 j 类样本的均值向量,定义 Σj(j = 1, 2, 3, ...k) 为第 j 类样本的协方差矩阵。在二类 LDA 里面定义的公式可以很容易的类推到多类LDA。
由于我们是多类向低维投影,则此时投影到的低维空间就不是一条直线,而是一个超平面了。假设我们投影到的低维空间的维度为 d,对应的基向量为 (w1, w2, ....wd),基向量组成的矩阵为 W,它是一个 n*d 的矩阵。
此时我们的优化目标应该可以变成为:

其中:μ 为所有样本均值向量
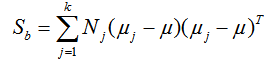
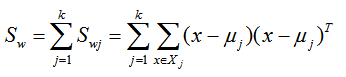
但是有一个问题,就是WTSbW 和 WTSwW 都是矩阵,不是标量,无法作为一个标量函数来优化!也就是说,我们无法直接用二类LDA的优化方法,怎么做呢?一般来说,我们使用其他的一些替代优化目标来实现。
常见的一个 LDA多类优化目标函数定义为:
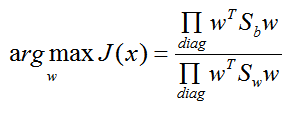
其中 ∏A 为 A的主对角线元素的乘积,W为 n*d 的矩阵。
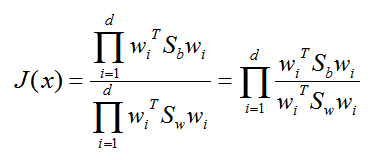
仔细观察上式最右边,这不就是广义瑞利商嘛!最大值是矩阵 Sw-1Sb 的最大特征值,最大的 d 个值的乘积就是矩阵 Sw-1Sb 的最大的 d个特征值的乘积,此时对应的矩阵 W 为这最大的 d 个特征值对应的特征向量张成的矩阵。
由于 W 是一个利用了样本的类别得到的投影矩阵,因此它的降维到的维度 d 最大值为 k-1,为什么最大维度不是类别 k 呢?因为Sb中每个 μj - μ 的秩为1,因此协方差矩阵相加后最大的秩为 k(矩阵的秩小于等于各个相加矩阵的秩的和),但是由于我们知道前 k-1 个 μj 后,最后一个 μk 可以由前 k-1 个 μj 线性表示,因此Sb的秩最大为 k-1,即特征向量最多有 K-1个。
7,LDA 算法流程
下面我们对 LDA 降维的流程做一个总结。
输入:数据集 D = {(x1, y1), (x2, y2), .... (xm, ym)},其中任意样本 xi 为 n维向量, yi € {C1, c2, ...Ck},降维到的维度 d。
输出:降维后的样本集 D'
- 1)计算类内散度矩阵Sw
- 2)计算类间散度矩阵 Sb
- 3)计算矩阵Sw-1Sb
- 4)计算 Sw-1Sb 的最大的 d个特征值和对应的 d个特征向量 (w1, w2, ... wd),得到投影矩阵 W
- 5)对样本集中的每一个样本特征 xi,转化为新的样本 zi = WTxi
- 6)得到输出样本集 D' = {(z1, y1), (z2, y2), .... (zm, ym)}
以上就是使用 LDA 进行降维的算法流程。实际上LDA除了可以用于降维以外,还可以用于分类。一个常见的LDA分类基本思想是假设各个类别的样本数据符合高斯分布,这样利于LDA进行投影后,可以利用极大似然估计计算各个类别投影数据的均值和方差,进而得到该类别高斯分布的概率密度函数。当一个新的样本到来后,我们可以将它投影,然后将投影后的样本特征分别代入各个类别的高斯分布概率密度函数,计算它属于这个类别的概率,最大的概率对应的类别即为预测类别。
8,LDA vs PCA
LDA用于降维,和PCA有很多相同,也有很多不同的地方,因此值得好好地比较一下两者的降维异同点。
首先我们看看相同点:
- 1,两者均可以对数据进行降维
- 2,两者在降维时均使用了矩阵特征分解的思想
- 3,两者都假设数据符合高斯分布
不同点:
- 1,LDA 是由监督的降维方法,而PCA是无监督的降维方法
- 2,LDA降维最多降到类别数 k-1 的维度,而PCA没有这个限制
- 3,LDA除了可以用于降维,还可以用于分类
- 4,LDA选择分类性能最好的投影方向,而PCA选择样本点投影具有最大方差的方向
第四点可以从下图形象的看出,在某些数据分布下LDA比PCA降维较优
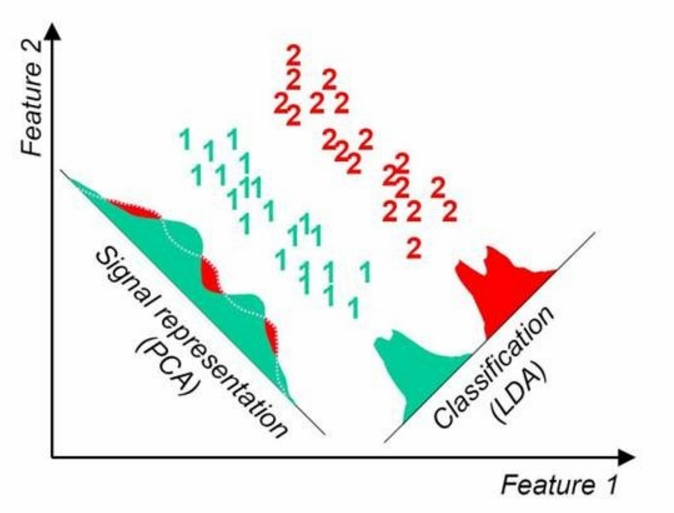
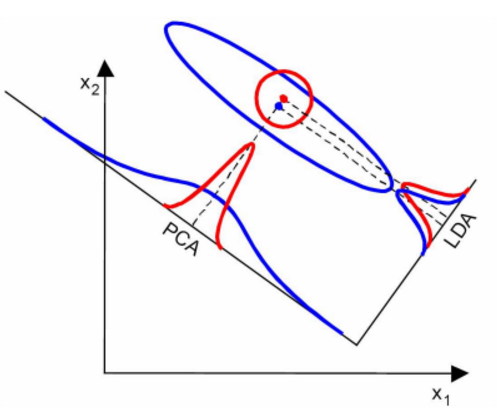
当然,某些数据分布下,PCA 比 LDA 降维较优,如下图所示:
9,LDA算法小结
LDA算法既可以用来降维,也可以用来分类,但是目前来说,主要还是用于降维。在我们进行图像识别相关的数据分析时,LDA是一个有力的工具,下面总结LDA算法的优缺点
LDA算法的主要优点:
- 1,在降维过程中可以使用类别的先验知识经验,而像PCA这样的无监督学习则无法使用类别先验知识
- 2,LDA在样本分类信息依赖均值而不是方差的时候,比PCA之类的算法较优
LDA算法的主要缺点:
- 1,LDA不适合对非高斯分布样本进行降维,PCA也有这个问题
- 2,LDA降维最多降到类别数 k-1 的维数,如果我们降维的维度大于 k-1,则不能使用 LDA。当然目前有一些LDA的进化版算法可以绕过这个问题
- 3,LDA在样本分类信息依赖方差而不是均值的时候,降维效果不好
- 4,LDA可能过度拟合数据,
用Sklearn 进行LDA降维
在scikit-learn中, LDA类是sklearn.discriminant_analysis.LinearDiscriminantAnalysis。那既可以用于分类又可以用于降维。当然,应用场景最多的还是降维。和PCA类似,LDA降维基本也不用调参,只需要指定降维到的维数即可。
1,LinearDiscriminantAnalysis 类概述
我们这里对LinearDiscriminantAnalysis类的参数做一个基本的总结。
1)solver : 即求LDA超平面特征矩阵使用的方法。可以选择的方法有奇异值分解"svd",最小二乘"lsqr"和特征分解"eigen"。一般来说特征数非常多的时候推荐使用svd,而特征数不多的时候推荐使用eigen。主要注意的是,如果使用svd,则不能指定正则化参数shrinkage进行正则化。默认值是svd
2)shrinkage:正则化参数,可以增强LDA分类的泛化能力。如果仅仅只是为了降维,则一般可以忽略这个参数。默认是None,即不进行正则化。可以选择"auto",让算法自己决定是否正则化。当然我们也可以选择不同的[0,1]之间的值进行交叉验证调参。注意shrinkage只在solver为最小二乘"lsqr"和特征分解"eigen"时有效。
3)priors :类别权重,可以在做分类模型时指定不同类别的权重,进而影响分类模型建立。降维时一般不需要关注这个参数。
4)n_components:即我们进行LDA降维时降到的维数。在降维时需要输入这个参数。注意只能为[1,类别数-1)范围之间的整数。如果我们不是用于降维,则这个值可以用默认的None。
从上面的描述可以看出,如果我们只是为了降维,则只需要输入n_components,注意这个值必须小于“类别数-1”。PCA没有这个限制。
2,LinearDiscriminantAnalysis 降维实例
我们知道,PCA和LDA都可以用于降维,两者没有绝对的优劣之分,使用两者的原则实际取决于数据的分布。由于LDA可以利用类别信息,因此某些时候比完全无监督的PCA会更好,下面我们举一个LDA降维可能更优的例子。
首先,生成三类三维特征的数据,代码如下:
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.datasets.samples_generator import make_classification
X, y = make_classification(n_samples=1000, n_features=3, n_redundant=0,
n_classes=3, n_informative=2, n_clusters_per_class=1,
class_sep=0.5, random_state=10)
fig = plt.figure()
ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=20, azim=20)
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker='o', c=y)
我们看看最初的三维数据的分布情况:

首先我们看看使用PCA降维到二维的情况,注意PCA无法使用类别信息来降维,代码如下:
pca = PCA(n_components=2) pca.fit(X) print(pca.explained_variance_ratio_) print(pca.explained_variance_) # [0.43377069 0.3716351 ] # [1.21083449 1.0373882 ] X_new = pca.transform(X) plt.scatter(X_new[:, 0], X_new[:, 1], marker='o', c=y) plt.show()
图如下:
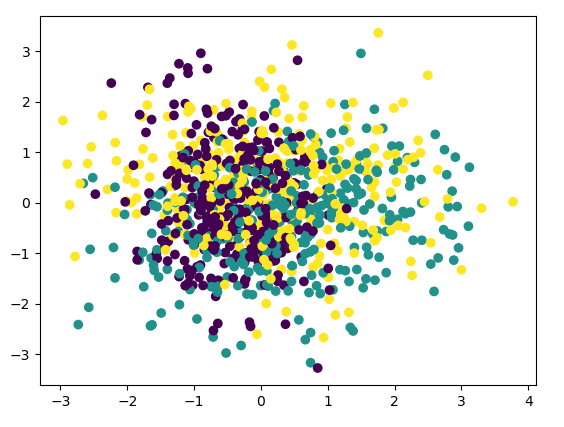
由于PCA没有利用类别信息,我们可以看到降维后,样本特征和类别的信息关联几乎完全丢失。
下面看看LDA的效果,代码如下:
lda = LinearDiscriminantAnalysis() lda.fit(X, y) X_new = lda.transform(X) plt.scatter(X_new[:, 0], X_new[:, 1], marker='o', c=y) plt.show()
输出的效果图如下:
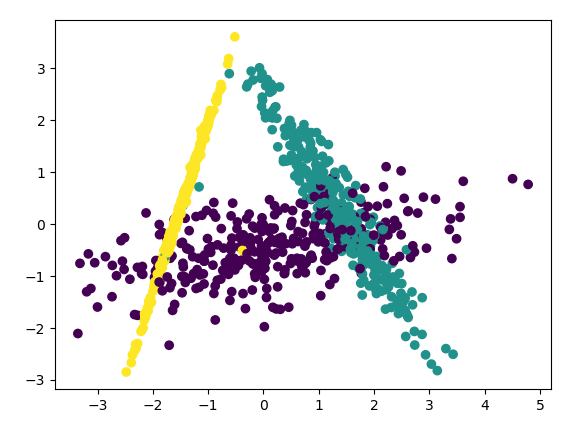
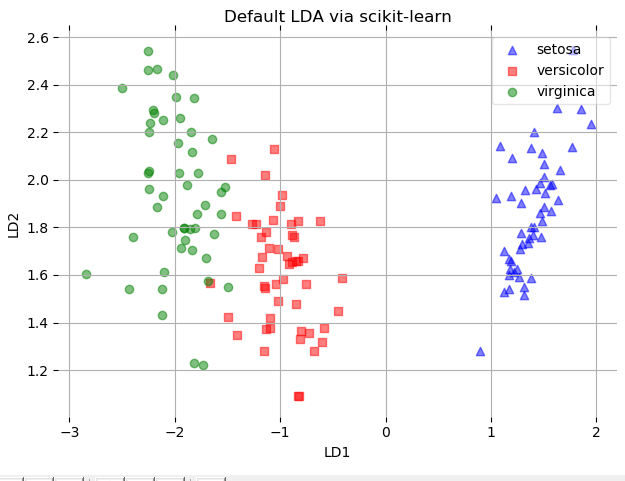
可以看出降维后样本特征信息之间的关系得以保留。
一般来说,如果我们的数据是由类别标签的,那么优先选择LDA去尝试降维;当然也可以使用PCA做很小幅度的降维去消去噪音,然后再使用LDA降维。如果没有类别标签,那么肯定PCA是最先考虑的一个选择了。
3,LDA实现iris 数据
首先,iris数据集可以去我的GitHub下载(地址:https://github.com/LeBron-Jian/MachineLearningNote)
下面我们分析导入的数据集,代码如下:
import pandas as pd
feature_dict = {i: label for i, label in zip(range(4),
("Sepal.Length",
"Sepal.Width",
"Petal.Length",
"Petal.Width",))}
# print(feature_dict) # {0: 'Sepal.Length', 1: 'Sepal.Width', 2: 'Petal.Length', 3: 'Petal.Width'}
df = pd.read_csv('iris.csv', header=None, sep=',')
df.columns = ["Number"] + [l for i, l in sorted(feature_dict.items())] + ['Species']
# to drop the empty line at file-end
df.dropna(how='all', inplace=True)
# print(df.tail()) # 打印数据的后五个,和 .head() 是对应的
后五个数据如下:

下面我们把数据分成data和label,如下形式:

代码如下:
X = df[["Sepal.Length", "Sepal.Width", "Petal.Length", "Petal.Width"]].values y = df['Species'].values enc = LabelEncoder() label_encoder = enc.fit(y) y = label_encoder.transform(y) + 1
这样我们对label进行分类,其中{1: 'Setosa', 2: 'Versicolor', 3: 'Virginica'},分类大概是这样的:
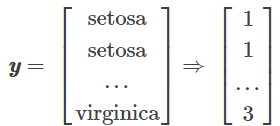
下面我们分别求三种鸢尾花数据在不同特征维度上的均值向量 Mi。

求均值的代码如下:
np.set_printoptions(precision=4)
mean_vectors = []
for c1 in range(1, 4):
mean_vectors.append(np.mean(X[y == c1], axis=0))
print('Mean Vector class %s : %s\n' % (c1, mean_vectors[c1 - 1]))
三个类别,求出的均值如下:
Mean Vector class 1 : [5.006 3.428 1.462 0.246] Mean Vector class 2 : [5.936 2.77 4.26 1.326] Mean Vector class 3 : [6.588 2.974 5.552 2.026]
下面计算两个 4*4 维矩阵:类内散布矩阵和类间散布矩阵,公式如下:
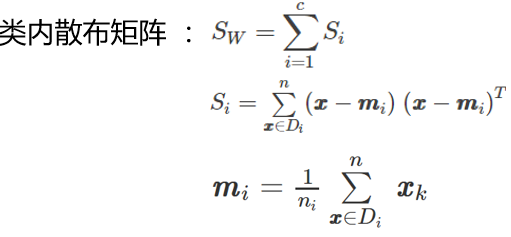
代码如下:
S_W = np.zeros((4, 4))
for c1, mv in zip(range(1, 4), mean_vectors):
# scatter matrix for every class
class_sc_mat = np.zeros((4, 4))
for row in X[y == c1]:
# make column vectors
row, mv = row.reshape(4, 1), mv.reshape(4, 1)
class_sc_mat += (row - mv).dot((row - mv).T)
# sum class scatter metrices
S_W += class_sc_mat
print('within-class Scatter Matrix:\n', S_W)
结果如下:
within-class Scatter Matrix: [[38.9562 13.63 24.6246 5.645 ] [13.63 16.962 8.1208 4.8084] [24.6246 8.1208 27.2226 6.2718] [ 5.645 4.8084 6.2718 6.1566]]
类间散布矩阵如下:
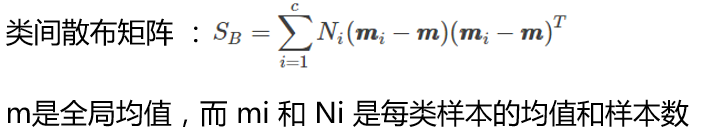
代码如下:
overall_mean = np.mean(X, axis=0)
S_B = np.zeros((4, 4))
for i, mean_vec in enumerate(mean_vectors):
n = X[y == i + 1, :].shape[0]
# make column vector
mean_vec = mean_vec.reshape(4, 1)
# make column vector
overall_mean = overall_mean.reshape(4, 1)
S_B += n*(mean_vec - overall_mean).dot((mean_vec - overall_mean).T)
print('between-class Scatter matrix:\n', S_B)
结果展示:
between-class Scatter matrix: [[ 63.2121 -19.9527 165.2484 71.2793] [-19.9527 11.3449 -57.2396 -22.9327] [165.2484 -57.2396 437.1028 186.774 ] [ 71.2793 -22.9327 186.774 80.4133]]
然后我们求解矩阵的特征值:

代码如下:
eig_vals, eig_vecs = np.linalg.eig(np.linalg.inv(S_W).dot(S_B))
for i in range(len(eig_vals)):
eigvec_sc = eig_vecs[:, i].reshape(4, 1)
print('\n Eigenvector {}: \n {}'.format(i+1, eigvec_sc.real))
print('Eigenvalue {: }: {:.2e}'.format(i+1, eig_vals[i].real))
结果如下:
Eigenvector 1: [[ 0.2087] [ 0.3862] [-0.554 ] [-0.7074]] Eigenvalue 1: 3.22e+01 Eigenvector 2: [[-0.0065] [-0.5866] [ 0.2526] [-0.7695]] Eigenvalue 2: 2.85e-01 Eigenvector 3: [[-0.868 ] [ 0.3515] [ 0.3431] [ 0.0729]] Eigenvalue 3: -1.04e-15 Eigenvector 4: [[-0.1504] [-0.2822] [-0.3554] [ 0.8783]] Eigenvalue 4: 1.16e-14
最后求特征值与特征向量,其中:
- 特征向量:表示映射方向
- 特征值:特征向量的重要程度
代码如下:
# make a list of (eigenvalue, eigenvector) tuples
eig_pairs = [(np.abs(eig_vals[i]), eig_vecs[:, i]) for i in range(len(eig_vals))]
# sort the (eigenvalue, eigenvector) tuples from high to low
eig_pairs = sorted(eig_pairs, key=lambda k: k[0], reverse=True)
# Visually cinfirm that the list is correctly sorted by decreasing eigenvalues
print('Eigenvalues in decreasing order: \n')
for i in eig_pairs:
print(i[0])
特征向量如下:
Eigenvalues in decreasing order: 32.19192919827802 0.28539104262306847 1.1615338676864736e-14 1.039562883345519e-15
特征值代码如下:
print('Variance explained:\n')
eigv_sum = sum(eig_vals)
for i, j in enumerate(eig_pairs):
print('eigenvalue {0:}: {1:.2%}'.format(i + 1, (j[0] / eigv_sum).real))
结果如下:
Variance explained: eigenvalue 1: 99.12% eigenvalue 2: 0.88% eigenvalue 3: 0.00% eigenvalue 4: 0.00%
我们从上可以知道,选择前两维特征
W = np.hstack((eig_pairs[0][1].reshape(4, 1), eig_pairs[1][1].reshape(4, 1)))
print('Matrix W: \n', W.real)
特征矩阵W如下
Matrix W: [[ 0.2087 -0.0065] [ 0.3862 -0.5866] [-0.554 0.2526] [-0.7074 -0.7695]]
lda如下:
X_lda = X.dot(W) assert X_lda.shape == (150, 2), 'The matrix is not 150*2 dimensional.'
下面画图:
def plt_step_lda():
ax = plt.subplot(111)
for label, marker, color in zip(range(1, 4), ('^', 's', 'o'), ('blue', 'red', 'green')):
plt.scatter(x=X_lda[:, 0].real[y == label],
y=X_lda[:, 1].real[y == label],
marker=marker,
color=color,
alpha=0.5,
label=label_dict[label])
plt.xlabel('LD1')
plt.ylabel('LD2')
leg = plt.legend(loc='upper right', fancybox=True)
leg.get_frame().set_alpha(0.5)
plt.title('LDA: Iris projection onto the first 2 linear discriminants')
# hide axis ticks
plt.tick_params(axis='both', which='both', bottom='off',
top='off', labelbottom='on', left='off',
labelleft='on')
# remove axis spines
ax.spines['top'].set_visible(False)
ax.spines['right'].set_visible(False)
ax.spines['bottom'].set_visible(False)
ax.spines['left'].set_visible(False)
plt.grid()
plt.tight_layout()
plt.show()
plt_step_lda()
如图所示:

使用sklearn实现lda:
# LDA
sklearn_lda = LDA(n_components=2)
X_lda_sklearn = sklearn_lda.fit_transform(X, y)
def plot_scikit_lda(X, title):
ax = plt.subplot(111)
for label, marker, color in zip(range(1, 4), ('^', 's', 'o'), ('blue', 'red', 'green')):
plt.scatter(x=X_lda[:, 0].real[y == label],
# flip the figure
y=X_lda[:, 1].real[y == label] * -1,
marker=marker,
color=color,
alpha=0.5,
label=label_dict[label])
plt.xlabel('LD1')
plt.ylabel('LD2')
leg = plt.legend(loc='upper right', fancybox=True)
leg.get_frame().set_alpha(0.5)
plt.title(title)
# hide axis ticks
plt.tick_params(axis='both', which='both', bottom='off',
top='off', labelbottom='on', left='off',
labelleft='on')
# remove axis spines
ax.spines['top'].set_visible(False)
ax.spines['right'].set_visible(False)
ax.spines['bottom'].set_visible(False)
ax.spines['left'].set_visible(False)
plt.grid()
plt.tight_layout()
plt.show()
plot_scikit_lda(X, title='Default LDA via scikit-learn')
结果如下:
PS:这里是整理的学习笔记,参考文献如下,还有自己老师的PPT,不喜勿喷,谢谢
参考文献:http://www.cnblogs.com/LeftNotEasy/archive/2011/01/08/lda-and-pca-machine-learning.html
https://www.cnblogs.com/pinard/p/6244265.html
https://www.cnblogs.com/pinard/p/6249328.html
https://blog.csdn.net/liuweiyuxiang/article/details/78874106



 浙公网安备 33010602011771号
浙公网安备 33010602011771号