【种群历史动态】评估方法SFS(Site/Allele Frequency Spectrum)
SFS解释原文:joint SFS – The G-cat
参考博客:种群动态历史评估方法之 SFS(Site/Allele Frequency Spectrum) - 简书 (jianshu.com)
基因组数据统计可以总结数据集中等位基因的变异或分布,而无需我们所有样品的整个基因序列。我们可以选择一个非常有效的汇总统计量SFS(等位基因频谱),这与等位基因频率测量(如Fst)混淆,SFS本质上是某些等位基因在我们的数据集中的频率的直方图。SFS根据每个等位基因的常见程度 将其分类为待定类别,并计算以该频率出现的等位基因的数量。类别的总数将是可能等位基因的最大数量:对于二倍体物种,每条染色体具有两个拷贝,这意味着包含的样本数量是其两倍。
SFS只是我们可以用来调查种群历史的众多工具之一。通过结合基因组技术,聚结理论和更强大的分析方法,SFS似乎准备解决地球上生命进化史的更微妙和更复杂的问题。
- SFS的应用
我们可以对种群历史进行推断,而无需模拟大型和复杂的基因序列,而是使用SFS。将我们观察到的SFS与瓶颈的模拟场景进行比较,并比较预期的SFS,使我们能够估计该场景的可能性。
例如,我们可以预测,在种群中最近出现遗传瓶颈的情况下,种群中罕见的等位基因将由于遗传漂移而不成比例地丢失。因此,SFS的整体形状将急剧向右移动,留下瓶颈的明确遗传信号。这与瓶颈的合并测试具有相同的理论背景。
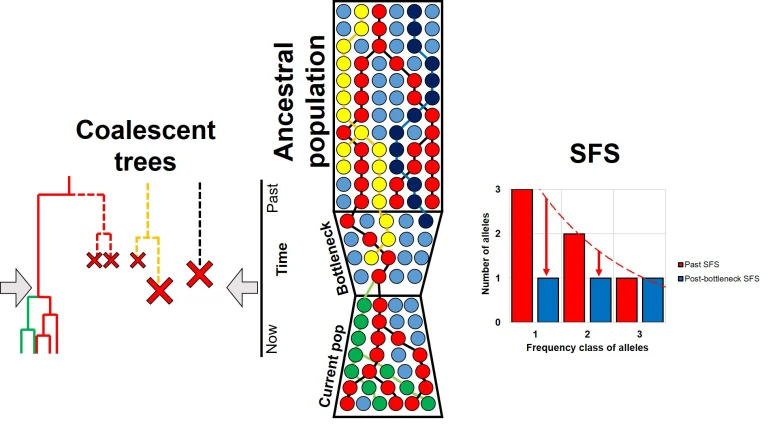
瓶颈如何导致 SFS 偏移的一个代表性示例。中间图:随时间变化的等位基因图,在瓶颈期间丢失了较稀有的变异(黄色和深蓝色),和但更常见的变异保留下来(红色)。左图:这种趋势反映在这些等位基因的合并树上,红色十字表示该等位基因完全丧失。右图:所描绘等位基因的瓶颈事件之前(红色)和之后(蓝色)的SFS。在瓶颈之前,变异以通常的指数形式分布:然而,之后,稀有变体的不成比例的损失会导致分布趋于平缓。通常,SFS将由比这里显示的更多的等位基因构建而成,并且延伸得更远。
相比之下,一个大型或不断种群将具有更多来自突然生长和遗传变异增加的稀有等位基因。因此,与瓶颈相反,SFS分布将偏向频谱的左端,并具有过多的低频变异。

与上图类似,但这次是扩展事件而不是瓶颈。种群的扩张以及随后的Ne的增加,促进了遗传漂移中新等位基因的突变(或减少漂移中等位基因的损失),导致更多新的稀有等位基因出现。左图合并树,右图SFS的偏移。
SFS甚至可用于检测自然选择下的等位基因。对于基因组的强选择部分,等位基因应以高(如果选择为正)或低(如果选择为负)的频率发生,并具有更多中间频率的。
- 评估SFS (estimate Site Allele Frequency)
使用ABGSD软件进行SFS Estimation,missing data和低depth位点。以bam文件为input。
软件下载目录:GitHub - ANGSD/angsd: Program for analysing NGS data.
软件教程:SFS Estimation - angsd (popgen.dk)
#first generate .saf file
./angsd -bam bam.filelist -doSaf 1 -out smallFolded -anc chimpHg19.fa -GL 2
#now try the EM optimization with 4 threads
misc/realSFS smallFolded.saf.idx -maxIter 100 -P 4 >smallFolded.sfs
#in R
sfs<-scan("smallFolded.sfs")
barplot(sfs[-1])
针对vcf中miss不多情况,可以直接用easySFS软件
软件下载:https://github.com/isaacovercast/easySFS
python3 /home/software/easySFS/easySFS.py -i vcf -p ./pops_file.txt -a -f --proj 20,16
第一个群体10个体,第二个群体8个体
最后得到的结果格式.obs
![]()
3.Fastsimcoal2,SFS建模模拟种群动态历史
fastsimcoal安装和使用教程:Demographic modeling with fastsimcoal2 | Speciation & Population Genomics: a how-to-guide (speciationgenomics.github.io)
使用Fastsimcoal2.6,利用SFS模拟种群动态历史需要三个输入文件:

- .tpl 模版文件(参数文件):用来指定历史事件、migration、样本大小、突变率重组率等等。注意,这里的突变率重组率都为【per generation】
- .est 参数评估文件(prior文件):在这里对参数施加prior,指定参数之间关系等等。
- .obs SFS观测文件:就是上一步生成的。
这三个文件的前缀必须相同,在这里为"6.GL1.folded.HN"。如果为unfolded SFS,SFS观测文件后缀应该是DAF之类的而不是MAF,具体得看说明书。
一般来说,有两种构建模型的方法:
1)可以构建很多模型(可能3~10个)来对种群历史建模,然后比较每个模型的lnL(也就是LRT检验),看那个模型最合适。比如第一个模型是有效群体大小先上升后下降在上升,第二个是先下降在上升等等。对于单个群体(不考虑migration)的简单模型,一般也就是设置几个bottleneck时间点,扩张时间点以及对应的种群大小参数。
2)只构建一个模型,限制“时间点”prior的范围,但是放松Ne prior的范围,并保持一致,让算法自己探索种群在某个时间点是扩张还是收缩。在RULES中只对时间做限制,而不对Ne做限制。这种就需要增加运行的次数,让模型达到收敛,比如设置-L 100。例如:
.est文件:
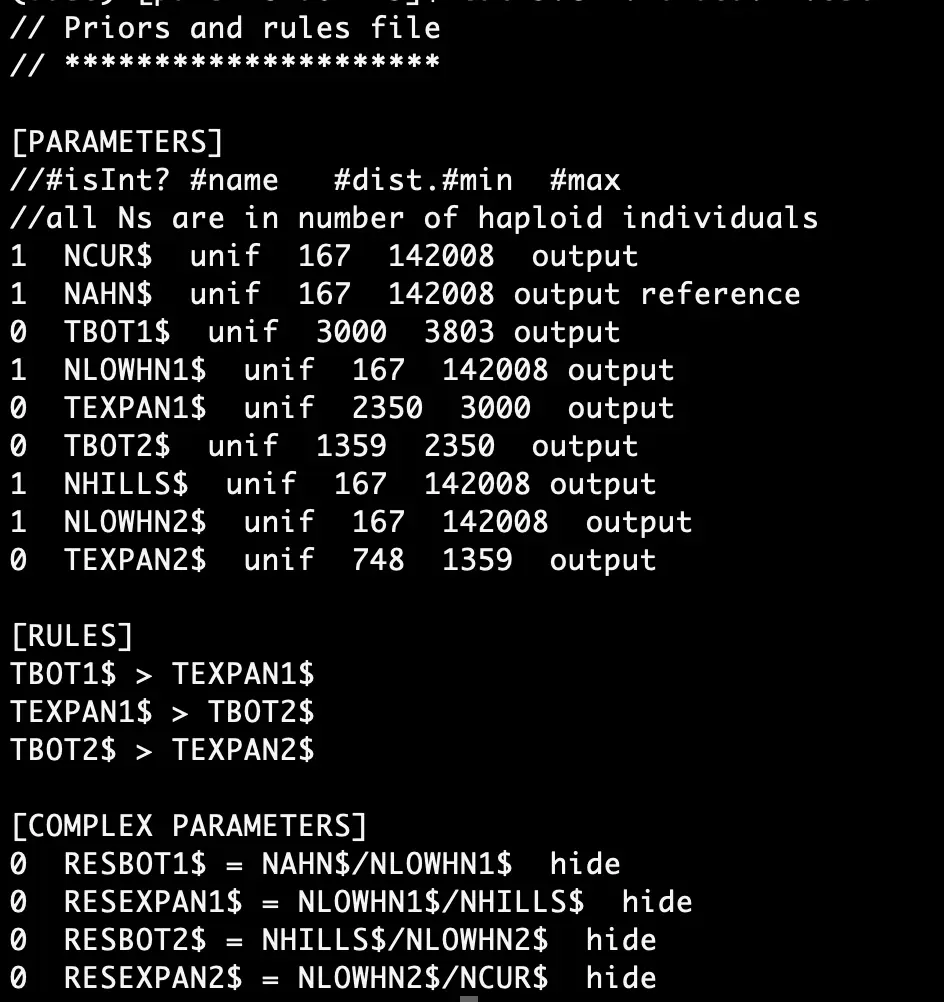
.tpl文件:

设置好配置文件以后,开跑:
# 对于folded SFS,记得设置 -m
fsc26 -t ${i}.GL1.folded.HN.tpl -e ${i}.GL1.folded.HN.est -m -0 -C 10 -n 100000 -L 40 -s 0 -M -q -c 5 &
网友评价:生成文件里有likelihood的评估,拿过来比较模型就行了。对最优模型可以再跑bootstrap。
我个人觉得对于单个种群而言,用SFS不一定是好的选择。PSMC从原理上更加靠谱一些。而fastsimcoal模型的优势在于其可以考虑多个种群的migration、融合与分裂、群体大小变化等等。
Stairway plot v2
分析方法:https://raw.githubusercontent.com/xiaoming-liu/stairway-plot-v2/master/READMEv2.1.pdf
后继如果看到SFS相关文献和分析方法,会续上!


 浙公网安备 33010602011771号
浙公网安备 33010602011771号