安装化学信息包RDKit以及ChemoPy(pychem),入了很多坑,发现自己对Python的掌握尚有不足,但最终还是成功将RDKit包以及ChemoPy包成功安装,并能够使用。记下自己最终实现两个包的安装过程,一方面希望记录下以防忘记,一方面希望对你们有所帮助:
一、RDKit包安装
1.安装RDKit包需下载安装Anaconda(下载网址:https://www.anaconda.com/download/),具体安装方法上网可查。
2.安装成功后,在开始菜单可打开Anaconda Prompt命令窗口。
3.输入命令 conda create -c rdkit -n my-rdkit-env rdkit即可等待安装。若安装失败,可采用以下两种方式解决:
1)使用命令conda install -c conda-forge rdkit进行安装
2)失败原因存在从官网中下载包慢导致安装失败,因此课采用国内清华镜像网站进行安装:
1 conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/msys2/ 2 conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/ 3 conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/ 4 conda config --set show_channel_urls yes 5 注:conda config --add channels 为添加镜像命令
4.包安装成功后使用Jupyter测试包是否成功时,会发现有以下警告,则原因是未激活创建的虚拟环境my-rdkit-env,因此需使用conda activate my-rdkit-env进行激活,使用conda deactivate进行取消。
激活成功与取消激活
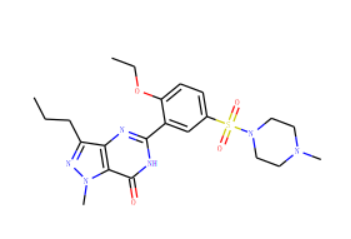
5.测试代码
1 from rdkit import Chem 2 from rdkit.Chem import Draw 3 smi = 'CCCc1nn(C)c2C(=O)NC(=Nc12)c3cc(ccc3OCC)S(=O)(=O)N4CCN(C)CC4' 4 m = Chem.MolFromSmiles(smi) 5 Draw.MolToImageFile(m,"mol.png")
6.测试结果
二、ChemoPy(pychem)包安装
1.ChemoPy包需从该网址http://code.google.com/p/pychem/downloads/list上下载,因为是外网,需使用插件才能够进行访问。
2.因ChemoPy包使用python2开发,应用于python3中需要将文件中的语法进行修改,主要包括引用路径的修改与输出print的修改。
3.将修改后的包放入Anaconda安装目录的pkgs文件夹下
4.使用命令行进入pkgs文件夹中的chemopy包中,使用python setup.py install 命令即可安装
5.使用代码进行测试是否成功
测试代码
1 from pychem.pychem import Chem 2 from pychem import topology 3 mol = Chem.MolFromMolFile("./1.mol") 4 res = topology.CalculateBalaban(mol)
(注:若出现openbabel包未安装,则使用命令conda install -c openbabel openbabel 即可安装)
三、总结
对两个包的安装,只有遇到问题并想办法解决问题,最终才能够实现想要达到的效果。记下这方面的内容,希望未来学习这方面内容的你们不要像我一样头疼焦急无法安装成功,导致后续工作无法进行,希望该文章能给及你们一些帮助。



