基于attractor landscape研究疾病发展及药物研发
本篇博客主要是关于用attractor landscape对疾病分析及其逆向应用——药物研发的一个总结,主要基于5篇论文,3篇KAIST的2015-2017的分析结直肠癌的论文,一篇Regan的应用型论文及一篇有关attractor landscape用于药物发现的2019年的综述。
KAIST 2015 Precritical State Transition Dynamics in the Attractor Landscape of a Molecular Interaction Network Underlying Colorectal Tumorigenesis
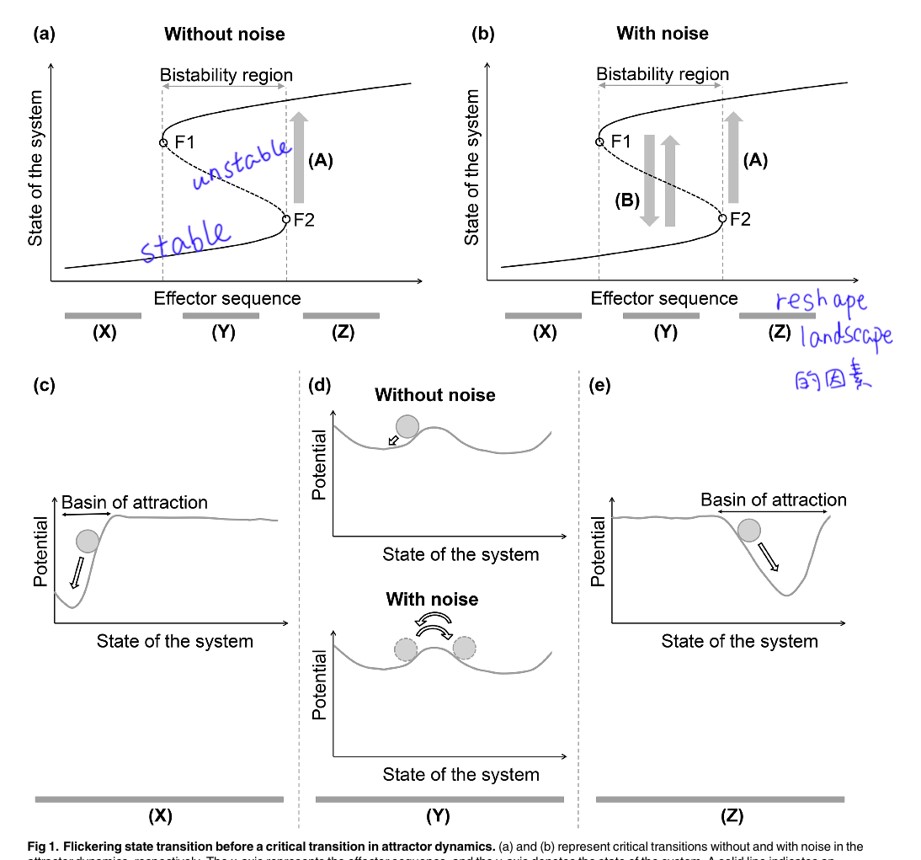
这篇文章主要应用了一个蛋白质分子网络(一个随机布尔网络),构造attractor landscape来分析直肠癌。主要的供献是为以下argument提供了证据:“Scheffer等人提出在广泛的自然系统中,在临界转换之前都会有早期警告信号。基于吸引子动态性,在随机系统中,抖动到其他的状态可能是一个临界转换之前的早期警告信号。”
实际上,肿瘤可以被认为是在增殖和凋亡吸引子之间的一种临界转换。作者在本文中证实在直肠癌中存在抖动状态转换。下面就是抖动的说明(b, d):
本文用到的随机布尔网络是Fumia在2013年提出的模型,包括96个节点和246条边。96个节点中,91个是和直肠癌相关的蛋白质,5个是环境刺激,包括致癌物,生长因子,低氧等。
文中提出了衡量attractor basin大小的方法及衡量有多少状态在噪声下在增殖(P),凋亡(A)和静止(Q)状态中来回切换(抖动)MF,MF=sqrt((BAN-BA0)2+(BPN-BP0)2+(BQN-BQ0)2),BX0 代表没有噪声的吸引子大小,BXN代表有噪声下的吸引子大小。
下图是实验结果及已定义好的指标分析:
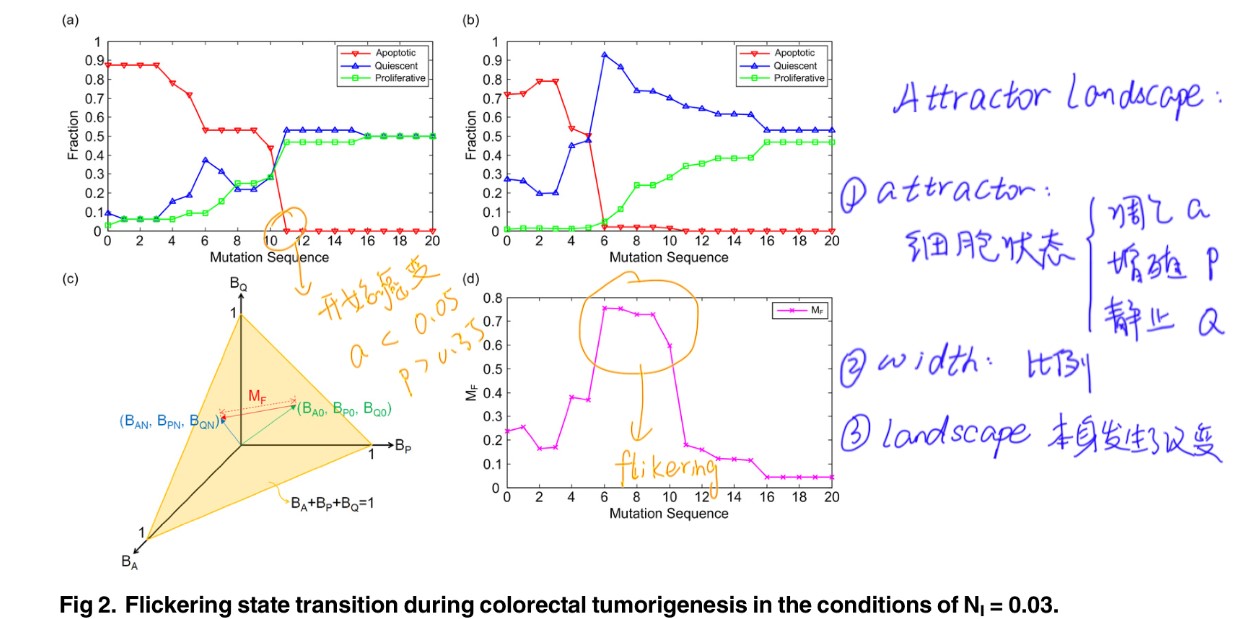
该图是在特定基因突变下和加噪声的结果,(a)中我们可以看到在第11个基因发生突变之后,开始癌变,因为凋亡吸引子没有了,都变成了增殖和静止状态。(a)和(b)分别是在噪声为0和0.03的结果,我们可以看到噪声可以加速癌变,在累积基因突变6个就开始发生癌变,图(d)是和(b)对应的抖动分析,用了我们之前定义的MF指标,我们可以看到在癌变前发生了抖动。上图右边标注的是有关landscape本身属性的一些分析。
这是这篇文章主要的分析,后面还有一些更一般性的假设检验,我这里就不说明了,这些数据就为前面的argument提供了证据。
KAIST 2016 Attractor landscape analysis of colorectal tumorigenesis and its reversion
本篇论文主要基于人类信号通路网络,用attractor landscape来分析结直肠肿瘤及其逆转(cancer reversion)。
肿瘤和信号通路有紧密的联系(信号通路是指当细胞里要发生某种反应时,信号从细胞外到细胞内传递了一种信息,细胞要根据这种信息来做出反应的现象),通常基因突变会影响通路,进而引发肿瘤。① 本文基于信号转导的实验数据及已有的模型构建了一个布尔网络模型(topology:大规模人类信号网络+dynamics:布尔逻辑),每个节点的状态值代表对应信号蛋白质的活动(1: active 0: inactive)。② 接着,他们用attractor landscape来分析结直肠肿瘤,验证了该肿瘤是串行基因突变的累积结果(从APC, KRAS, PTEN到TP53串行突变)。③ 最后,识别该网络中最小控制节点逆向转化癌细胞到正常增殖的细胞或者静止的细胞。
① 布尔网络构建
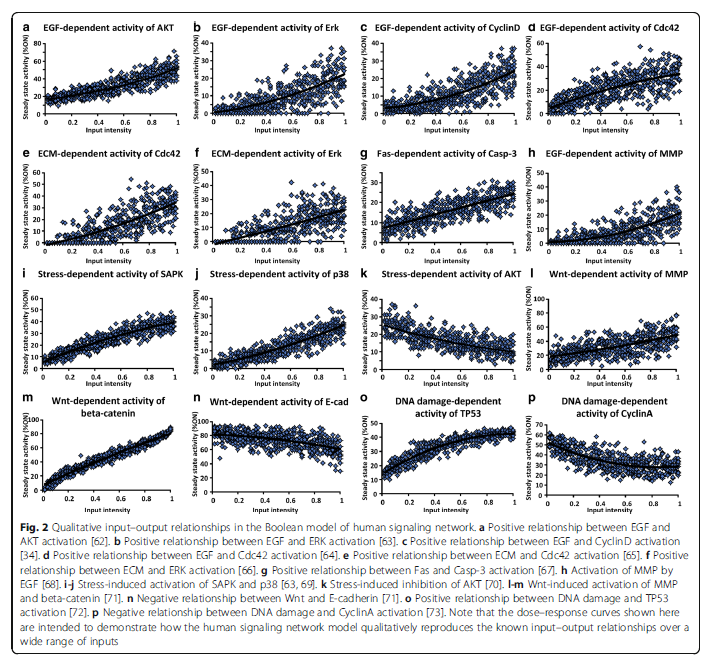
拓扑结构上,该网络是在已有的网络上进行补充(貌似是他们之前提出的一个网络),最终构建出的网络是197个节点和688有向边(其中,有13个外部输入nodes)。网络动态性方面(布尔逻辑),基于之前的模型及手工查文献来构建。为了验证该布尔网络的可靠性,他们进行了输入输出关系的分析,发现网络表现与实际是一致的,这说明构建出来的网络是可靠的,相关图如下:
② attractor landscape分析结直肠肿瘤
对于给定的布尔网络,给定一个输入条件,获得其attractor landscape:随机采样10,000个初始状态,直到一个初始状态的状态轨迹到达一个点attractor(固定状态)或者一个周期的attractor(周期性重复状态)(这里我的理解就是前者最终只到达了一个状态值,如0,没有出边;后者可能是010101这样,可以看作一个大attractor)。
构造出的landscape上,基本的细胞表型有凋亡,转移,增殖和静止,对应就是4个attractor。然后基于已有的生物分析肿瘤演化的知识,我们在attractor landscape上用节点扰动来模拟基因突变,先是删除APC,接着激活KRAS,删除PTEN,最后删除TP53,实验结果如下:
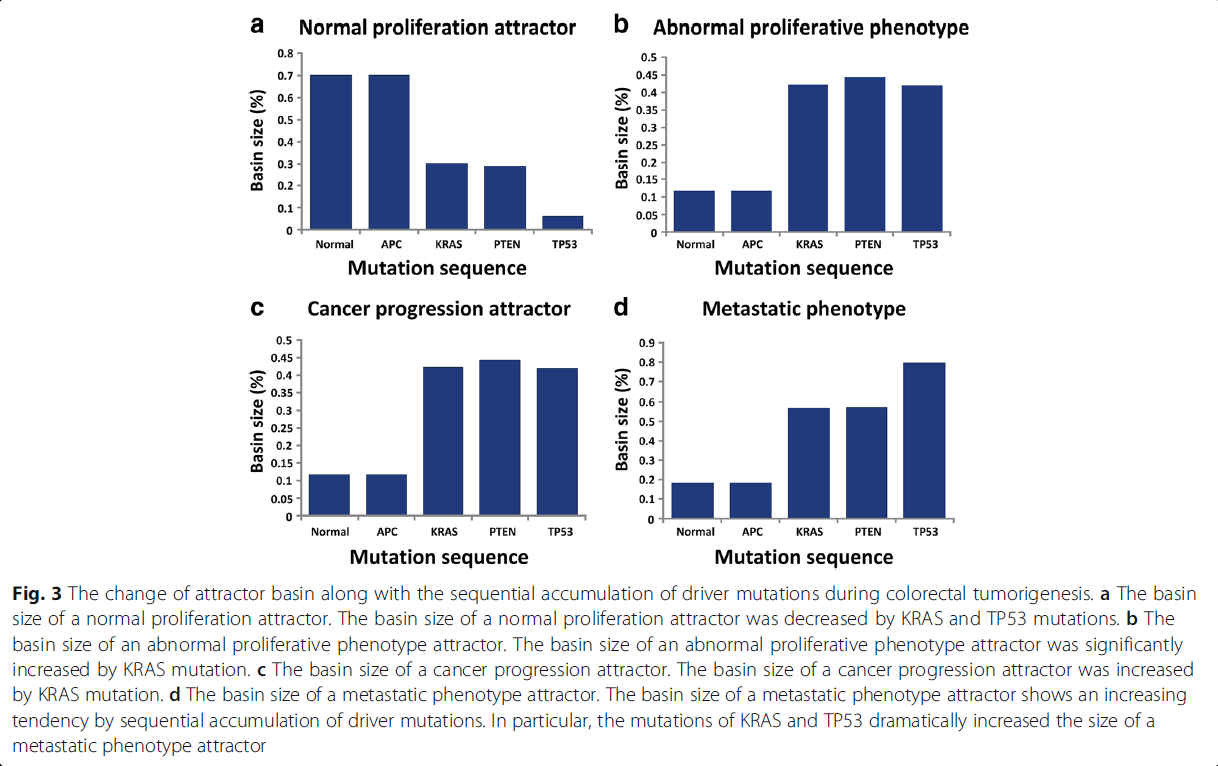
子图名字是对应吸引子的情况,横轴是stage,刚开始正常,APC突变,接着KRAS突变等。我们可以看到条形图变化趋势和生物知识相符,这样我们就验证了结直肠癌是串行突变的结果。
③ 肿瘤逆转
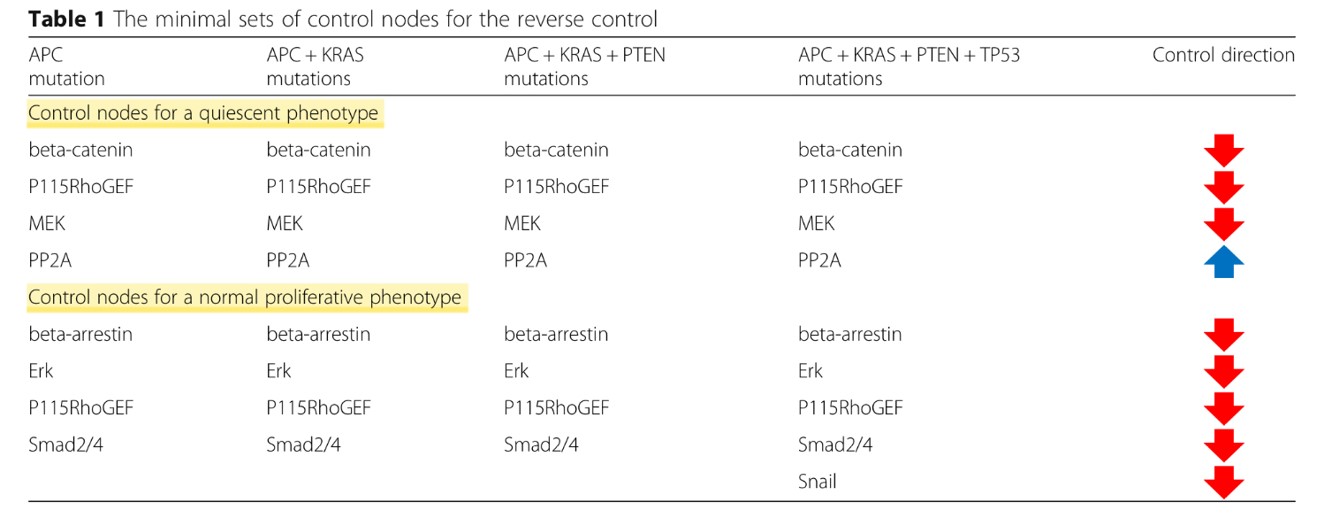
为了找到驱动肿瘤细胞发生逆转的控制节点,他们借鉴了他们之前提出的一个算法识别最小的节点集合来确保所有的初始状态收敛到期望的attractor,这里的期望attractor是细胞类型,如正常增殖等。他们使用遗传算法来找最少控制节点,选出了最优的节点。最后将最小节点集合与FDA批准的靶向药物进行富集分析,及将最小节点集合与随机选择的节点进行了统计分析(one-sided two-sample chi-squared tests)。
具体来说,在选择控制节点时,控制目标是将癌细胞最终变为静止或者正常增殖的表型,下图是不同的累积突变及控制逆向转化的节点分析:
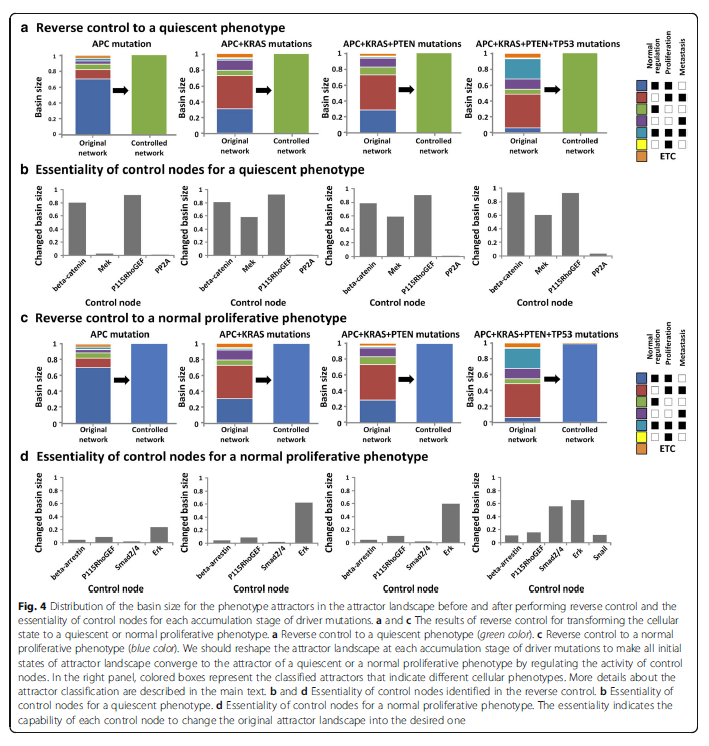
我们可以看出在不同的突变情况下,驱动逆向转化为特定的表型的控制节点是哪些。汇总表格如下:
最后对用算法识别出来的控制节点和批准的靶向药物进行了富集分析,发现逆转为静止状态的靶点和药物靶点符合较好,但是逆转为正常增殖状态的靶点和药物靶点符合度不是很高,和随机选出的节点差不多,分析图如下:
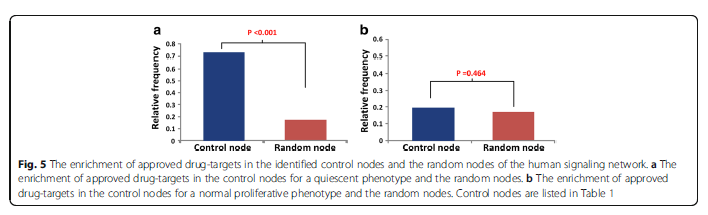
左图是逆转为静止态的,右图是逆转为正常增殖态的。
这种方法为找到药物靶点提供了新的insight。
(BB一句,统计学真重要啊!)
KAIST 2017 Quantitative evaluation and reversion analysis of the attractor landscapes of an intracellular regulatory network for colorectal cancer
本篇论文和上一篇目的一样,都是分析结直肠癌及其逆转。
与上一篇文章基于信号通路的网络不同,这篇文章是基于分子网络,通过landscape来分析结直肠癌的特征及识别癌症逆转的药物靶点。大体流程相同,具体方法不同,应用布尔网络模型去描述分子交互的逻辑关系,通过扰动网络来观察landscape的变化,从而得到癌症逆转的控制节点,结果与先前的实验观察一致。
① 构造分子调控网络
基于KEGG数据库构建了基本分子调控网络(34个节点,135条边),基于信号通路,又增加了3个节点(EGF, Wnt, DNA damage),还有8个marker nodes(Snail, SLUG, MMP, E-cadherin, CyclinE, CyclinD, p21 and Caspase-3),这是为了分出网络中结果吸引子的生物状态,最后又基于文献和PPI数据加入了额外的边。
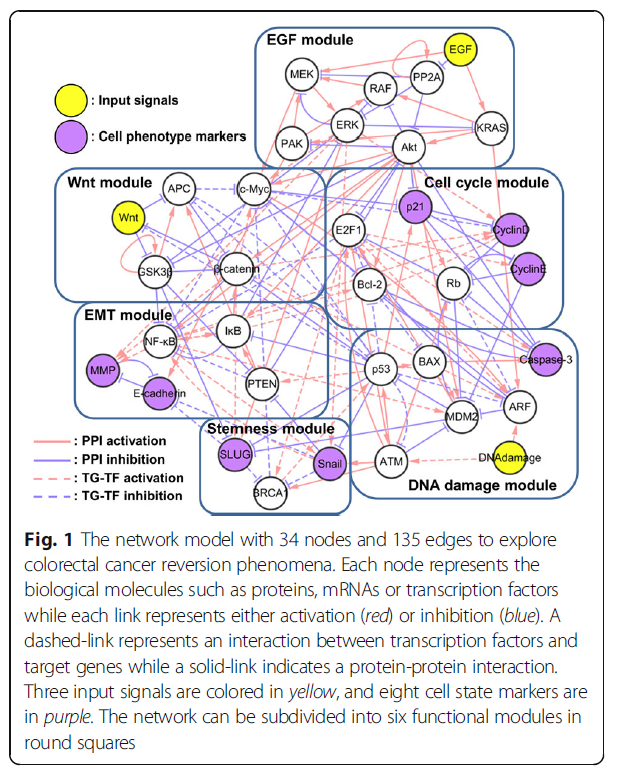
最终构建出来的网络如上图(值得学习的一点是,这里融合了多种生物相关的数据,构建出来的网络更加准确,类似多模态数据)。
② 网络动态性
基于生物行为机制,转化为了加权和逻辑,下一状态Vit+1由输入到节点i的权重和(Ws)i决定:
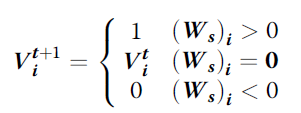
③ attractor分析
因为网络太大,很难获得整个状态转移表,这里通过随机采样来估计attractor landscape(随机定义初始值,看不同的轨迹发展情况),这里同样验证了网络的输入输出关系来评价网络。
比较有意思的是这篇文章在landscape上定义了一个肿瘤恶性程度的分数,基于8个marker node来定义,就是对attractor state进行分类(量化),基于CPSs(cancer proximity scores),再细化就是三个指标(增殖,EMT,干细胞特性),低分代表癌化,高分代表正常。
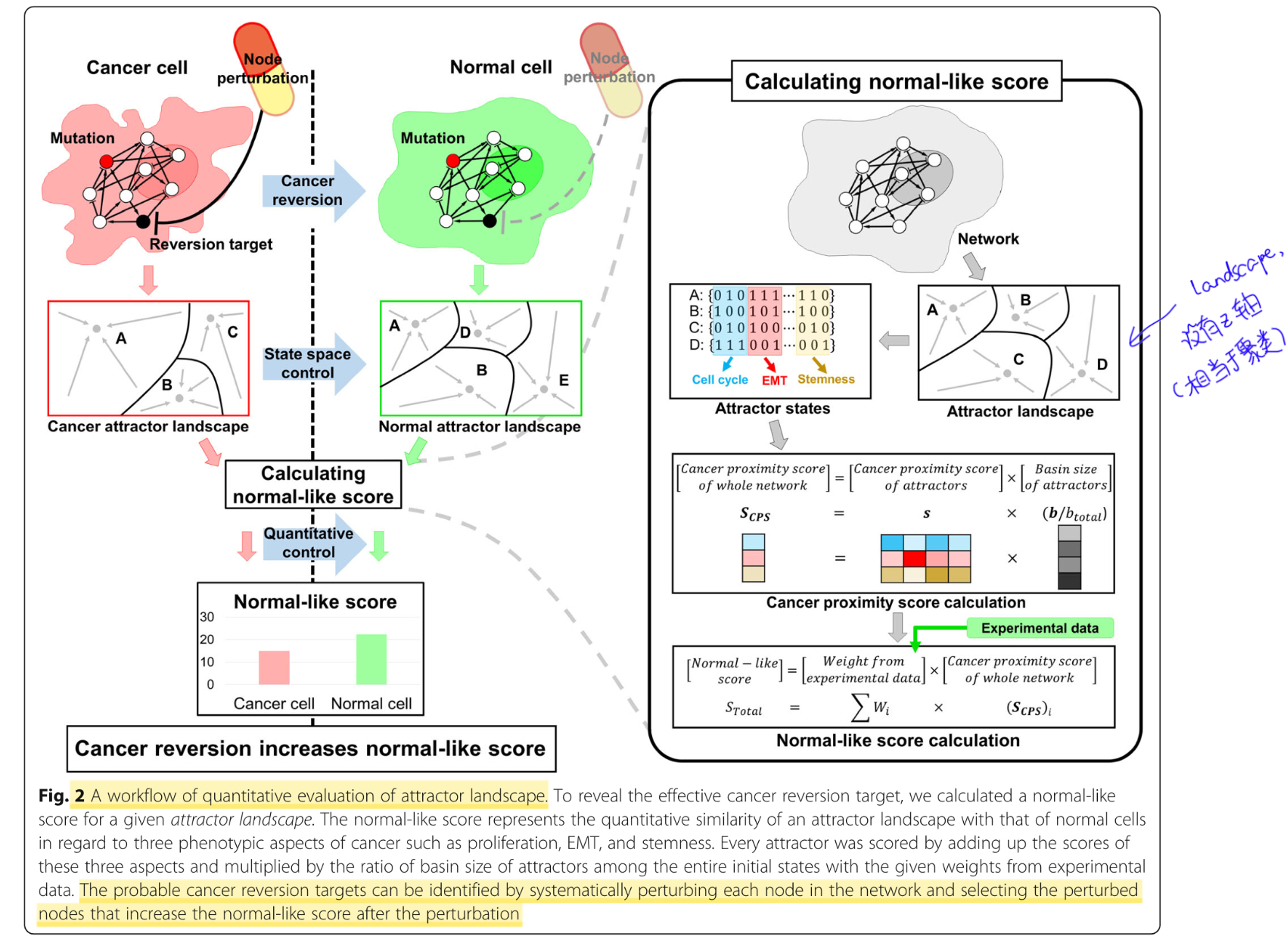
④ 扰动性分析
可以基于上面的打分机制来识别驱动癌症逆转的靶点,当扰动单个节点导致normal-like分数变高,说明该节点可以驱动cancer reversion。这里有单节点扰动和双节点扰动,因为有可能两个节点之间协同作用。扰动有3种类型,激活,抑制和恢复扰动。双节点扰动的类型又可以分为3种,协同,相加和对抗。
功能性网络motif,可以被理解为是癌症逆转的功能单元块。如果网络中的一个节点突变了,扰动任何motif中的其他节点都不能导致癌症逆转。但是,其他网络motif中的节点扰动可以导致癌症逆转。我们通过下述过程提取功能性网络motif:首先,计算扰动一个癌症逆转靶点A导致的norm-like增加的分数,然后固定单一节点,通过A再计算一次,接着选择一个固定节点B,该节点极大减少norm-like分数,接着固定B,再找一个类似B的节点C,重复这个过程直到扰动A造成的norm-like分数的增加可以被忽略,这些节点就组成了一个功能性网络motif。
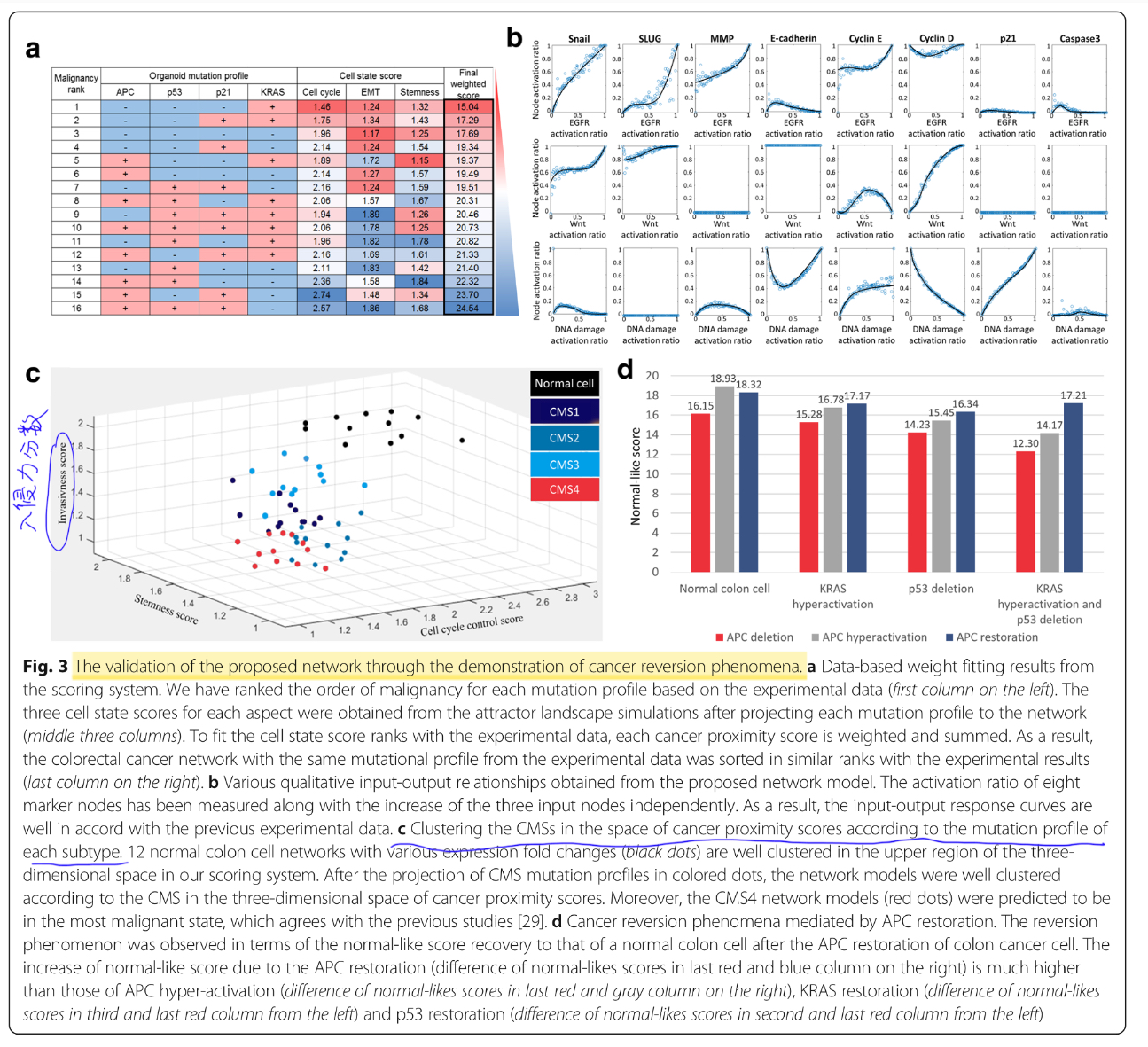
扰动分析结果如下:
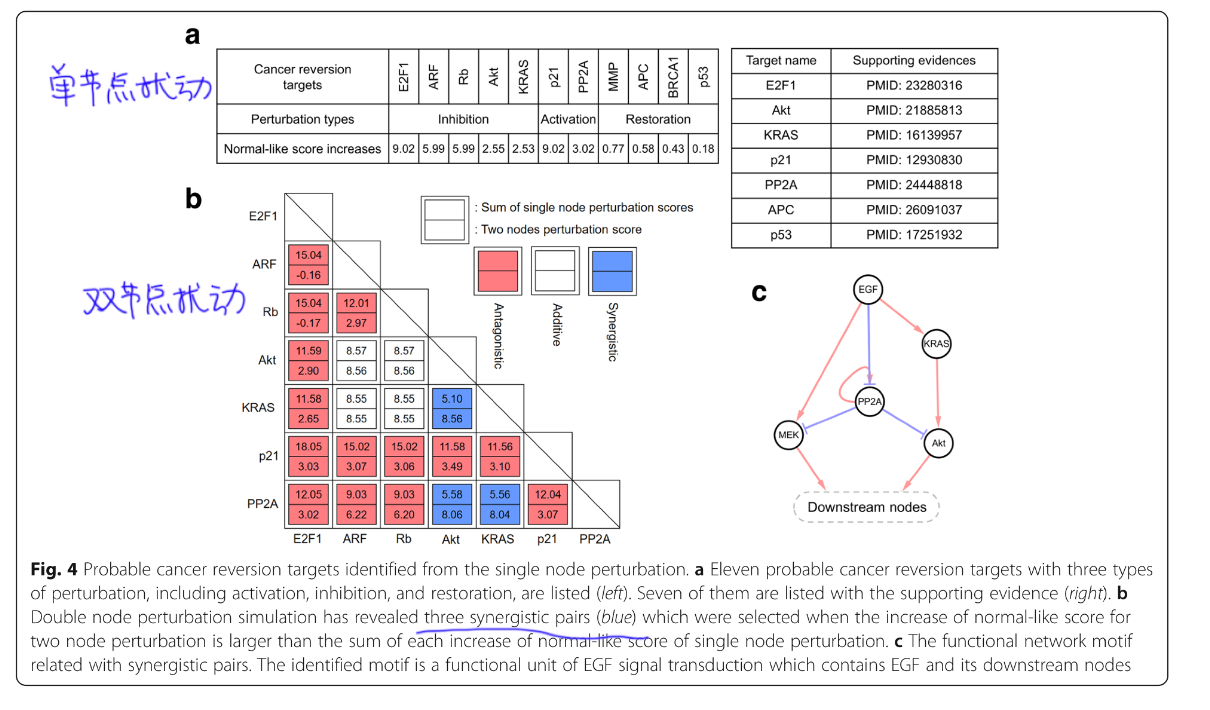
下图是11个癌症逆转靶标的4个稳定的motif,这些motif用于增强癌症状态中吸引子对外部扰动的稳定性。
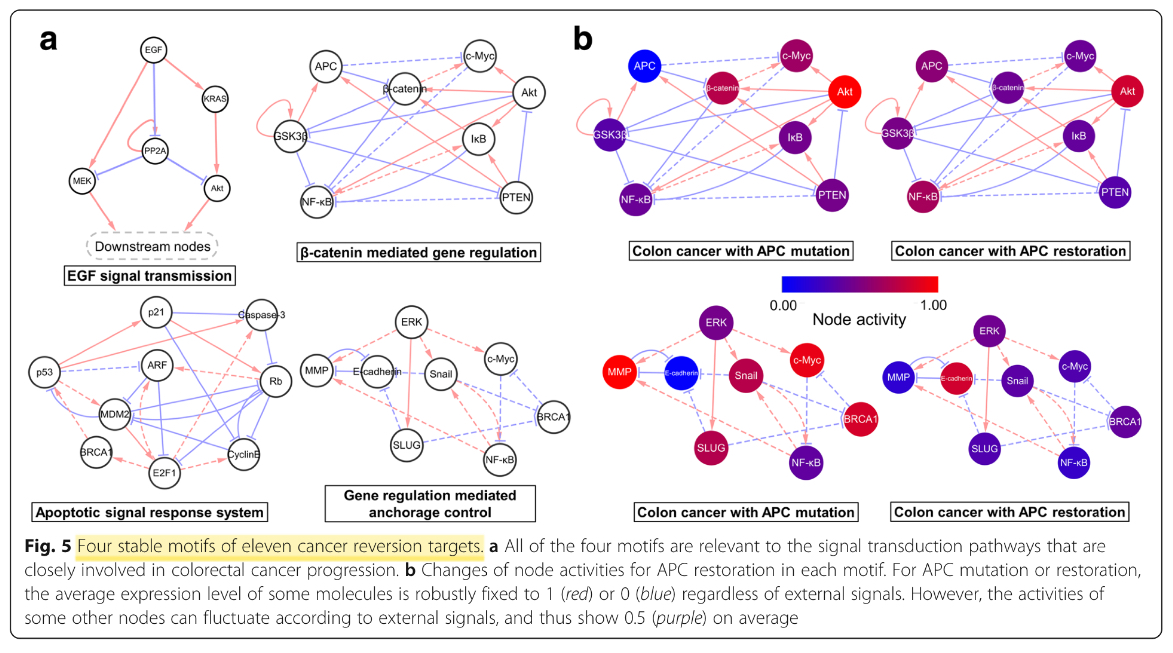
⑤ 稳定性分析
为判断网络对外部扰动的稳定性,我们检验了吸引子的个数及5个主要吸引子的平均basin大小。如果吸引子数比较少或者basin大小比较大,外部扰动很难改变网络状态。
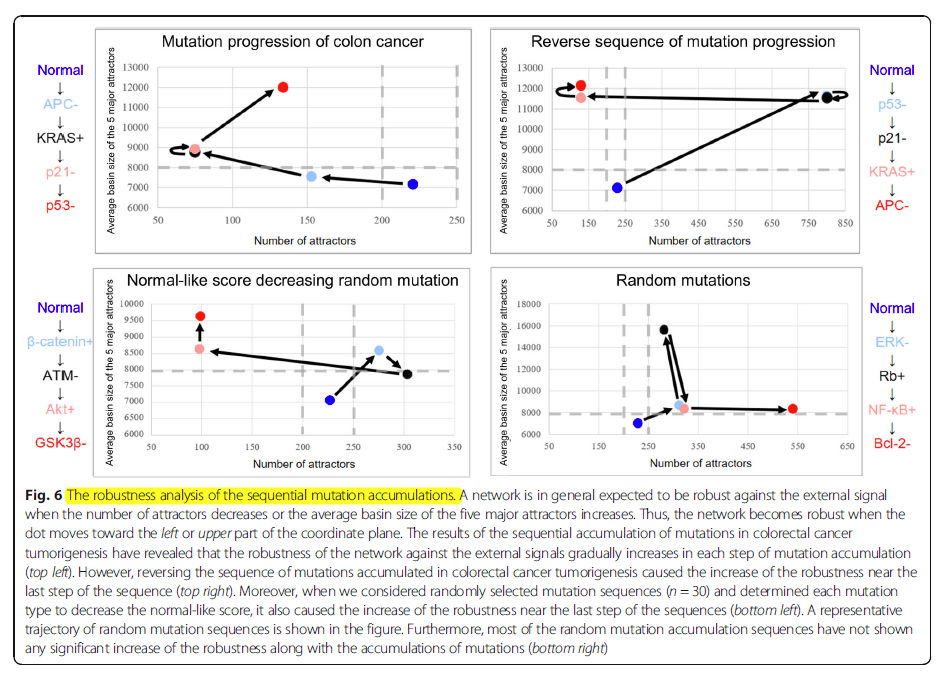
结果说明癌症逆转的关键就是通过扰动稳定的癌细胞的functional motif来增强其对外部信号的响应。
Review 2019 Attractor – a new turning point in drug discovery
这篇文章是应用attractor landscape进行药物发现的一个简单综述,可能因为做这个点的人比较少,还没有引用量。但是对整个流程有一个清晰的概括,值得一看。
吸引子指的就是系统的稳态,吸引子周围所有的不平衡状态随着系统的演化最终都会进入吸引子状态。从系统动力学的角度看药物发现和设计,可以应用吸引子的概念。
和attractor相关的生物医学研究范例:① attractor代表的一些生物重要性:如Kauffman的attractor-cell type;Huang的attractor-stable cell phenotype ② 利用attractor理论研究疾病治疗:如前面介绍的几篇文章。
药物发现和设计的整个历史进程:
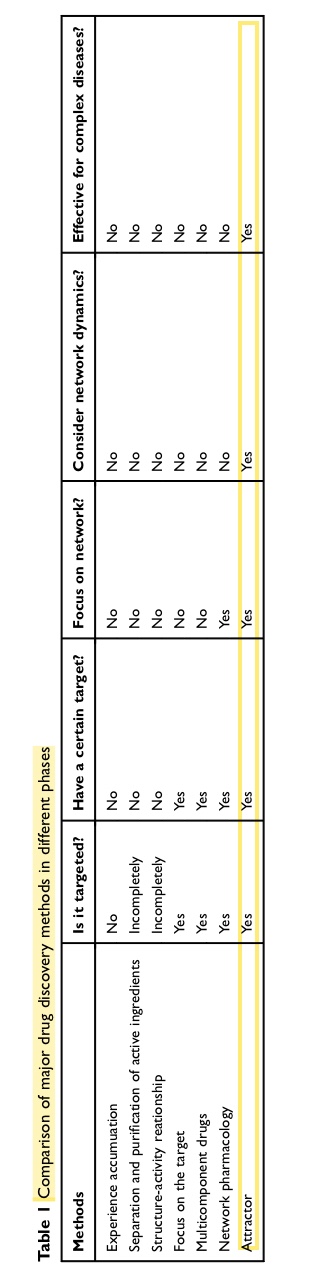
① 21世纪前,药物发展4阶段:Phase I 经验积累 Phase II 分离和纯化 Phase III 结构活性关系(SAR) Phase IV 靶点为中心
② 21世纪后,生物网络等
吸引子的方法可能是下一个趋势。
吸引子分析的pipeline:
1. 构造基因调控网络(如之前的布尔网络)
2. 计算attractor和计算工具:几个计算吸引子状态的算法,BoolNet, GINsim, ADAM, ATLANTIS

3. 构造和分析attractor landscape
4. 实现状态改变(扰动):通常有三种扰动方法,基因干预,外部干预和结构干预。基因干预指的是使用特定的方法去抑制基因表达,或破坏其结构使其不能表达。外部干预指的是在不改变网络结构的前提下改变控制基因在当前时刻的状态,一些外部干预算法,mean-first-passage-time (MFPT) algorithm, the steady-state distribution (SSD) and conservative steadystate distribution (CSSD) algorithm, the basin of attraction (BOA) algorithm based on the attractor, the unconstrained (UC) algorithm and the phenotypically constrained (PC) algorithm. 结构干预是改变网络,如消除特定节点间的调控关系,目前最简单最常见的方法就是one-bit Boolean function 干预,特定节点的状态翻转。

5. 实现药物发现和设计:在前述分析中,发现可以被干预的潜在靶点和扰动方法之后,药物设计便水到渠成。
Regan 2017 Modeling the attractor landscape of disease progression: a network-based approach
这篇论文其实就是他们之前一篇提出构建landscape方法论文(“Characterizing cancer subtypes as attractors of Hopfield networks”)的应用,应用在疾病数据集上进行分析的文章。这篇文章就是用他们之前提出的方法构造attractor landscape然后分析了三种疾病的发展,帕金森,神经胶质瘤和结直肠癌。
如在帕金森数据集上的landscape可视化:
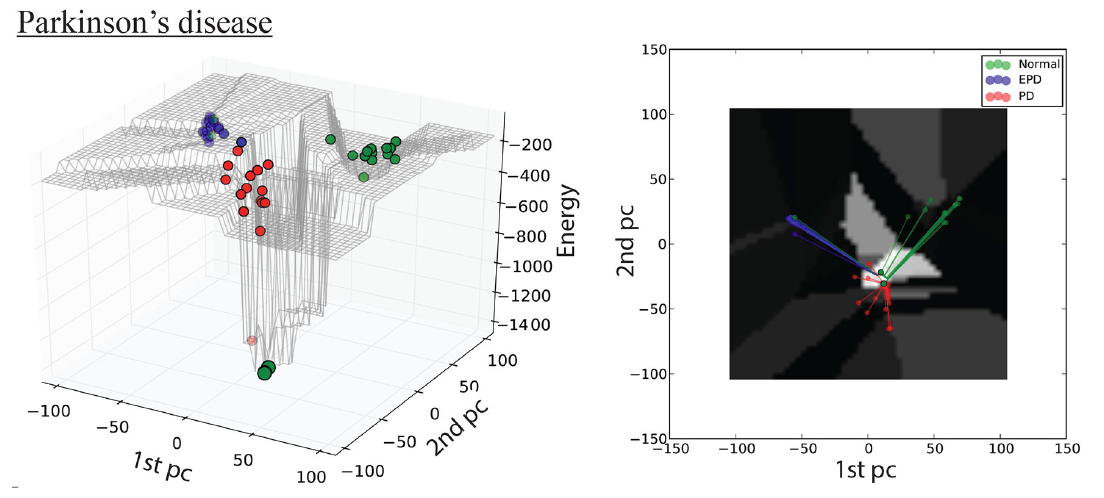
我们可以看到随着病情的加重,样本有向attractor发展的趋势。
下面再简要总结一下本篇论文用到的方法:
因为基于Hopfield网络,就是一个全连接网络,其权重矩阵就是一个基因维度的方阵,这里权重矩阵W用Pearson相关系数来表示,易知W是对称的,且对角线为1(因为基因自身的相关性为1),所以这里将对角线元素-1使得W对角线为0。网络的动态性定义为P(t+1)=sgn(P(t)*W), P(t)是在t时刻网络的表达谱,P(t)逐渐变化趋于稳态,能量函数是E[P(s)]=-0.5*P*W*PT。最后用PCA降维,这样就可以画出landscape。最后谈一下相关的分析方法,这里扰动性分析是直接作用在W上的,给W中特定边的权重赋随机的值。衡量attractor宽度和深度的方法,宽度就是一个吸引子中所有样本间的距离,有许多距离计算方法,求和,最小,最大,平均pair对距离,所有点和中心点的距离,这里用的是平均pair对间的欧式距离,深度就是样本收敛前后的能量差。
最后mark一点就是构建好attractor landscape之后,分析时考虑其自身的一些特性,比如attractor basin的宽度和深度,basin越宽说明大多数细胞的类型都是其对应的类型,可以反应肿瘤细胞的异质性,深度可以反应其不容易被扰动(robustness特性),如肿瘤细胞。下面用一张图(来自上一篇review)来解释深度和robustness之间的关系:
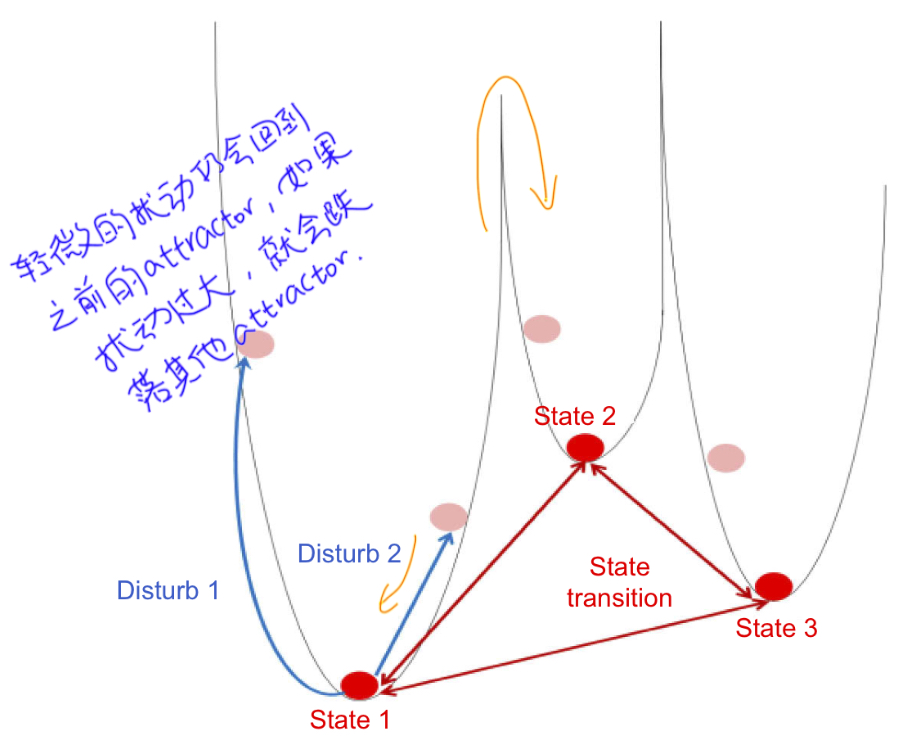
当深度越深,其稳定性就越强,轻微的扰动不会改变其中细胞的命运。
Sum up: 最后最后自己总结一下,用attractor landscape来分析疾病发展和做药物发现呢,根据这5篇论文可以分为两种方法,第一种就像Regan的那篇数据驱动,直接用Hopfield网络,但是权重(e.g. Pearson相关系数),更新规则需要自己建模(数学公式),第二种呢就是方法驱动,比如KAIST机构都是手工建立布尔网络(基于已有的model或者数据库里的进行补充),包括网络的拓扑结构以及动态性(布尔表达式),这一步对于非生物背景的可能还是比较难。有了网络之后呢,我们就可以找网络的稳态,吸引子的位置(和聚类有一点像,但是聚类是静态的过程),接着就可以分析attractor的属性,宽度(异质性),深度(robustness)等来解释生物意义。接下来,很重要的一步就是进行扰动,KAIST的这几篇文章中,基本上都是在网络中有几个输入节点,通过输入不同的信号达到扰动的目的,从而观察attractor的变化(用属性来定量化变化),得出特定基因与phenotype之间的关系,这样就可以得到相应的靶点(这一块理论上还是有点复杂,和控制论,强化学习有关,比如topic“如何找到最小的控制节点集驱动sample从一个attractor到另外一个attractor”)。为了验证靶点,可以与批准药物的靶点做富集分析之类的。
[1]. Chu H, Lee D, Cho K H. Precritical state transition dynamics in the attractor landscape of a molecular interaction network underlying colorectal tumorigenesis[J]. PloS one, 2015, 10(10): e0140172.
[2]. Cho S H, Park S M, Lee H S, et al. Attractor landscape analysis of colorectal tumorigenesis and its reversion[J]. BMC systems biology, 2016, 10(1): 96.
[3]. Kim Y, Choi S, Shin D, et al. Quantitative evaluation and reversion analysis of the attractor landscapes of an intracellular regulatory network for colorectal cancer[J]. BMC systems biology, 2017, 11(1): 1-11.
[4]. Hou X, Li M, Jia C, et al. Attractor–a new turning point in drug discovery[J]. Drug design, development and therapy, 2019, 13: 2957.
[5]. Taherian Fard A, Ragan M A. Modeling the attractor landscape of disease progression: a network-based approach[J]. Frontiers in genetics, 2017, 8: 48.


 浙公网安备 33010602011771号
浙公网安备 33010602011771号