openGauss源码解析(103)
openGauss源码解析:SQL引擎源解析(18)
2. 种群初始化
在使用遗传算法前,可以利用参数Gepo_threshold的数值来调整触发的条件。为了方便代码解读,将这个边界条件降低至4(即RelOptInfo数量或者说基表数量为4的时候就尝试使用遗传算法)。下面在解读代码的过程中,以t1,t2,t3,t4四个表为例进行说明。
RelOptInfo作为遗传算法的基因,首先需要进行基因编码,openGauss数据库采用实数编码的方式,也就是用{1,2,3,4}分别代表t1,t2,t3,t4这4个表。
然后通过gimme_pool_size函数来获得种群的大小,种群的大小受Geqo_pool_size和Geqo_effort两个参数的影响,种群用Pool结构体进行表示,染色体用Chromosome结构体来表示。代码如下:
/* 染色体Chromosome结构体 */
typedef struct Chromosome {
/* string实际是一个整型数组,它代表基因的一种排序方式,也就对应一棵连接树 */
/* 例如{1,2,3,4}对应的就是t1 JOIN t2 JOIN t3 JOIN t4 */
/* 例如{2,3,1,4}对应的就是t2 JOIN t3 JOIN t1 JOIN t4 */
Gene* string;
Cost worth; /* 染色体的适应度,实际上就是路径代价 */
} Chromosome;
/* 种群Pool结构体 */
typedef struct Pool {
Chromosome *data; /* 染色体数组,数组中每个元组都是一个连接树 */
int size; /* 染色体的数量,即data中连接树的数量,由gimme_pool_size生成 */
int string_length; /* 每个染色体中的基因数量,和基表的数量相同 */
} Pool;
另外通过gimme_number_generations函数来获取染色体交叉的次数,交叉的次数越多则产生的新染色体也就越多,也就更可能找到更好的解,但是交叉次数多也影响性能,用户可以通过Geqo_generations参数来调整交叉的次数。
在结构体中确定的变量如下。
(1) 通过gimme_pool_size确定的染色体的数量(Pool.size)。
(2) 每个染色体中基因的数量(Pool.string_length),和基表的数量相同。
然后就可以开始生成染色体,染色体的生成采用的是Fisher-Yates洗牌算法,最终生成Pool.size条染色体。具体的算法实现如下:
/* 初始化基因序列至{1,2,3,4} */
for (i = 0; i < num_gene; i++)
tmp[i] = (Gene)(i + 1);
remainder = num_gene - 1; /* 定义剩余基因数 */
/* 洗牌方法实现,多次随机挑选出基因,作为基因编码的一部分 */
for (i = 0; i < num_gene; i++) {
/* choose value between 0 and remainder inclusive */
next = geqo_randint(root, remainder, 0);
/* output that element of the tmp array */
tour[i] = tmp[next]; /* 基因编码 */
/* and delete it */
tmp[next] = tmp[remainder]; /* 将剩余基因序列更新 */
remainder--;
}
表6-18是生成一条染色体的流程,假设4次的随机结果为{1,1,1,0}。
表6-18 生成染色体的流程
基因备选集 Tmp | 结果集 tour | 随机数 范围 | 随机数 | 说明 |
1 2 3 4 | 2 | 0~3 | 1 | 随机数为1,结果集的第一个基因为tmp[1],值为2,更新备选集tmp,将未被选中的末尾值放在前面被选中的位置上 |
1 4 3 | 2 4 | 0~2 | 1 | 随机数为1,结果集的第二个基因为4,再次更新备选集tmp |
1 3 | 2 4 3 | 0~1 | 1 | 随机数为1,结果集的第三个基因为3,由于末尾值被选,无须更新备选集 |
1 | 2 4 3 1 | 0~0 | 0 | 最后一个基因为1 |
在多次随机生成染色体后,得到一个种群,假设Pool种群中共有4条染色体,用图来描述其结构,如图6-13所示。
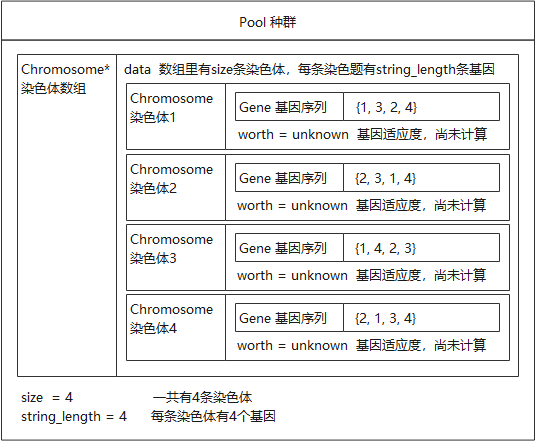
图6-13 染色体结构




 浙公网安备 33010602011771号
浙公网安备 33010602011771号