整合多组学先验信息来提升肉牛基因组预测的准确性
研究目标
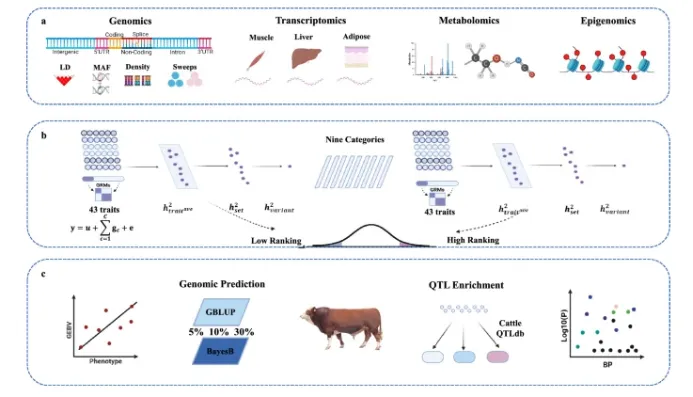
研究方法
- 数据整合
-
- 基因组• :eQTL(表达数量性状位点)
- 代谢组• :开放染色质区(OCR)、低甲基化区(HMR)
- 功能变异贡献评估
- • 计算每个变异的“功能评分”,筛选前5%、10%、30%为高影响变异(top sets)。
- 预测模型比较
- GWAS与QTL富集分析
-
关键结果
- 功能变异贡献
- 保守区选择信号• (如启动子、UTR)虽仅占0.11%基因组,但解释6.22%遗传方差(172.96倍富集)。
- 代谢组数据
- 预测准确性提升
- Top 10%变异集
- BayesB模型• :平均提升7.54%。
- BayesB
- GWAS验证
- 体重相关QTL
结论
- • 选择信号、保守区及调控变异是肉牛复杂性状遗传架构的核心。
-
创新点
- 进化、表观、转录、代谢• 提出策略,突破传统WGS预测瓶颈。
文章链接:
DOI: 10.1186/s13062-024-00574-y
![]()
本文来自博客园,作者:生物信息与育种,转载请注明原文链接:https://www.cnblogs.com/miyuanbiotech/p/19011733。若要及时了解动态信息,请关注同名微信公众号:生物信息与育种。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号