NC | STICI:用于基因型插补的深度学习transformer框架,可试试育种应用
目前各种方法已被广泛用于基因型插补,但要插补某些基因组区域和大型结构变异仍然具有挑战性。2025年1月,《Nature Communications 》发表了一个基于transformer的框架——STICI,用于精确的基因型插补。
基因型成本仍然是育种的重要痛点之一,填补方法没有很好地用起来,可以试试这个。

STICI是什么?![]()
STICI是一种新的基因型插补模型,该模型基于变换器框架和卷积块中的注意力机制。该模型利用注意力来捕捉所分析的基因组集合中SNV和SV之间的模式。开发团队发现STICI以适中的内存消耗成本实现了高插补精度,这是通过将数据划分为块来实现的,这使得STICI能够有效地应用于长序列。此外,与其他DL模型不同,STICI只需要训练一次。经过这次训练,STICI中的插补时间与使用经典方法相当或更快。
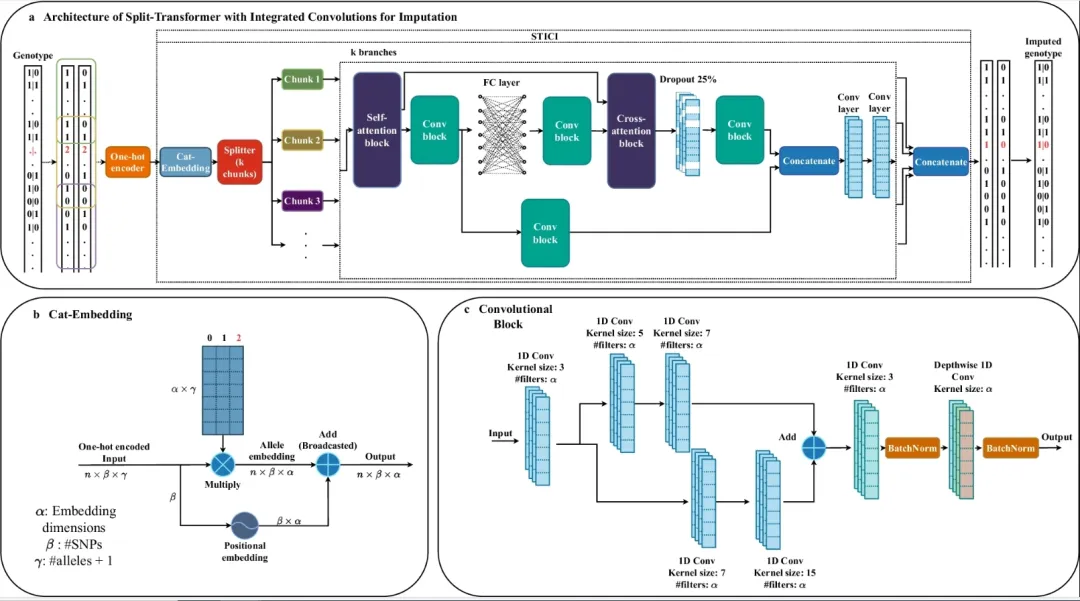
STICI的架构
STICI的性能测试![]()
STICI模型自动学习全基因组连锁不平衡模式,在具有高度连锁变异的地区,插补精度要高得多。开发团队在千人基因组计划和非人类基因组上的插补结果表明,STICI可以实现与最先进的基因型插补方法相当的高插补精度,并具有插补多等位基因变异和各种类型遗传变异的额外能力。STICI可以利用自我监督功能针对任何基因组集合进行自动训练。此外,STICI无需对非人类基因组集合中的基本模式进行任何特殊预设,就能显示出卓越的性能,这表明STICI 可适应和应用于任何物种中缺失基因型的推算。
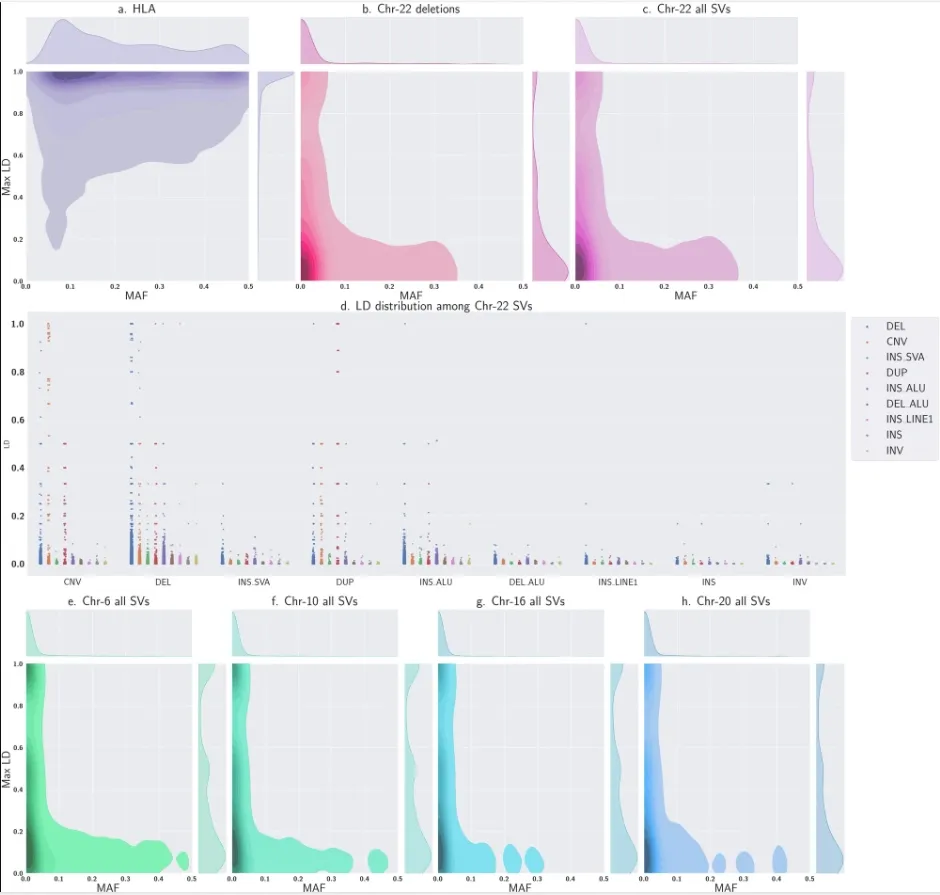
千人基因组计划基准数据集的MAF和LD分布
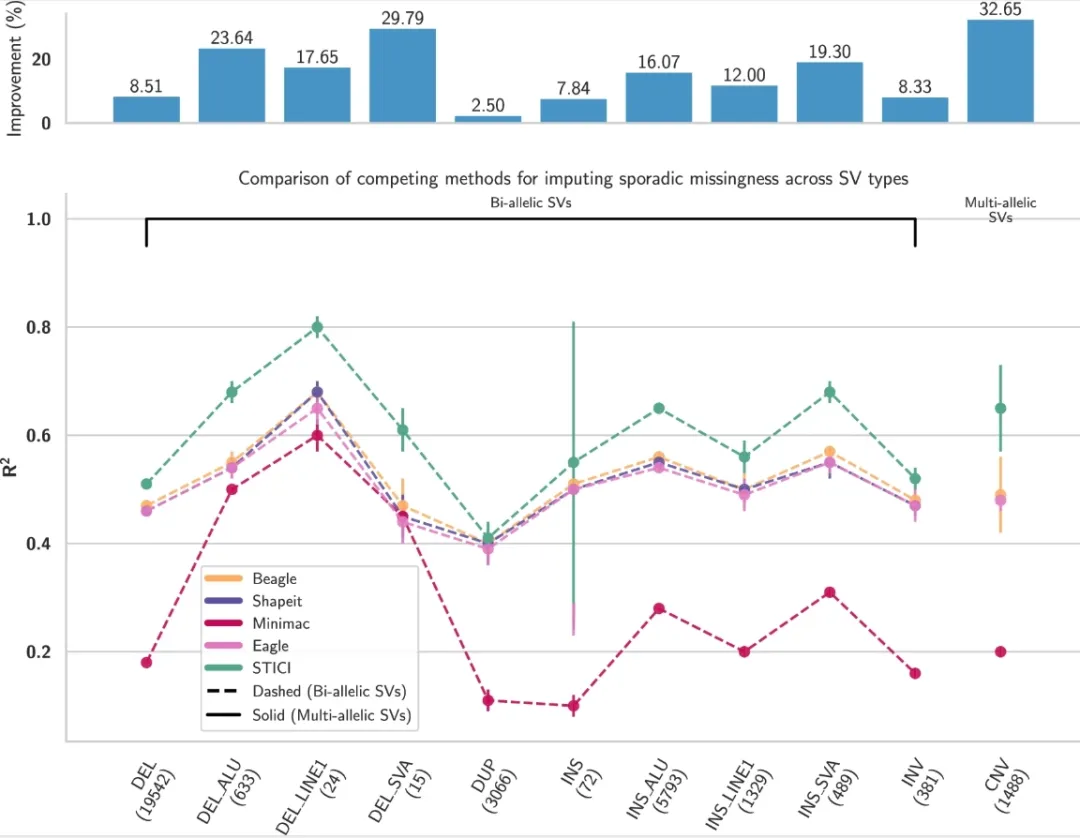
比较不同 SV 类型的竞争方法的零星插补结果
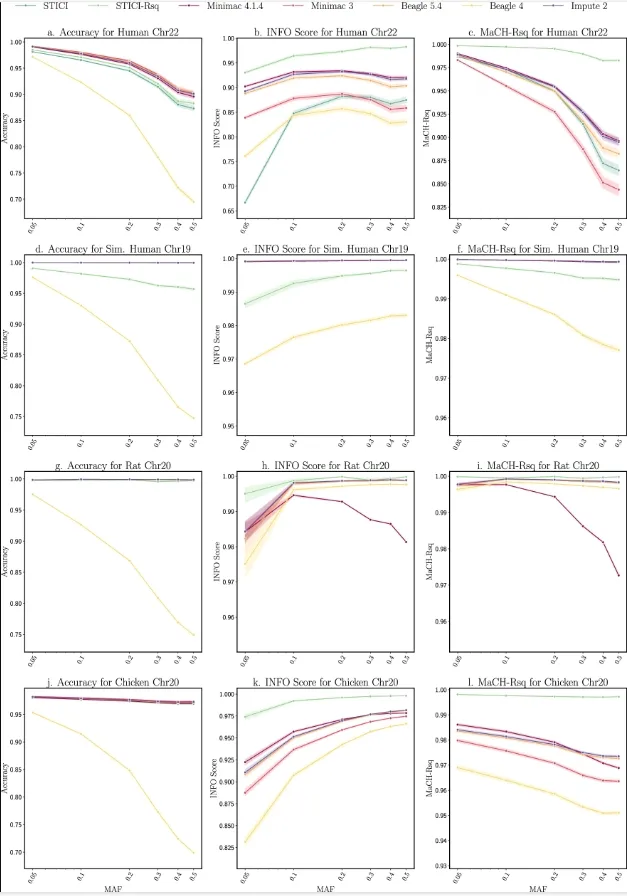
跨不同数据集的系统缺失值插补结果
综上,STICI是一个专门用于解决基因型插补问题的深度学习transformer框架;其插补不需要标准参考panel,这使得它更普遍地适用于任何数据集;其擅长SV插补,其中变异具有更高的复杂性,同时实现了与SNV插补竞争模型相当的性能。
STICI的源代码已在GitHub以及Zenodo上公开发布:
👉 https://github.com/shilab/STICI
参考文献:
Mowlaei, M.E., Li, C., Jamialahmadi, O. et al. STICI: Split-Transformer with integrated convolutions for genotype imputation. Nat Commun 16, 1218 (2025). https://doi.org/10.1038/s41467-025-56273-3
本文来自博客园,作者:生物信息与育种,转载请注明原文链接:https://www.cnblogs.com/miyuanbiotech/p/18981211。若要及时了解动态信息,请关注同名微信公众号:生物信息与育种。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号