基因测序中碱基质量值的计算
当使用k-means将碱基聚类后,可使用下面的方法计算碱基质量
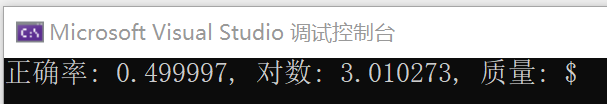
#include <stdio.h> #include <math.h> int main() { double proba = 0.0; // 某点的概率值初始化为0.0 const int num_centers = 4; // 质心数量为4个 double sum_distances = 0.0; // 某点到所属质心的距离除以到每个质心距离的num_centers次方总和初始化为0.0 double distances[num_centers] = { 1, 1, 20, 20 }; // 某点距离四个质心的距离 double label_distance = 1.0; // 某点距离所属质心的距离 for (int i = 0; i < num_centers; i++) sum_distances += pow(label_distance / distances[i], num_centers); // 某点到所属质心的距离除以到每个质心距离的num_centers次方,并累加到总和中 proba = 1.0 / sum_distances; // 计算概率值 double log = -10 * log10(1 - proba); // 计算对数值 char qual = '!' + (log < '(' ? log : '('); // 根据对数值计算一个字符,即质量值 printf("正确率: %f, 对数: %f, 质量: %c\n", proba, log, qual); // 打印概率、对数和质量值 return 0; }
运行结果如下
质量值是Q20,则错误识别的概率是1%,即错误率1%,或者正确率是99%;
质量值是Q30,则错误识别的概率是0.1%,即错误率0.1%,或者正确率是99.9%;
质量值是Q40,则错误识别的概率是0.01%,即错误率0.01%,或者正确率是99.99%;




 浙公网安备 33010602011771号
浙公网安备 33010602011771号