SYRI实现基因组间结构变异可视化
https://www.jianshu.com/p/93eec573e1a0
https://github.com/schneebergerlab/plotsr#genomes
https://github.com/schneebergerlab/syri
比对可以用到minimap2做比对,也可以用mummer做比对处理,两者输出结果都可以容易的通过syri进行可视化。
需要注意的是plotsr可视化工具v1.6和v1.4的参数设置并不相同。
plotsr v1.6功能更加强大。
话不多说,直接开始,流程记录如下。
一、安装
#syri分析工具,plotsr为配套可视化工具
conda create -n syri syri plotsr -y二、两基因组比对
nucmer --mum -t 16 -p refgenome.fa querygenome.fa
delta-filter -i 90 -l 15000 -q out.delta > out.filtered.delta
#此处也可用mummerplot可视化,但点图看不太清楚,只能看大致的比对情况。所以后续尝试用syri作染色体水平结构变异的细致可视化
#格式处理
show-coords -THrd out.filtered.delta > out.filtered.coords三、Syri分类处理处理
需要syri生成输出的syri.out文件,用于plotsr可视化
syri -c out.filtered.coords -d out.filtered.delta -r refgenome.fa -q querygenome.fa四、plotsr可视化
vim genomes.txt
#file name tags
refgenome.fa ref lw:2
querygenome.fa query lw:2plotsr可视化
plotsr \
--sr syri.out \
--genomes genomes.txt \
-o ref_query_findSV.pdf五、效果图
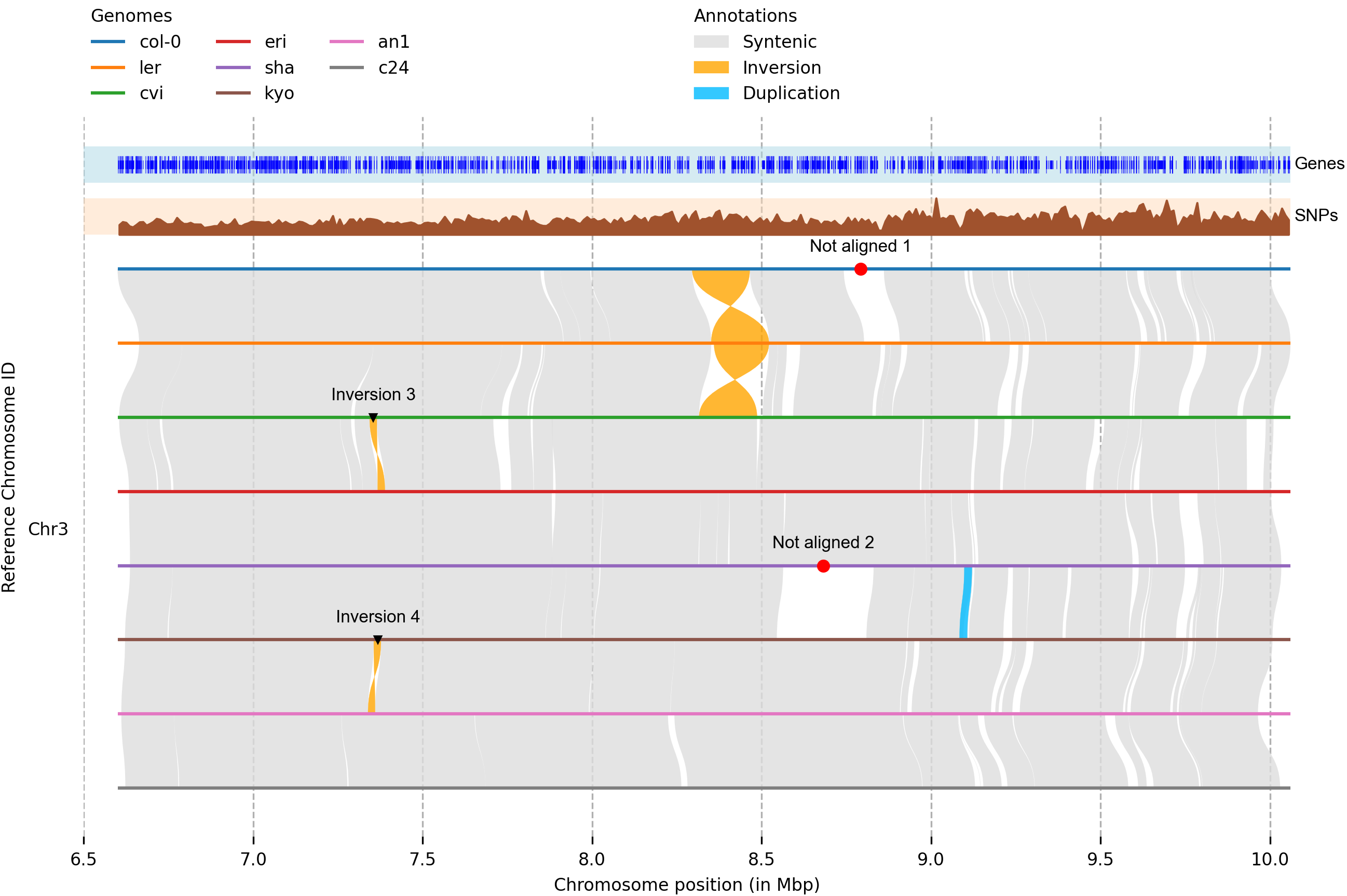
图片来自syri github官网。如下图添加tracks,可以实现更多的可视化效果。




 浙公网安备 33010602011771号
浙公网安备 33010602011771号