20251212组会
将gigapath的分类数据从脑肿瘤换成了卵巢癌
这个卵巢癌的ovarian_cancer分类数据状况,是一个五分类任务,数据分布情况如下
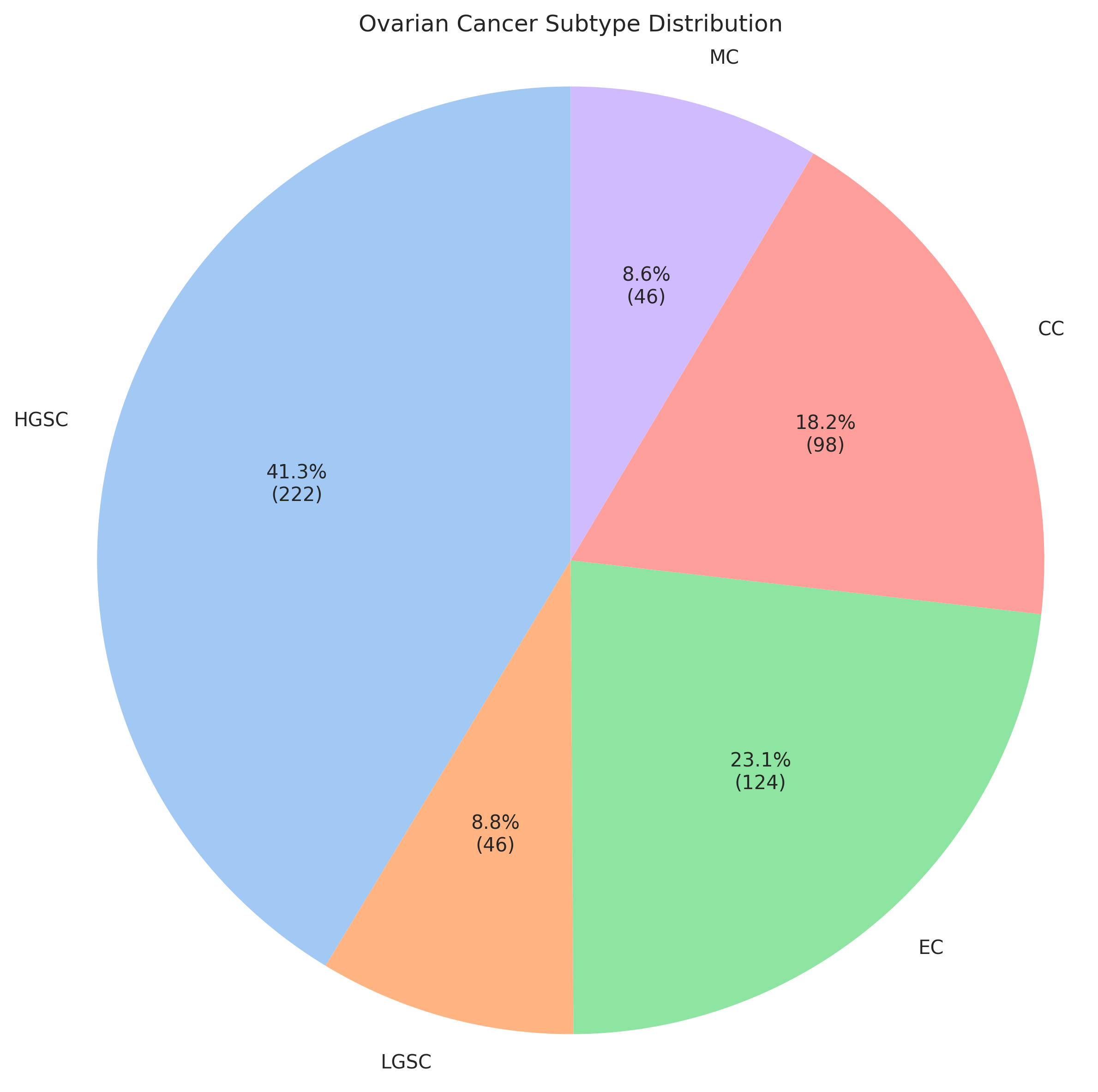
可以看出数据分布不均,如果直接对分布不均的数据做逻辑回归,可能会导致分类效果不佳
为此我们采用了以下方法
我们手写了一个线性层来对模型进行分类,其实我说白了, 本质是逻辑回归就是让数据经过一个softmax的线性层
当 MC 样本被分错时(假设模型预测该 MC 样本是 MC 的概率 pt = 0.3):
普通 Focal Loss :
惩罚项 = (1 - 0.3)^2 = 0.7^2 = 0.49 倍的 Loss。
针对 MC 的激进 Focal Loss (gamma=4.5):
惩罚项 = (1 - 0.3)^4.5 = 0.7^4.5 约等于0.20 倍的 Loss
这里有个常见的误解。 Focal Loss 的公式是 -(1-pt)^gamma*log(pt)。
关注Gradient的变化。gamma 越大,当 pt 接近 0 时(分类错误错),梯度会保持较大;而当 pt 接近 1 时(分对),梯度会衰减得极快。
更直观的理解是:高 Gamma 会让模型对“还没学好”(pt 低)的样本产生极大的 Loss 占比,而完全忽略那些已经学得还可以(pt > 0.5)的样本。
如果我们将 MC 的 gamma 设为 4.5:
只要 MC 的预测概率不是 非常高(比如 > 0.8),模型就会觉得“我很痛”,Loss 很大。
这会迫使优化器疯狂调整权重,直到 MC 的预测概率被推得非常高为止。
我们设计了一组参数
gamma_values = [2.0, 2.0, 2.0, 2.5, 2.8]
其中LGSC和MC的惩罚参数是2.5和2.8这样设计
由于LGSC和MC的占比较小,在分类时的混淆率较高,故需要增强分类错误的惩罚强度
(这组参数我后面慢慢微调)
分类后的混淆矩阵如下
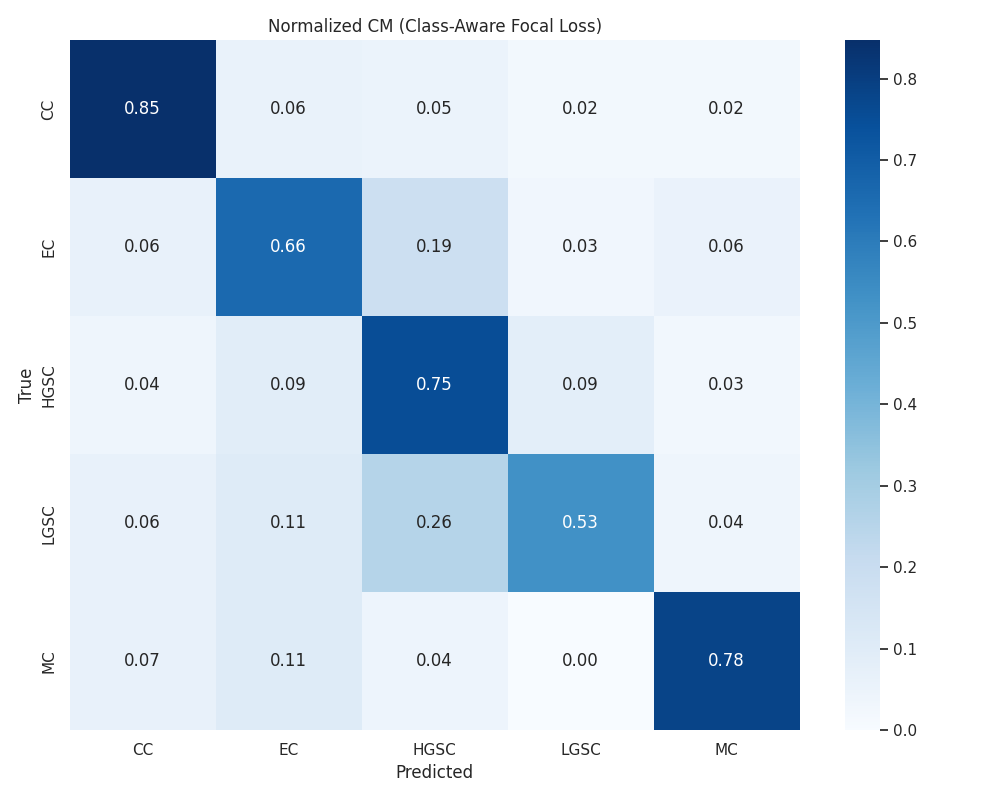
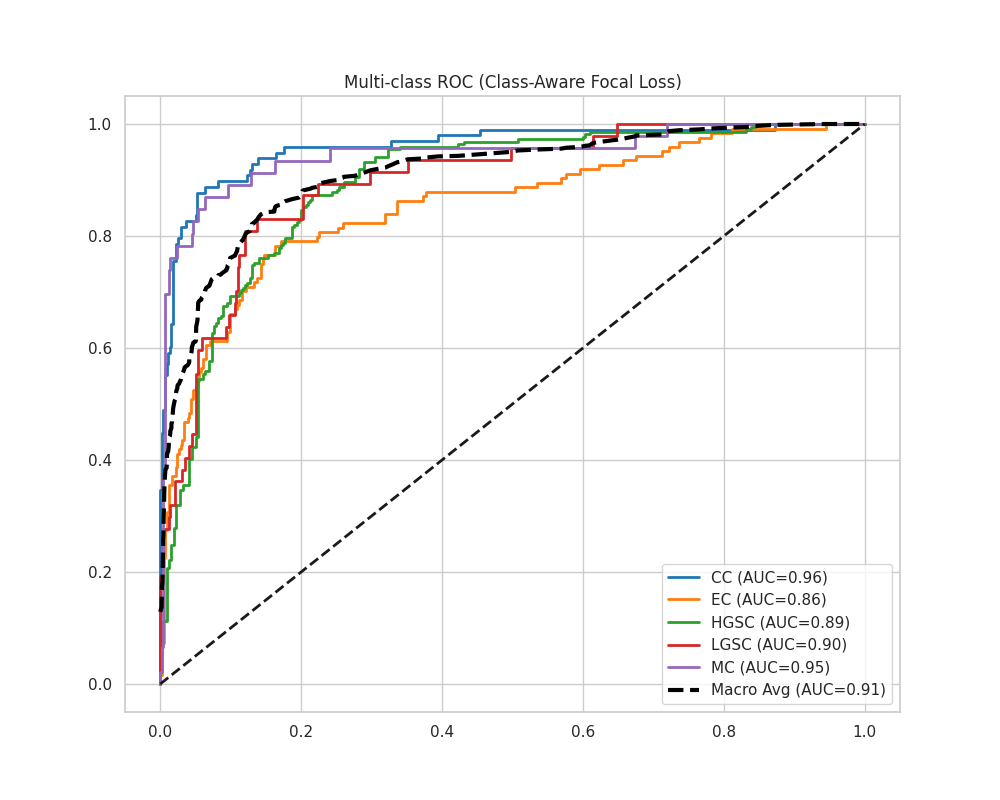
可以发现,在对大多数类进行分类的时候,模型准确率都极高,说明前面的gigapath模型极好的分析出来了这个病理切片的差异,模型本身很强大
只是这个数据集某些数据量实在太小,导致模型分类效果不佳
我还考虑了另外一个层面的原因
我们可以看到,模型在lgsc和HGSC两者之间的分类效果较差,准确率仅为0.53
我仔细研究了LGSC和HGSC两个亚型的在组织学上的差异
lgsc是低级别浆液性癌,而hgsc是高级别
这两种癌形在组织学上的差异很小,唯一的差异在细胞核上
具体来说hgsc的细胞核已经出现明显的异性了,而lgsc的细胞核还没有出现明显的异形
如果直接放大图片,观察细胞核是很糊的
现在有两个选择,一个是选择对图片进行局部增强,
另外一个是在后续分类的时候引入一些图片相关的基因组层面的信息,做一个多专家模型
以上




 浙公网安备 33010602011771号
浙公网安备 33010602011771号