PCA 主成分分析实践 gcta软件
1、测试数据下载
链接:https://pan.baidu.com/s/1EfffExvtxZYI1QLuxUZQ_g
提取码:5wfe
数据为plink 格式数据test.map、test.ped ;
一共包含三个品种,DOR、GMM、SUN各20个样本。
2、下载gcta软件,官网:
https://cnsgenomics.com/software/gcta/#Overview
wget https://cnsgenomics.com/software/gcta/bin/gcta_1.93.1beta.zip

解压:

3、将测试数据转化为二进制:
plink --file test --make-bed --sheep --out test

4、
4、运行gcta:
gcta64 --bfile test --make-grm --autosome-num 26 --out tmp
注意 --autosome-num 设定染色体数目
gcta64 --grm tmp --pca 5 --out pca
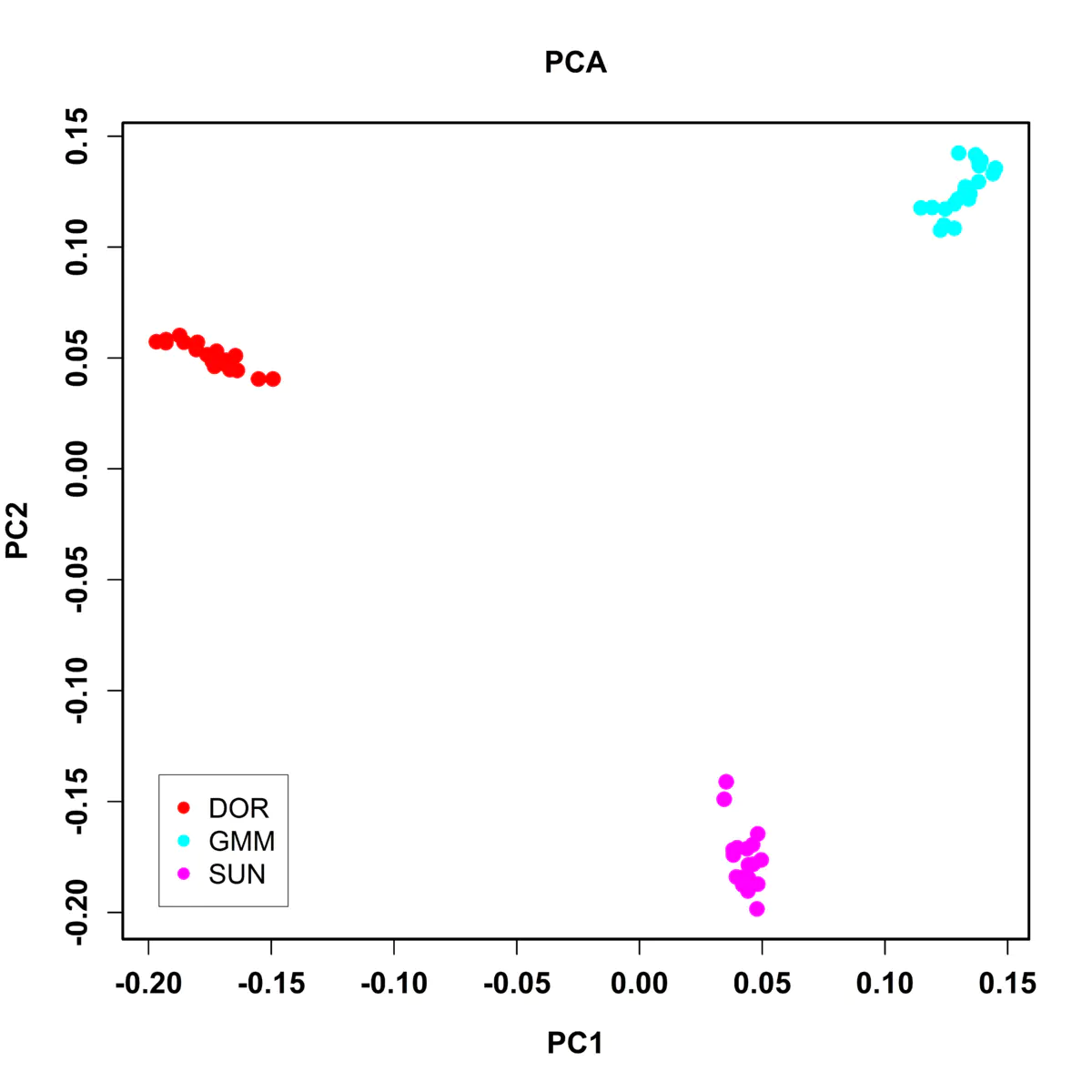
5、根据pca.eigenvec文件绘图
mydat<-read.table("pca.eigenvec",as.is = T,header = F,stringsAsFactors = F)
png("PCA.png",width = 7000,height = 7000,pointsize = 160)
plot(mydat$V3,mydat$V4,lwd.ticks=10,font.axis=2,cex.lab=1.2,cex.axis=1.2,font.lab=2,ylab="PC2",xlab = "PC1",main="PCA",cex=1.4,pch=19,col=c(rep("red",20),rep("cyan",20),rep("magenta",20)))box(which="plot",col="black",lwd=18)
legend("bottomleft",box.lwd=5,cex=1.1,inset = 0.04,c("DOR","GMM","SUN"),pch=19,col=c("red","cyan","magenta"))
dev.off()
绘图结果:




 浙公网安备 33010602011771号
浙公网安备 33010602011771号