异常检测:K-Means算法
K-Means算法是一个聚类算法,在样本没有类别的情况下可以通过算法聚出类别,比如某公司中有大量的用户,现在需要我们将用户划分出类别,但是现在我们并不知道怎么划分更好,这个时候就可以通过K-Means算法来帮助我们划分出类别。其原理是通过计算样本到质心的距离来对样本聚出类别,其中的距离我们可以理解为样本间的相似性,也就是“物以类聚”。
其中提到的质心是通过所有样本点的均值来计算出来的,如果存在异常值那么就会导致质心的计算出现偏差。进而影响结果。如图2-1:在没有异常值的情况下的质心的计算和聚类结果:
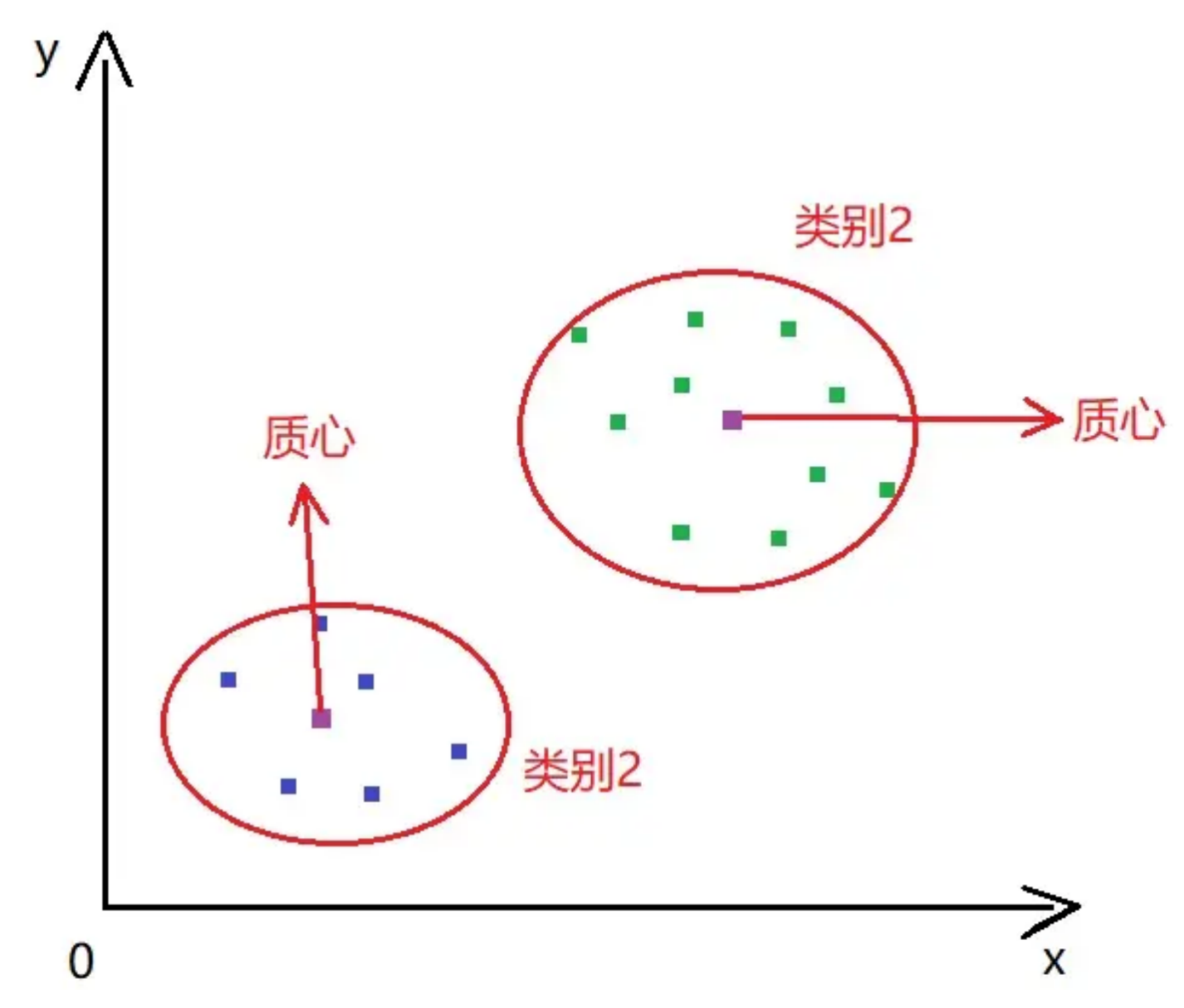
图2-1
通过图2-1我们可以看出聚类的结果还是比较理想的,接下来我们引入一个异常值,再看下聚类结果的变化,如图2-2:
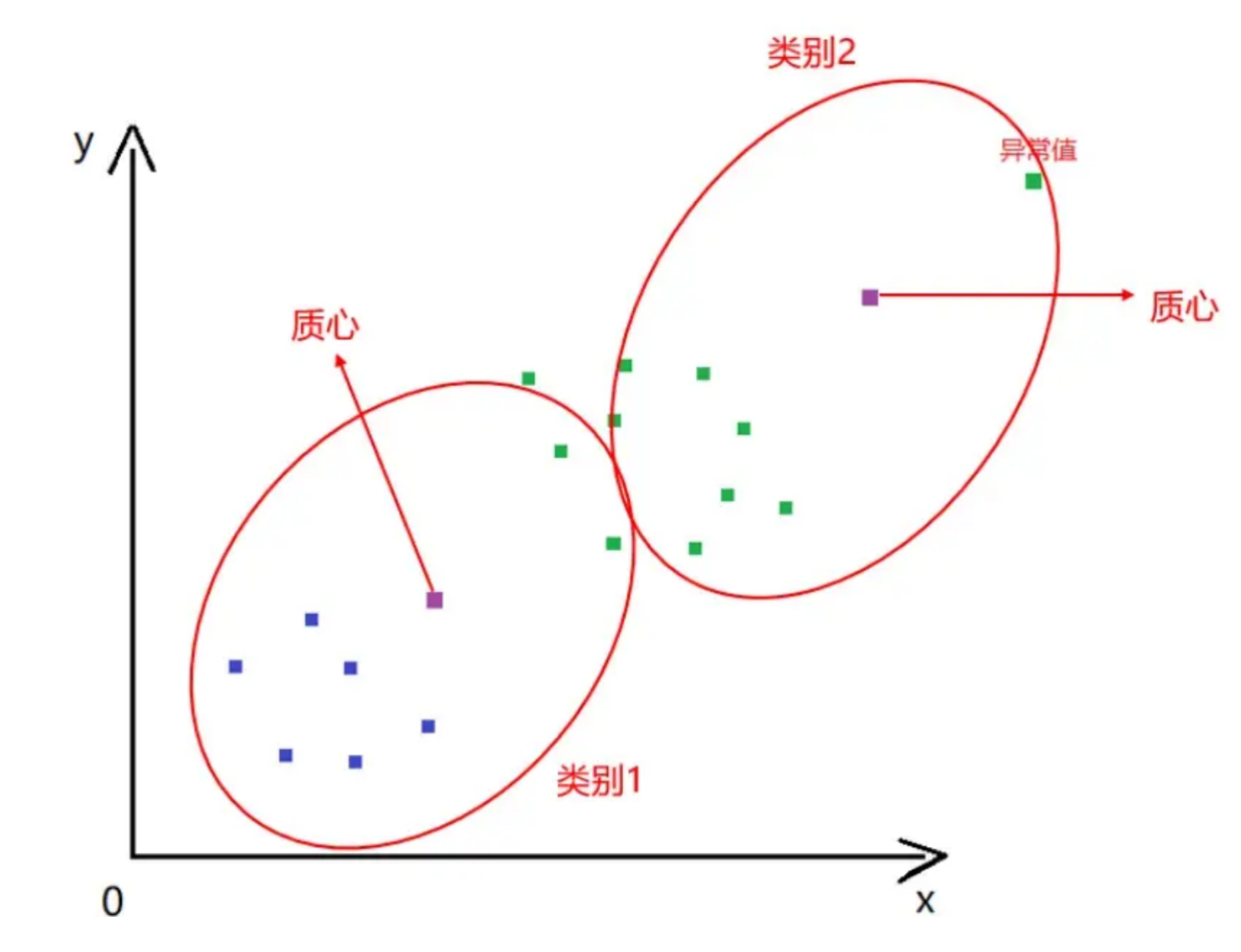
图2-2
通过图2-2可以看出,异常值导致聚类的结果出现了错误,让本应该是类别2的样本分到了类别1中。
因此在机器学习中异常值的存在对算法最终的结果影响还是很大的,那么就需要我们在机器学习中对异常值进行处理。
一、K-Means算法原理
K-Means算法:
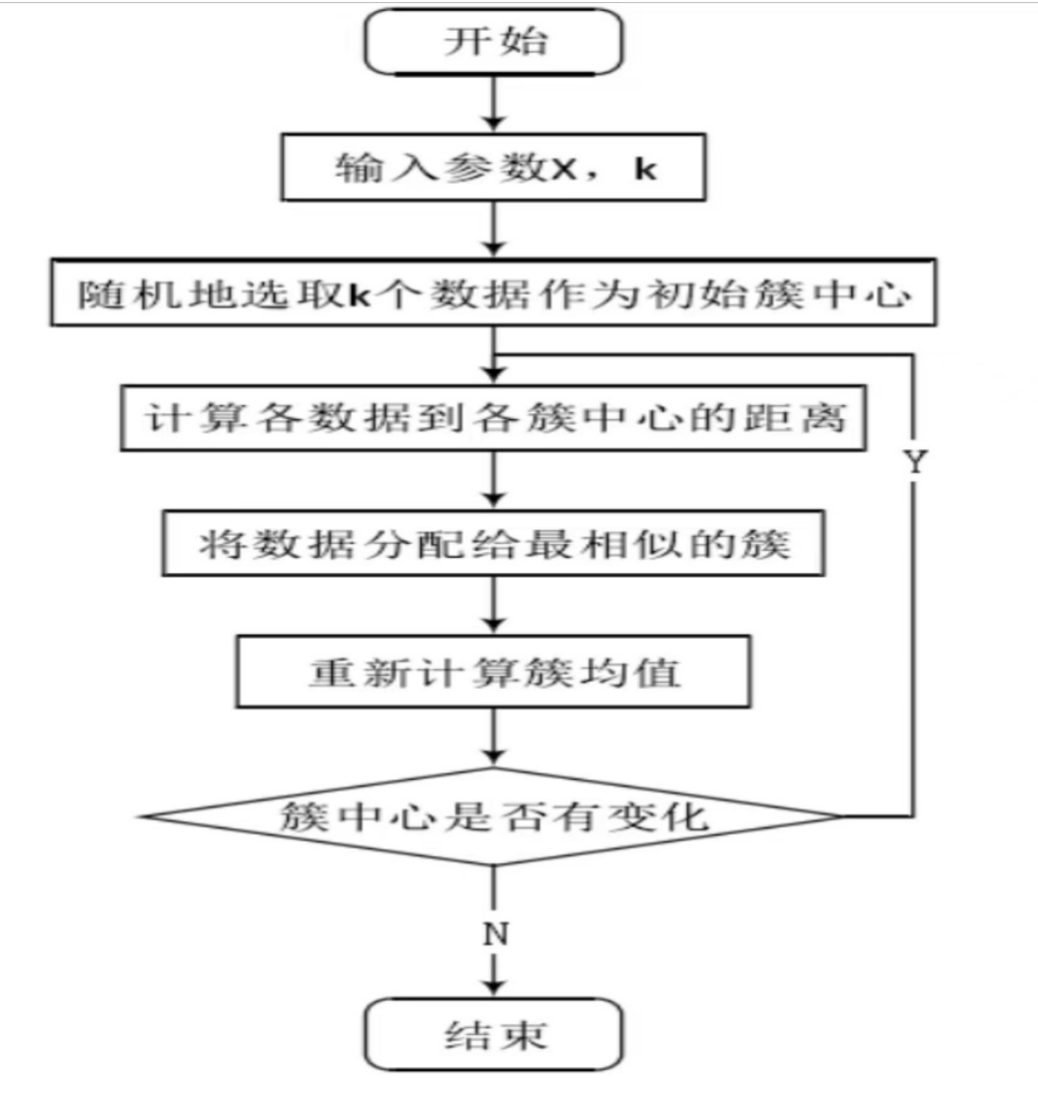
图2-3
如图2-3所示:
1. 对于给定的一组数据,随机初始化K个聚类中心(簇中心)
2. 计算每个数据到簇中心的距离(一般采用欧氏距离),并把该数据归为离它最近的簇。
3. 根据得到的簇,重新计算簇中心。
4. 对步骤2、步骤3进行迭代直至簇中心不再改变或者小于指定阈值。
二、K-means实现(Python3)
本文使用的数据集为UCI数据集,分别使用鸢尾花数据集Iris、葡萄酒数据集Wine、小麦种子数据集seeds进行测试,本文从UCI官网上将这三个数据集下载下来,并放入和python文件同一个文件夹内即可。同时由于程序需要,将数据集的列的位置做出了略微改动。数据集具体信息如下表:
代码如下:
import time
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
from numpy import nonzero, array
from sklearn.cluster import KMeans
from sklearn.metrics import f1_score, accuracy_score, normalized_mutual_info_score, rand_score, adjusted_rand_score
from sklearn.preprocessing import LabelEncoder
from sklearn.decomposition import PCA
# 数据保存在.csv文件中
iris = pd.read_csv("dataset/Iris.csv", header=0) # 鸢尾花数据集 Iris class=3
wine = pd.read_csv("dataset/wine.csv") # 葡萄酒数据集 Wine class=3
seeds = pd.read_csv("dataset/seeds.csv") # 小麦种子数据集 seeds class=3
wdbc = pd.read_csv("dataset/wdbc.csv") # 威斯康星州乳腺癌数据集 Breast Cancer Wisconsin (Diagnostic) class=2
glass = pd.read_csv("dataset/glass.csv") # 玻璃辨识数据集 Glass Identification class=6
df = iris # 设置要读取的数据集
columns = list(df.columns) # 获取数据集的第一行,第一行通常为特征名,所以先取出
features = columns[:len(columns) - 1] # 数据集的特征名(去除了最后一列,因为最后一列存放的是标签,不是数据)
dataset = df[features] # 预处理之后的数据,去除掉了第一行的数据(因为其为特征名,如果数据第一行不是特征名,可跳过这一步)
attributes = len(df.columns) - 1 # 属性数量(数据集维度)
original_labels = list(df[columns[-1]]) # 原始标签
def initialize_centroids(data, k):
# 从数据集中随机选择k个点作为初始质心
centers = data[np.random.choice(data.shape[0], k, replace=False)]
return centers
def get_clusters(data, centroids):
# 计算数据点与质心之间的距离,并将数据点分配给最近的质心
distances = np.linalg.norm(data[:, np.newaxis] - centroids, axis=2)
cluster_labels = np.argmin(distances, axis=1)
return cluster_labels
def update_centroids(data, cluster_labels, k):
# 计算每个簇的新质心,即簇内数据点的均值
new_centroids = np.array([data[cluster_labels == i].mean(axis=0) for i in range(k)])
return new_centroids
def k_means(data, k, T, epsilon):
start = time.time() # 开始时间,计时
# 初始化质心
centroids = initialize_centroids(data, k)
t = 0
while t <= T:
# 分配簇
cluster_labels = get_clusters(data, centroids)
# 更新质心
new_centroids = update_centroids(data, cluster_labels, k)
# 检查收敛条件
if np.linalg.norm(new_centroids - centroids) < epsilon:
break
centroids = new_centroids
print("第", t, "次迭代")
t += 1
print("用时:{0}".format(time.time() - start))
return cluster_labels, centroids
# 计算聚类指标
def clustering_indicators(labels_true, labels_pred):
if type(labels_true[0]) != int:
labels_true = LabelEncoder().fit_transform(df[columns[len(columns) - 1]]) # 如果数据集的标签为文本类型,把文本标签转换为数字标签
f_measure = f1_score(labels_true, labels_pred, average='macro') # F值
accuracy = accuracy_score(labels_true, labels_pred) # ACC
normalized_mutual_information = normalized_mutual_info_score(labels_true, labels_pred) # NMI
rand_index = rand_score(labels_true, labels_pred) # RI
ARI = adjusted_rand_score(labels_true, labels_pred)
return f_measure, accuracy, normalized_mutual_information, rand_index, ARI
# 绘制聚类结果散点图
def draw_cluster(dataset, centers, labels):
center_array = array(centers)
if attributes > 2:
dataset = PCA(n_components=2).fit_transform(dataset) # 如果属性数量大于2,降维
center_array = PCA(n_components=2).fit_transform(center_array) # 如果属性数量大于2,降维
else:
dataset = array(dataset)
# 做散点图
label = array(labels)
plt.scatter(dataset[:, 0], dataset[:, 1], marker='o', c='black', s=7) # 原图
# plt.show()
colors = np.array(
["#FF0000", "#0000FF", "#00FF00", "#FFFF00", "#00FFFF", "#FF00FF", "#800000", "#008000", "#000080", "#808000",
"#800080", "#008080", "#444444", "#FFD700", "#008080"])
# 循换打印k个簇,每个簇使用不同的颜色
for i in range(k):
plt.scatter(dataset[nonzero(label == i), 0], dataset[nonzero(label == i), 1], c=colors[i], s=7, marker='o')
# plt.scatter(center_array[:, 0], center_array[:, 1], marker='x', color='m', s=30) # 聚类中心
plt.show()
if __name__ == "__main__":
k = 3 # 聚类簇数
T = 100 # 最大迭代数
n = len(dataset) # 样本数
epsilon = 1e-5
# 预测全部数据
labels, centers = k_means(np.array(dataset), k, T, epsilon)
F_measure, ACC, NMI, RI, ARI = clustering_indicators(original_labels, labels) # 计算聚类指标
print("F_measure:", F_measure, "ACC:", ACC, "NMI", NMI, "RI", RI, "ARI", ARI)
draw_cluster(dataset, centers, labels=labels)
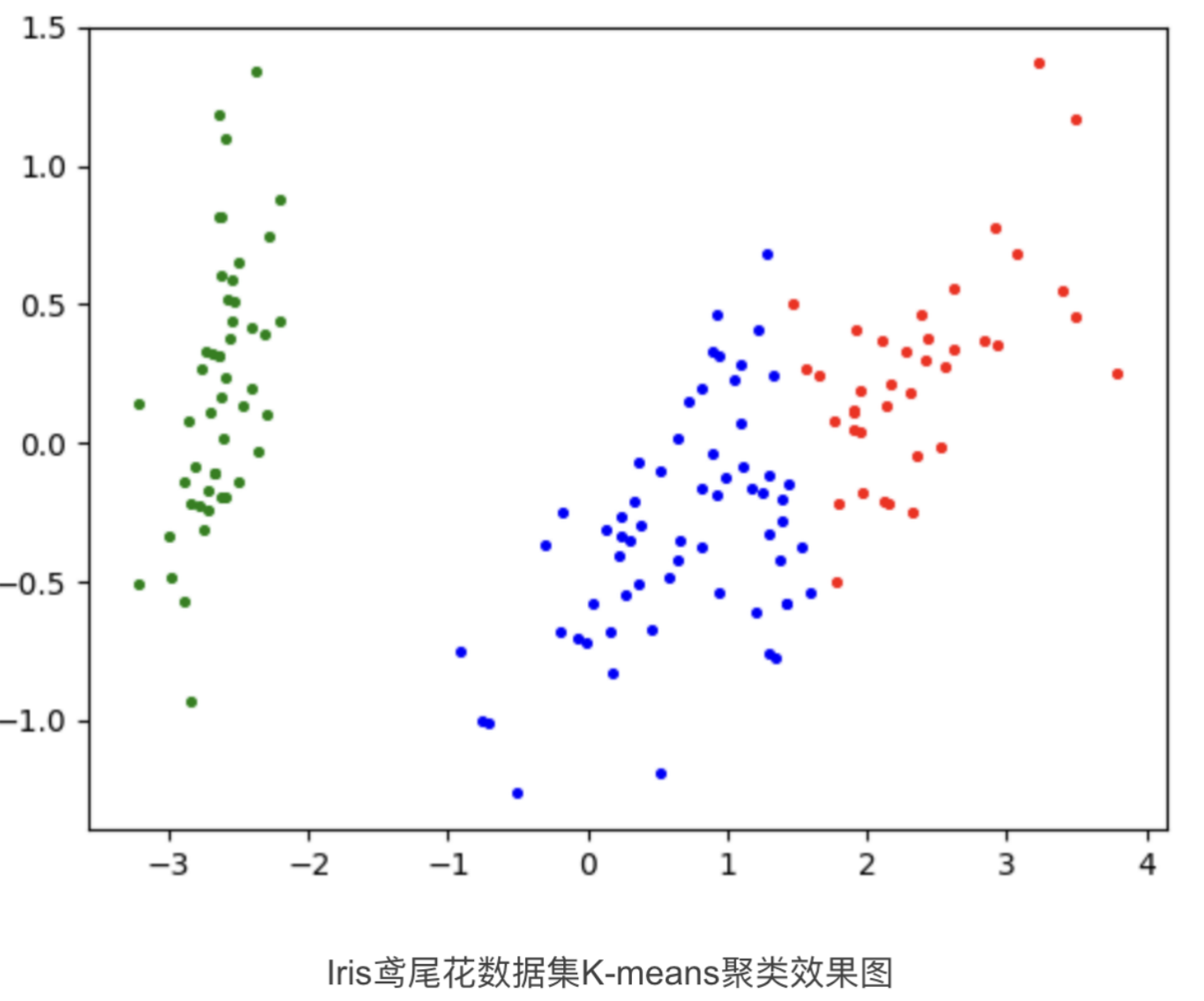
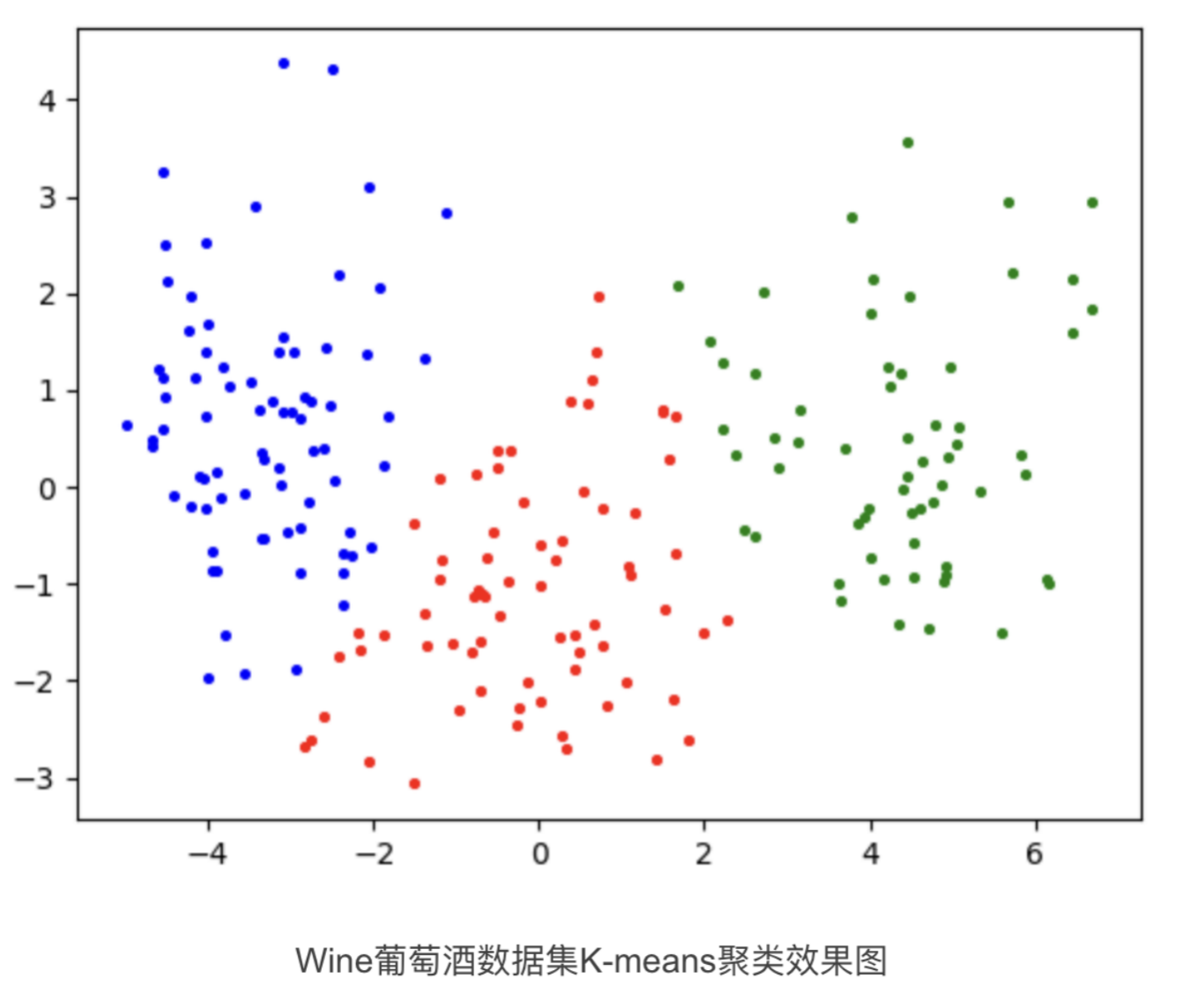
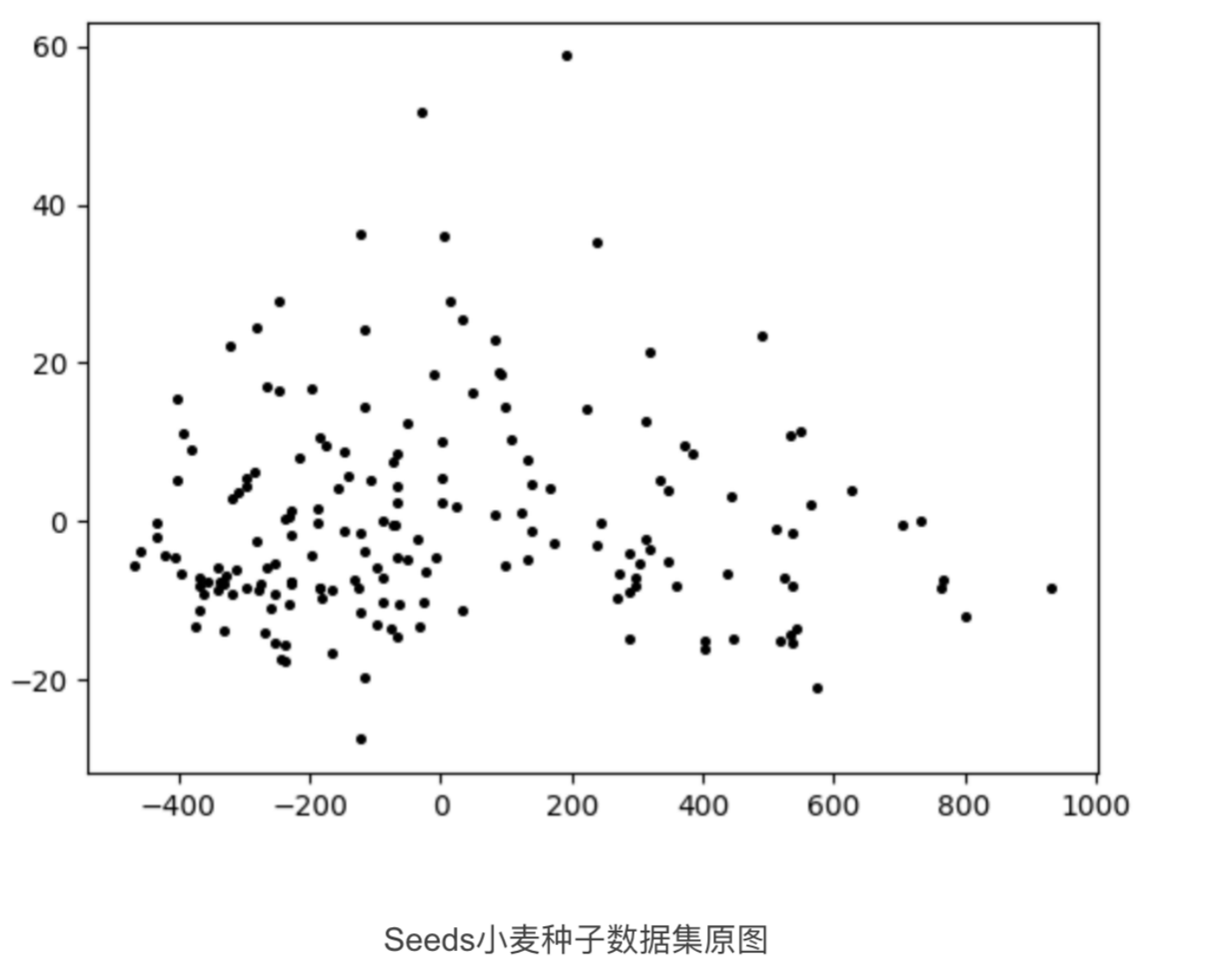
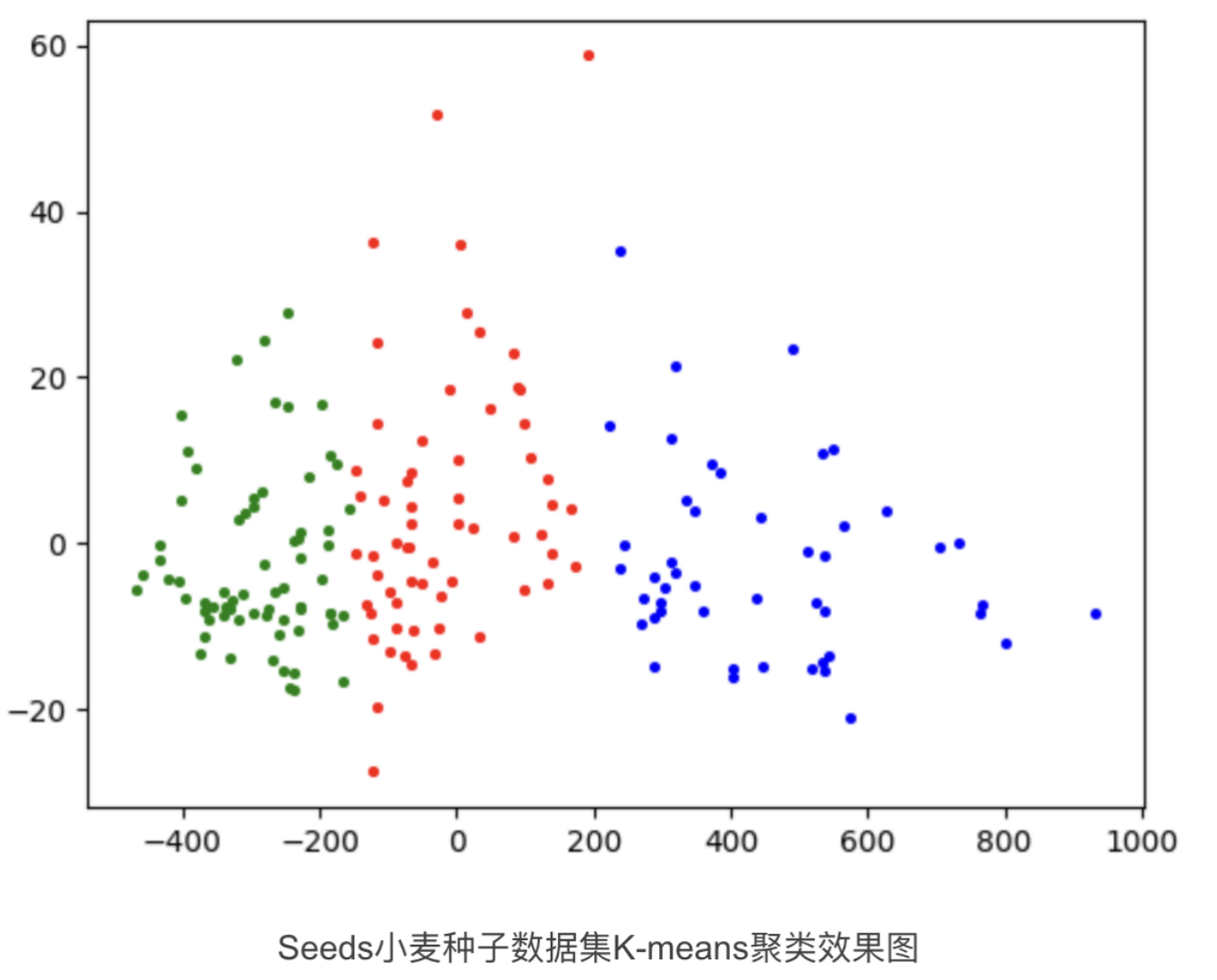
K-means优缺点总结:
(1) K-means优点:
① K-means算法收敛速度快,时间复杂度低,计算效率较高;
② K-means算法算法可解释性好,原理简单易于理解;
③ K-means算法调参简单,需要调的参数只有聚类簇数K。
(2) K-means缺点:
① K-means算法的参数簇数目(K值)需要手动指定,K值的确定直接影响聚类的结果,通常情况下,K值需要经验和对数据集的理解指定,因此指定的数值未必理想,聚类的结果也就无从保证。
② K-means算法对噪声和异常值敏感,聚类结果容易受到噪声点的影响;
③ K-means算法欧氏距离进行相似性度量,适用于簇是凸形或球形的数据集,在非凸形数据集中难以达到良好的聚类效果。;
④ K-means算法的初始中心点选取上采用的是随机的方法。K-means算法极为依赖初始中心点的选取:一旦错误地选取了初始中心点,对于后续的聚类过程影响极大,很可能得不到最理想的聚类结果,同时聚类迭代的次数也可能会增加。而随机选取的初始中心点具有很大的不确定性,也直接影响着聚类的效果;
⑤ K-means算法结果不一定是全局最优,只能保证局部最优。

本文来自博客园,作者:limingqi,转载请注明原文链接:https://www.cnblogs.com/limingqi/p/18986344



 浙公网安备 33010602011771号
浙公网安备 33010602011771号