sed 取行,删行,新增行,替换字符串 ;grep 取匹配行
nl /etc/passwd |sed '2,5 d' 删除文件2-5行
nl命令 输出的文件内容自动加上行号
sed [-nefr] [动作] 以行为单位
注意: 在-i 时,mac 与linux稍有不同,需要加空格,如 sed -i '' '/CMDCB0003112/d' 467gvcf.list.deleteNA #否则报错sed: 1: "467gvcf.list.deleteNA": extra characters at the end of g command # sed -i '' 这里是两个单引号括起来空格的意思
1.
-n 仅显示script 处理后的结果
-e 在命令行模式进行sed的动作编辑
-f 后接script文件名
-r 支持扩展式正则表达式
-i 直接修改文件内容,不打印输出
2.动作
[n1[[2]]function sed后用单引号括起
n1 n2表行号
function参数
2.1以行为单位 删除新增
a: 新增字符串为新的一行出现在下一行 sed'2a drink tar'第二行后面
d: 删除
2.2 行为单位,替换、显示
整行替换: nl /etc/passwd |sed ‘2,5c No 2-5 number’
c:后接字符串替换 n1,n2之间的行
显示整行: nl /etc/passwd |sed -n '5,7p' 直接取处5-7行打印
-n不重复输出
p:将sed-n 数据打印出来
2.3替换字符串:sed's/要被替换的字符串/新的字符串/g'
s:替换。搭配正则表达式
如: /sbin/ifconfig ech0 |grep 'inet addr' | sed 's/^.*addr://g'
取出一个字符穿的前50个字符 cat your.fa | grep -v ">" | sed -r 's/(.{50}).*/\1/g
zcat All_20170710.vcf.gz | sed -E '/^[^#]/{s/^([^#]+)/chr\1/; s/^chrMT/chrM/}' > All_20170710.fixed.vcf # b37-hg19
sed 取特定行
sed -n ‘1,2p’ infile #print line 1 to 2
sed -n '2,$p' file ##print line 2 to end of line
sed '/pattern/!p' infile ## 匹配pattern的行不输出
sed -n -e '/build/p' /etc/cli.cfg p-打印行 n - e --e<script>或--expression=<script>:以选项中的指定的script来处理输入的文本文件
sed 替换:
sed -i "s/ /\t/g" COVID19-284.301.all.Severity_binary.ALL_1.SAIGE.vcf.genotype.txt.tst # 把文件中空格替换为\t
注:其他方式取第二行-末尾的方法
1. cat filename | tail -n +2 即tail -n +K # use -n +K to output lines starting with the Kth
2.awk '{if(NR!=1) print }' filename
sed
sed 取特定行
sed -n ‘1,2p’ infile #print line 1 to 2
sed -n '2,$p' file ##print line 2 to end of line
sed -n '/pattern/p' infile ## 只输出匹配pattern的行
匹配多个字符串:
|前需要用转义符号 如 sed -n '/SNP\|INDEL/p' outdir_summary_metrics/concordance.402_58.Y21030000454415_1540.genotype_concordance_summary_metrics
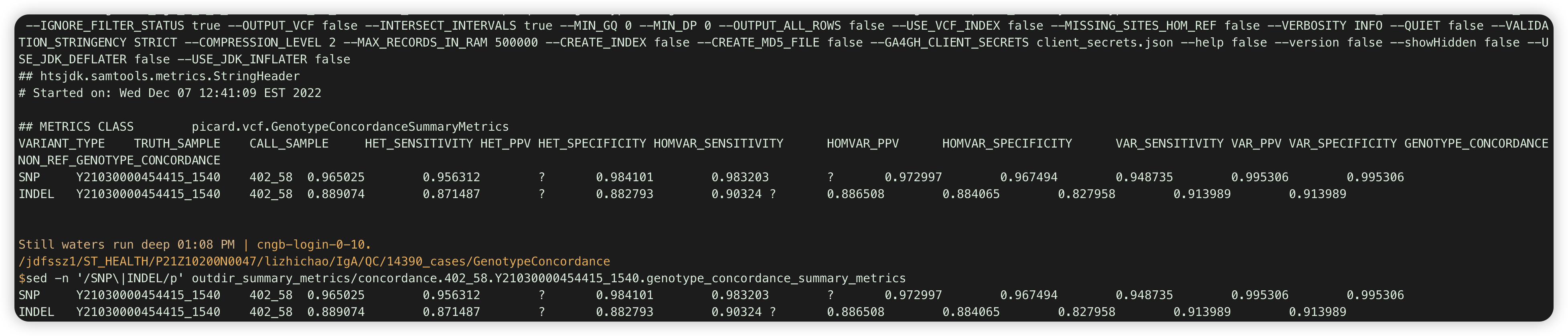
sed '/pattern/d' #删除包含pattern的行,其余都显示
sed -n -e '/build/p' /etc/cli.cfg p-打印行 n - e --e<script>或--expression=<script>:以选项中的指定的script来处理输入的文本文件
sed 替换:
sed -i "s/ /\t/g" COVID19-284.301.all.Severity_binary.ALL_1.SAIGE.vcf.genotype.txt.tst # 把文件中空格替换为\t
注:其他方式取第二行-末尾的方法
1. cat filename | tail -n +2 即tail -n +K # use -n +K to output lines starting with the Kth
2.awk '{if(NR!=1) print }' filename
nl /etc/passwd |sed '2,5 d' 删除文件2-5行
#nl命令 输出的文件内容自动加上行号
- 删除新增行
a: 新增字符串为新的一行出现在下一行 sed'2a drink tar'第二行后面
新增一行在第一行前
sed -i '1i\要添加的内容' yourfile 。如 sed -i '1i\sample cov_depth chrX_cov chrY_cov normalized_depth_chrX normalized_depth_chrY' bamsex_496
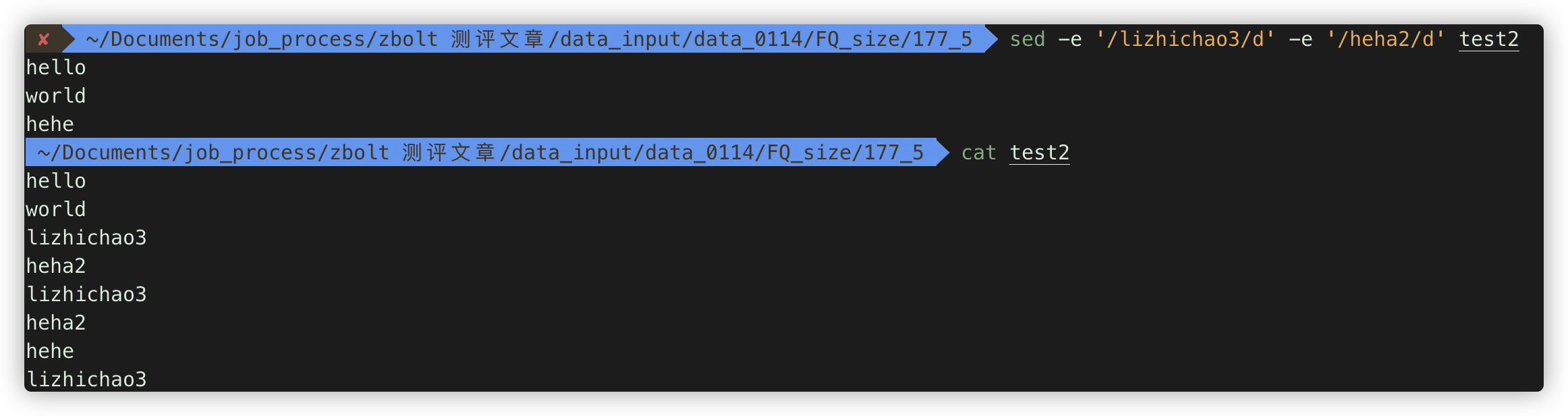
d: 删除 .注:-e 可以多个sed操作如sed -e '/lizhichao3/d' -e '/heha2/d' test2
-
行为单位,替换、显示 整行替换: nl /etc/passwd |sed '2,5c No 2-5 number’ 注:c:后接字符串替换 n1,n2之间的行
显示整行: nl /etc/passwd |sed -n '5,7p' 直接取处5-7行打印
-n不重复输出
p:将sed-n 数据打印出来注:nl 输出文件的每一行前加行号
- 替换字符串:sed 's/要被替换的字符串/新的字符串/g'
s:替换,可以搭配正则表达式
如: /sbin/ifconfig ech0 |grep 'inet addr' | sed 's/^.*addr://g'
例1取出一个字符串的前50个字符 cat your.fa | grep -v ">" | sed -r 's/(.{50}).*/\1/g
例2.去掉sRNA 的fasta中N序列。 sed 'N;s/\n/\t/'sRNA.fa|sed -e '/\t.*N/d'|tr"\t""\n"
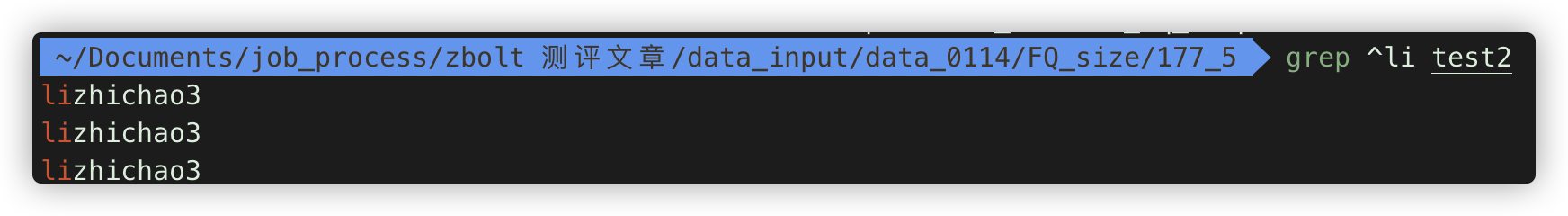
grep 适用正则 如grep ^li test2
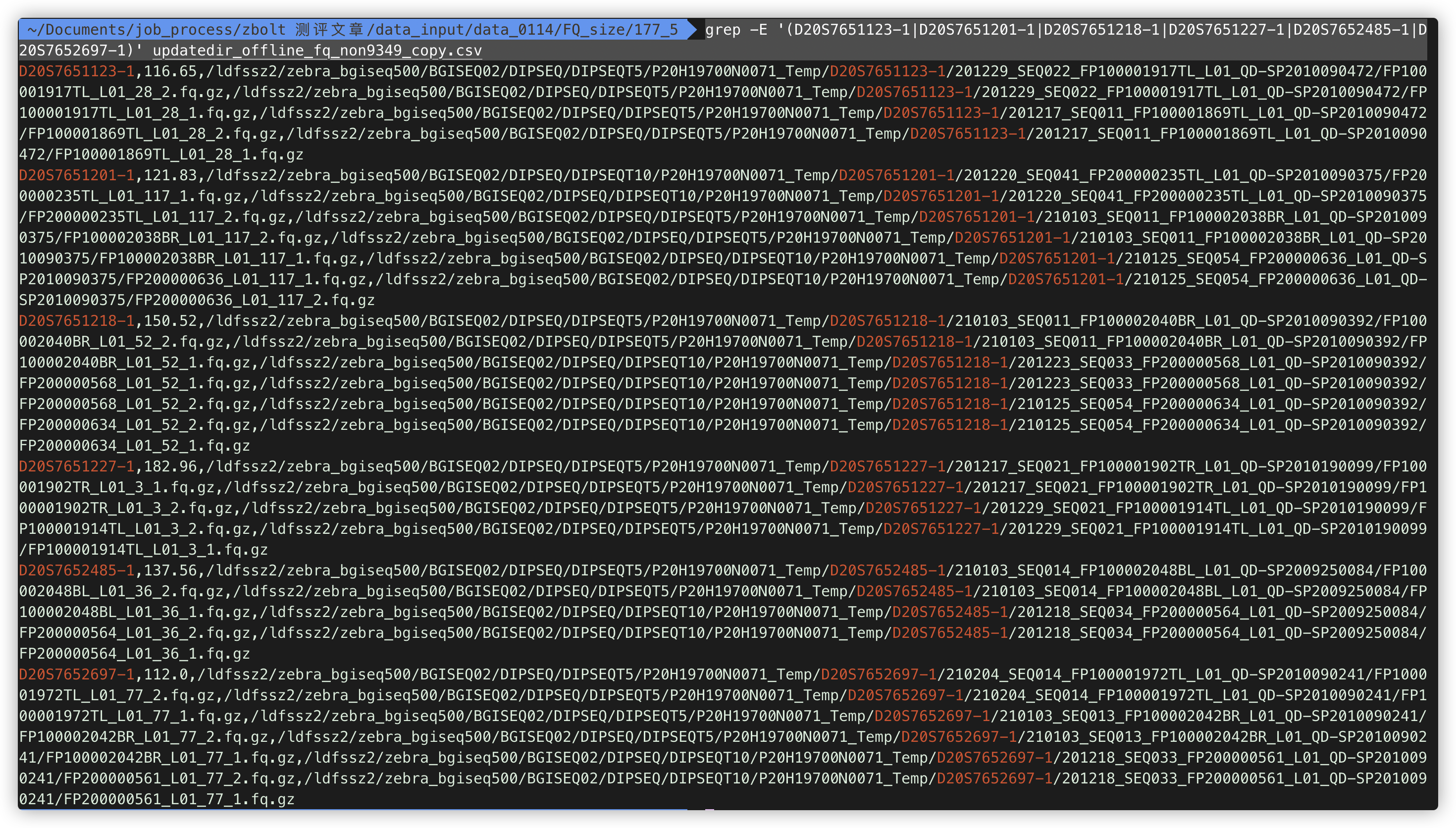
取多个匹配行可以:grep -E '(pattern1|pattern2|...)' $file 如grep -E '(D20S7651123-1|D20S7651201-1|D20S7651218-1|D20S7651227-1|D20S7652485-1|D20S7652697-1)' updatedir_offline_fq_non9349_copy.csv
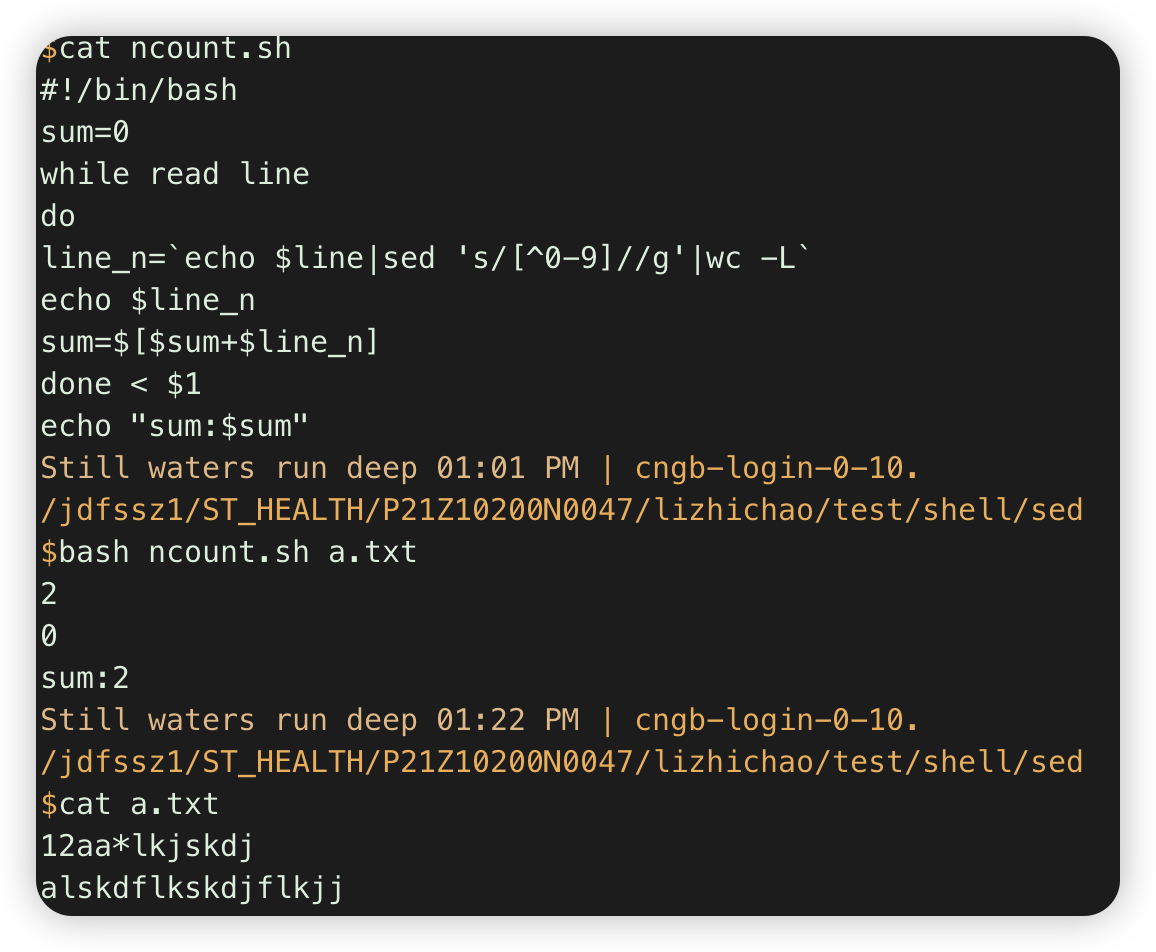
以下脚本计算每一行的数字出现的次数,以及总次数
#!/bin/bash sum=0 while read line do line_n=`echo $line|sed 's/[^0-9]//g'|wc -L` echo $line_n sum=$[$sum+$line_n] done < $1 echo "sum:$sum"
说明: sed 's/[^0-9]//g' 把非数字替换为空。
wc -L 计算文件最长行的长度
$[$sum+$line_n] linux的计算表达式
while read line;do;done<$1 从传递参数$1,读取内容。
运行记录:
sed 搭配awk 取文件中匹配行的特定列 。 cat [file]|sed xxx|awk xxx 。 搭配写个for循环遍历多个文件,取出所有列表文件内匹配行的指定列
cat outdir_summary_metrics/concordance.402_58.Y21030000454415_1540.genotype_concordance_summary_metrics|sed -n '/VARIANT_TYPE/p'|awk '{print $1,$2,$3,$(NF-1) }'
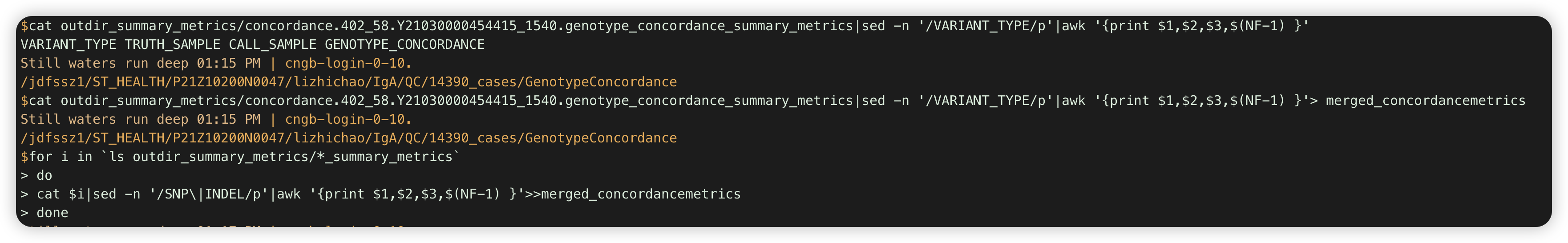
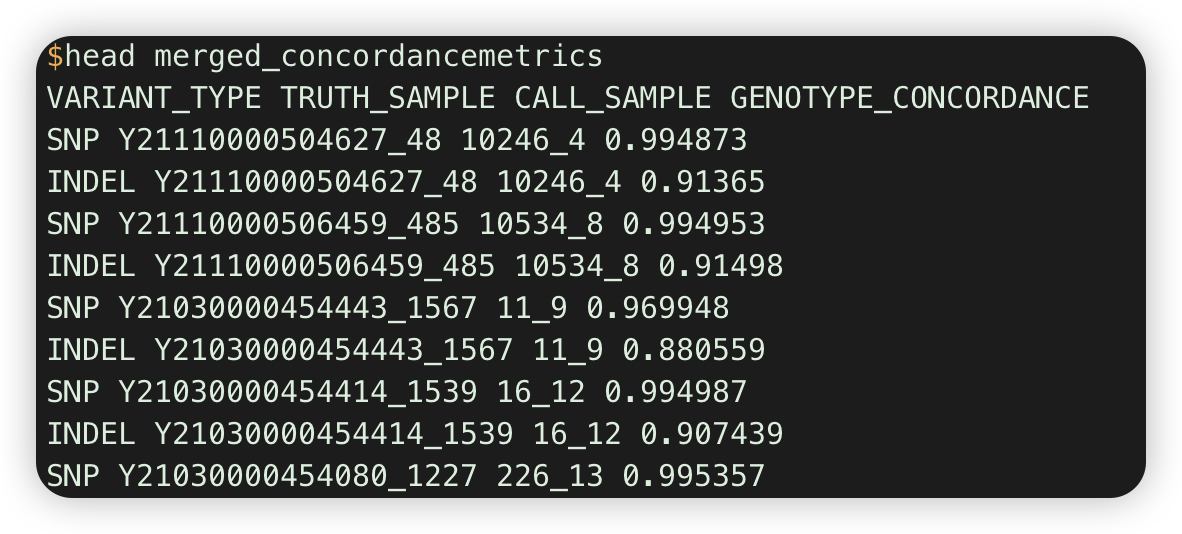
注:不可以sed xxx [file]|awk,会报sed: can't read |awk: No such file or directory
本文来自博客园,作者:BioinformaticsMaster,转载请注明原文链接:https://www.cnblogs.com/koujiaodahan/p/13636761.html
posted on 2020-09-09 04:12 BioinformaticsMaster 阅读(1088) 评论(0) 收藏 举报




 浙公网安备 33010602011771号
浙公网安备 33010602011771号