RANSAC算法在图像拼接上的应用的实现
关于算法原理请参考《基于SURF特征的图像与视频拼接技术的研究》。



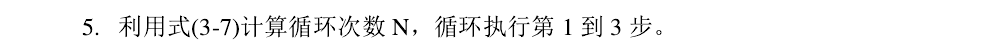


一、问题提出
RANSAC的算法原理并不复杂,比较复杂的地方在于“建立模型”和“评价模型”。我们经常看到的是采用“直线”或者“圆”作为基本模型进行“建立”,而采用所有点到该“直线”或“圆”的欧拉距离作为标准来“评价”(当然是越小越好)。在经典的图像拼接算法中,需要对特征点进行配对。采用的模型简单来说为“根据4对特征点计算出单应矩阵,而后计算图1上所有对应的特征点经过这个‘
单应矩阵’
变化后得到的图片和图2上的距离之和“(当然也是越小越好)。
为了提高识别的效率,前辈对算法进行了不懈的研究和提升,目前看来,用于图像拼接的RANSAC算法应该如下:
及其改进算法:
二、算法实现
a)数据准备
某大学图片,很明显的有视场变化
b)算法分析,参考《
基于SURF特征的图像与视频拼接技术的研究和实现一 和 二
》
现在思考,RANSAC算法其实是”基于统计的配对算法“,在进入RANSAC算法流程之前,已经计算出来图1和图2上的特征点值了。我们不仅需要根据这些点值去预测模型,而且需要去检测模型。这个模型也不是凭空随便找的,而是以”透视变换“作为基础的(关于透视变化请参考我前面的博文)。
寻找的方法是首先找到符合某一模型的”内点集“,而后根据这一”内点集“,创建变换模型。
寻找”内点集“的方法就是直接从现有的数据中找出一部分数据计算出一个模型,而后根据这个模型计算所有点的误差,迭代多次,得到最小误差的情况(和对应的点集),这个时候的模型就是接近正确的模型。
这个误差的计算方法也是设计出来的(很可能还是统计值)。
所以RANSAC很像是基于统计的一种计算可行解的模式。很多时候你不是需要从很多的数据中找出一个模型来吗?比如马尔萨斯模型?这个模型可能有函数,还有参数。你猜测的是函数,但是参数就可以通过这种模式来进行计算。
如果有比较好的评价函数,
最后你还可以比较几种函数的选择。所以RANSAC就是一种单模型下基于离散数据计算模型的方法。(其实也是直观的、基础的、简洁的、有效的)
这样我想起之前研究过的一种叫做”交叉检验“(cross check /cross validation)的东西。
定义:在给定的建模样本中,拿出大部分样本进行模型建立,留小部分对建立的模型进行预报,并将这小部分进行误差预报,计算平方加和。(然后当然是选取误差最小的模型和)
相比较RANSAC和CROSS VALIDATION,有两点不同。一个是模型的建立,RANSAC是选择很少量的数据建立模型(比如圆、线、透视变换),而后大量数据做验证;而CROSS需要较多的数据建立模型(比如MLP,神网),较少的数据进行验证(它也只有较少的数据了)
c)解析
OPENCV中的实现
为了实现图像的特征点的匹配,并且最后实现图像拼接,在OPENCV中实现了RANSAC算法及其改进算法
c.1 调用方法
//-- Step 3: 匹配
FlannBasedMatcher matcher;//BFMatcher为强制匹配
std::vector< DMatch > matches;
matcher.match( descriptors_1, descriptors_2, matches );
//取最大最小距离
double max_dist = 0; double min_dist = 100;
for( int i = 0; i < descriptors_1.rows; i++ )
{
double dist = matches[i].distance;
if( dist < min_dist ) min_dist = dist;
if( dist > max_dist ) max_dist = dist;
}
std::vector< DMatch > good_matches;
for( int i = 0; i < descriptors_1.rows; i++ )
{
if( matches[i].distance <= 3*min_dist )//这里的阈值选择了3倍的min_dist
{
good_matches.push_back( matches[i]);
}
}
//画出"good match"
Mat img_matches;
drawMatches( img_1, keypoints_1, img_2, keypoints_2,
good_matches, img_matches, Scalar::all(-1), Scalar::all(-1),
vector<char>(), DrawMatchesFlags::NOT_DRAW_SINGLE_POINTS );
//-- Localize the object from img_1 in img_2
std::vector<Point2f> obj;
std::vector<Point2f> scene;
for( int i = 0; i < (int)good_matches.size(); i++ )
{
obj.push_back( keypoints_1[ good_matches[i].queryIdx ].pt );
scene.push_back( keypoints_2[ good_matches[i].trainIdx ].pt );
}
//直接调用ransac,计算单应矩阵
Mat H = findHomography( obj, scene, CV_RANSAC );
c.2 原理分析
opencv中已经封装的很好了,注意的是在实际使用中还可以对识别出来的结果做进一步的处理。
findHomography
的定义
参数分为points1和points2,参数3是method,然后是threshold(一般为3),最后为mask
cv
:
:Mat cv
:
:findHomography( InputArray _points1, InputArray _points2,
int method,
double ransacReprojThreshold, OutputArray _mask )
{
const
double confidence
=
0.
995;
const
int maxIters
=
2000;
const
double defaultRANSACReprojThreshold
=
3;
bool result
=
false;
Mat points1
= _points1.getMat(), points2
= _points2.getMat();
Mat src, dst, H, tempMask;
int npoints
=
-
1;
for(
int i
=
1; i
<
=
2; i
++ )
{
Mat
& p
= i
==
1
? points1
: points2;
Mat
& m
= i
==
1
? src
: dst;
npoints
= p.checkVector(
2,
-
1,
false);
if( npoints
<
0 )
{
npoints
= p.checkVector(
3,
-
1,
false);
if( npoints
<
0 )
CV_Error(Error
:
:StsBadArg,
"The input arrays should be 2D or 3D point sets");
if( npoints
==
0 )
return Mat();
convertPointsFromHomogeneous(p, p);
}
p.reshape(
2, npoints).convertTo(m, CV_32F);
}
CV_Assert( src.checkVector(
2)
== dst.checkVector(
2) );
if( ransacReprojThreshold
<
=
0 )
ransacReprojThreshold
= defaultRANSACReprojThreshold;
Ptr
<PointSetRegistrator
:
:Callback
> cb
= makePtr
<HomographyEstimatorCallback
>();
if( method
==
0
|| npoints
==
4 )
{
tempMask
= Mat
:
:ones(npoints,
1, CV_8U);
result
= cb
-
>runKernel(src, dst, H)
>
0;
}
else
if( method
== RANSAC )
result
= createRANSACPointSetRegistrator(cb,
4, ransacReprojThreshold, confidence, maxIters)
-
>run(src, dst, H, tempMask);
else
if( method
== LMEDS )
result
= createLMeDSPointSetRegistrator(cb,
4, confidence, maxIters)
-
>run(src, dst, H, tempMask);
else
CV_Error(Error
:
:StsBadArg,
"Unknown estimation method");
if( result
&& npoints
>
4 )
{
compressPoints( src.ptr
<Point2f
>(), tempMask.ptr
<uchar
>(),
1, npoints );
npoints
= compressPoints( dst.ptr
<Point2f
>(), tempMask.ptr
<uchar
>(),
1, npoints );
if( npoints
>
0 )
{
Mat src1
= src.rowRange(
0, npoints);
Mat dst1
= dst.rowRange(
0, npoints);
src
= src1;
dst
= dst1;
if( method
== RANSAC
|| method
== LMEDS )
cb
-
>runKernel( src, dst, H );
Mat H8(
8,
1, CV_64F, H.ptr
<
double
>());
createLMSolver(makePtr
<HomographyRefineCallback
>(src, dst),
10)
-
>run(H8);
}
}
if( result )
{
if( _mask.needed() )
tempMask.copyTo(_mask);
}
else
H.release();
return H;
}
和RANSAC相关的是
Ptr
<PointSetRegistrator
> createRANSACPointSetRegistrator(
const Ptr
<PointSetRegistrator
:
:Callback
>
& _cb,
int _modelPoints,
double _threshold,
double _confidence,
int _maxIters)
{
CV_Assert(
!RANSACPointSetRegistrator_info_auto.name().empty() );
return Ptr
<PointSetRegistrator
>(
new RANSACPointSetRegistrator(_cb, _modelPoints, _threshold, _confidence, _maxIters));
}
class
RANSACPointSetRegistrator
:
public
PointSetRegistrator
{
public
:
RANSACPointSetRegistrator(
const Ptr
<PointSetRegistrator
:
:Callback
>
& _cb
=Ptr
<PointSetRegistrator
:
:Callback
>(),
int _modelPoints
=
0,
double _threshold
=
0,
double _confidence
=
0.
99,
int _maxIters
=
1000)
: cb(_cb), modelPoints(_modelPoints), threshold(_threshold), confidence(_confidence), maxIters(_maxIters)
{
checkPartialSubsets
=
true;
}
int findInliers(
const Mat
& m1,
const Mat
& m2,
const Mat
& model, Mat
& err, Mat
& mask,
double thresh )
const
{
cb
-
>computeError( m1, m2, model, err );
mask.create(err.size(), CV_8U);
CV_Assert( err.isContinuous()
&& err.type()
== CV_32F
&& mask.isContinuous()
&& mask.type()
== CV_8U);
const
float
* errptr
= err.ptr
<
float
>();
uchar
* maskptr
= mask.ptr
<uchar
>();
float t
= (
float)(thresh
*thresh);
int i, n
= (
int)err.total(), nz
=
0;
for( i
=
0; i
< n; i
++ )
{
int f
= errptr[i]
<
= t;
maskptr[i]
= (uchar)f;
nz
+= f;
}
return nz;
}
bool getSubset(
const Mat
& m1,
const Mat
& m2,
Mat
& ms1, Mat
& ms2, RNG
& rng,
int maxAttempts
=
1000 )
const
{
cv
:
:AutoBuffer
<
int
> _idx(modelPoints);
int
* idx
= _idx;
int i
=
0, j, k, iters
=
0;
int esz1
= (
int)m1.elemSize(), esz2
= (
int)m2.elemSize();
int d1
= m1.channels()
>
1
? m1.channels()
: m1.cols;
int d2
= m2.channels()
>
1
? m2.channels()
: m2.cols;
int count
= m1.checkVector(d1), count2
= m2.checkVector(d2);
const
int
*m1ptr
= m1.ptr
<
int
>(),
*m2ptr
= m2.ptr
<
int
>();
ms1.create(modelPoints,
1, CV_MAKETYPE(m1.depth(), d1));
ms2.create(modelPoints,
1, CV_MAKETYPE(m2.depth(), d2));
int
*ms1ptr
= ms1.ptr
<
int
>(),
*ms2ptr
= ms2.ptr
<
int
>();
CV_Assert( count
>
= modelPoints
&& count
== count2 );
CV_Assert( (esz1
%
sizeof(
int))
==
0
&& (esz2
%
sizeof(
int))
==
0 );
esz1
/=
sizeof(
int);
esz2
/=
sizeof(
int);
for(; iters
< maxAttempts; iters
++)
{
for( i
=
0; i
< modelPoints
&& iters
< maxAttempts; )
{
int idx_i
=
0;
for(;;)
{
idx_i
= idx[i]
= rng.uniform(
0, count);
for( j
=
0; j
< i; j
++ )
if( idx_i
== idx[j] )
break;
if( j
== i )
break;
}
for( k
=
0; k
< esz1; k
++ )
ms1ptr[i
*esz1
+ k]
= m1ptr[idx_i
*esz1
+ k];
for( k
=
0; k
< esz2; k
++ )
ms2ptr[i
*esz2
+ k]
= m2ptr[idx_i
*esz2
+ k];
if( checkPartialSubsets
&&
!cb
-
>checkSubset( ms1, ms2, i
+
1 ))
{
iters
++;
continue;
}
i
++;
}
if(
!checkPartialSubsets
&& i
== modelPoints
&&
!cb
-
>checkSubset(ms1, ms2, i))
continue;
break;
}
return i
== modelPoints
&& iters
< maxAttempts;
}
bool run(InputArray _m1, InputArray _m2, OutputArray _model, OutputArray _mask)
const
{
bool result
=
false;
Mat m1
= _m1.getMat(), m2
= _m2.getMat();
Mat err, mask, model, bestModel, ms1, ms2;
int iter, niters
= MAX(maxIters,
1);
int d1
= m1.channels()
>
1
? m1.channels()
: m1.cols;
int d2
= m2.channels()
>
1
? m2.channels()
: m2.cols;
int count
= m1.checkVector(d1), count2
= m2.checkVector(d2), maxGoodCount
=
0;
RNG rng((uint64)
-
1);
CV_Assert( cb );
CV_Assert( confidence
>
0
&& confidence
<
1 );
CV_Assert( count
>
=
0
&& count2
== count );
if( count
< modelPoints )
return
false;
Mat bestMask0, bestMask;
if( _mask.needed() )
{
_mask.create(count,
1, CV_8U,
-
1,
true);
bestMask0
= bestMask
= _mask.getMat();
CV_Assert( (bestMask.cols
==
1
|| bestMask.rows
==
1)
&& (
int)bestMask.total()
== count );
}
else
{
bestMask.create(count,
1, CV_8U);
bestMask0
= bestMask;
}
if( count
== modelPoints )
{
if( cb
-
>runKernel(m1, m2, bestModel)
<
=
0 )
return
false;
bestModel.copyTo(_model);
bestMask.setTo(Scalar
:
:all(
1));
return
true;
}
for( iter
=
0; iter
< niters; iter
++ )
{
int i, goodCount, nmodels;
if( count
> modelPoints )
{
bool found
= getSubset( m1, m2, ms1, ms2, rng );
if(
!found )
{
if( iter
==
0 )
return
false;
break;
}
}
nmodels
= cb
-
>runKernel( ms1, ms2, model );
if( nmodels
<
=
0 )
continue;
CV_Assert( model.rows
% nmodels
==
0 );
Size modelSize(model.cols, model.rows
/nmodels);
for( i
=
0; i
< nmodels; i
++ )
{
Mat model_i
= model.rowRange( i
*modelSize.height, (i
+
1)
*modelSize.height );
goodCount
= findInliers( m1, m2, model_i, err, mask, threshold );
if( goodCount
> MAX(maxGoodCount, modelPoints
-
1) )
{
std
:
:swap(mask, bestMask);
model_i.copyTo(bestModel);
maxGoodCount
= goodCount;
niters
= RANSACUpdateNumIters( confidence, (
double)(count
- goodCount)
/count, modelPoints, niters );
}
}
}
if( maxGoodCount
>
0 )
{
if( bestMask.data
!= bestMask0.data )
{
if( bestMask.size()
== bestMask0.size() )
bestMask.copyTo(bestMask0);
else
transpose(bestMask, bestMask0);
}
bestModel.copyTo(_model);
result
=
true;
}
else
_model.release();
return result;
}
void setCallback(
const Ptr
<PointSetRegistrator
:
:Callback
>
& _cb) { cb
= _cb; }
AlgorithmInfo
* info()
const;
Ptr
<PointSetRegistrator
:
:Callback
> cb;
int modelPoints;
bool checkPartialSubsets;
double threshold;
double confidence;
int maxIters;
};
d)如何复用OPENCV中的实现于数据的统计研究
opencv中的封装是专门用于特征点的,如果需要使用在其它地方还需要修改。但是我更关心的是RANSAC的算法应该用在哪里?
四、反思小结
即使是这样一个原理来说比较清楚的算法,如果想从零开始进行实现,还是很不容易的。所以积累算法资源、提高算法实现能力,可能都是很重要的事情。
和cross validation算法比较,
和lmeds 最小平方中值估计法




 浙公网安备 33010602011771号
浙公网安备 33010602011771号