pytorch深度学习实战:自定义数据集类型
第10章 读书笔记:将数据源组合成统一的数据集
本章核心目标
本章的核心任务是实现数据加载(Data Loading)——将原始的CT扫描数据和人工标注信息转换为PyTorch可以使用的训练样本。
原始数据 (.mhd/.raw文件 + CSV标注)
↓
Python数据结构 (Ct类, CandidateInfoTuple)
↓
PyTorch Dataset (LunaDataset)
↓
训练样本元组 (candidate_t, pos_t, series_uid, center_irc)
一、原始数据结构
1.1 CT扫描文件
- 格式:MetaIO格式(由DICOM转换而来)
- 组成:每个CT扫描由两个文件构成
.mhd文件:元数据头信息(体素大小、坐标原点、方向矩阵等).raw文件:三维数组的原始体素数据
- 命名:以系列UID(series_uid)命名,如
1.2.3.mhd和1.2.3.raw
1.2 标注CSV文件
| 文件名 | 内容 | 行数 |
|---|---|---|
candidates.csv |
所有候选肿块的位置和结节状态 | ~551,000行 |
annotations.csv |
确认为结节的候选者的直径信息 | ~1,200行 |
candidates.csv 格式:
seriesuid, coordX, coordY, coordZ, class
1.3...6860, -56.08, -67.85, -311.92, 0 # class=0 非结节
1.3...6860, 53.21, -244.41, -245.17, 1 # class=1 结节
annotations.csv 格式:
seriesuid, coordX, coordY, coordZ, diameter_mm
1.3.6...6860, -128.69, -175.31, -298.38, 5.65
⚠️ 注意:两个文件的坐标可能不完全对齐,需要模糊匹配!
二、数据预处理流程
2.1 统一标注数据:getCandidateInfoList() 函数
目的:合并candidates.csv和annotations.csv的信息,生成统一的候选者列表。
from collections import namedtuple
# 定义命名元组存储候选者信息
CandidateInfoTuple = namedtuple(
'CandidateInfoTuple',
'isNodule_bool, diameter_mm, series_uid, center_xyz',
)
@functools.lru_cache(1) # 内存缓存,避免重复解析
def getCandidateInfoList(requireOnDisk_bool=True):
# 1. 获取磁盘上存在的CT文件列表
mhd_list = glob.glob(os.path.join(data_dir, 'subset*/*.mhd'))
presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list}
# 2. 解析annotations.csv,按series_uid分组存储直径信息
diameter_dict = {}
with open(os.path.join(data_dir, 'annotations.csv'), "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0]
annotationCenter_xyz = tuple([float(x) for x in row[1:4]])
annotationDiameter_mm = float(row[4])
diameter_dict.setdefault(series_uid, []).append(
(annotationCenter_xyz, annotationDiameter_mm)
)
# 3. 解析candidates.csv,并与annotations进行模糊匹配
candidateInfo_list = []
with open(os.path.join(data_dir, 'candidates.csv'), "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0]
# 过滤不在磁盘上的数据
if series_uid not in presentOnDisk_set and requireOnDisk_bool:
continue
# 载入candidates.csv数据
isNodule_bool = bool(int(row[4]))
candidateCenter_xyz = tuple([float(x) for x in row[1:4]])
# 模糊匹配:查找距离足够近的标注
candidateDiameter_mm = 0.0
for annotation_tup in diameter_dict.get(series_uid, []):
annotationCenter_xyz, annotationDiameter_mm = annotation_tup
for i in range(3):
delta_mm = abs(candidateCenter_xyz[i] - annotationCenter_xyz[i])
if delta_mm > annotationDiameter_mm / 4:
break
else: # for-else: 如果循环没有break
candidateDiameter_mm = annotationDiameter_mm
break
candidateInfo_list.append(CandidateInfoTuple(
isNodule_bool,
candidateDiameter_mm,
series_uid,
candidateCenter_xyz,
))
# 4. 排序:确保结节在前,便于划分训练/验证集
candidateInfo_list.sort(reverse=True)
return candidateInfo_list
设计要点:
- 使用
@functools.lru_cache(1)进行内存缓存 requireOnDisk_bool参数支持在部分数据上运行- 排序确保训练/验证集都能获得代表性样本
三、Ct类:加载单个CT扫描
3.1 类结构
import SimpleITK as sitk
class Ct:
def __init__(self, series_uid):
# 1. 查找并加载.mhd文件
mhd_path = glob.glob(
os.path.join(data_dir, 'subset*/{}.mhd'.format(series_uid))
)[0]
# 2. 转换为NumPy数组
ct_mhd = sitk.ReadImage(mhd_path)
ct_a = np.array(sitk.GetArrayFromImage(ct_mhd), dtype=np.float32) # 整个Ct数据
# 3. 数据清洗:裁剪亨氏单位(HU)值到合理范围
ct_a.clip(-1000, 1000, ct_a)
# 4. 保存数据和元数据
self.series_uid = series_uid
self.hu_a = ct_a
self.origin_xyz = XyzTuple(*ct_mhd.GetOrigin())
self.vxSize_xyz = XyzTuple(*ct_mhd.GetSpacing())
self.direction_a = np.array(ct_mhd.GetDirection()).reshape(3, 3)
def getRawCandidate(self, center_xyz, width_irc):
"""从CT中裁剪出候选区域"""
# 坐标转换:病人坐标(XYZ) → 数组索引(IRC)
center_irc = xyz2irc(
center_xyz,
self.origin_xyz,
self.vxSize_xyz,
self.direction_a,
)
# 计算裁剪范围
slice_list = []
for axis, center_val in enumerate(center_irc):
# patch的起点和终点
start_ndx = int(round(center_val - width_irc[axis]/2))
end_ndx = int(start_ndx + width_irc[axis])
assert center_val >= 0 and center_val < self.hu_a.shape[axis], repr([self.series_uid, center_xyz, self.origin_xyz, self.vxSize_xyz, center_irc, axis])
# 处理边界patch
if start_ndx < 0:
# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(
# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))
start_ndx = 0
end_ndx = int(width_irc[axis])
if end_ndx > self.hu_a.shape[axis]:
# log.warning("Crop outside of CT array: {} {}, center:{} shape:{} width:{}".format(
# self.series_uid, center_xyz, center_irc, self.hu_a.shape, width_irc))
end_ndx = self.hu_a.shape[axis]
start_ndx = int(self.hu_a.shape[axis] - width_irc[axis])
# 创建切片对象
slice_list.append(slice(start_ndx, end_ndx))
ct_chunk = self.hu_a[tuple(slice_list)]
return ct_chunk, center_irc # 小切块, 中心坐标
亨氏单位
3.2 亨氏单位(Hounsfield Unit, HU)
| 物质 | HU值 | 密度 (g/cm³) |
|---|---|---|
| 空气 | -1000 | ~0 |
| 水 | 0 | 1 |
| 肿瘤 | ~0 | ~1 |
| 骨骼 | ≥1000 | 2-3 |
裁剪原因:
- 低于-1000的值表示CT扫描仪视野外,应视为空气
- 高于1000的值(骨骼、金属植入物)与肿瘤检测无关
- 保持数据范围在[-1000, 1000]
坐标系统转换
四、坐标系统转换
4.1 两种坐标系
| 坐标系 | 名称 | 单位 | 表示 |
|---|---|---|---|
| (X, Y, Z) | 病人坐标系 | 毫米 | 解剖位置 |
| (I, R, C) | 数组坐标系 | 体素 | 数组索引 |
病人坐标系 (LPS):
- X轴:指向病人左侧 (Left)
- Y轴:指向病人后方 (Posterior)
- Z轴:指向病人头部 (Superior)
4.2 坐标转换函数 (当做黑盒使用即可)
IrcTuple = collections.namedtuple('IrcTuple', ['index', 'row', 'col'])
XyzTuple = collections.namedtuple('XyzTuple', ['x', 'y', 'z'])
def irc2xyz(coord_irc, origin_xyz, vxSize_xyz, direction_a):
"""数组坐标 → 病人坐标"""
# 步骤: IRC → CRI → 缩放 → 旋转 → 平移
cri_a = np.array(coord_irc)[::-1] # IRC翻转为CRI
origin_a = np.array(origin_xyz)
vxSize_a = np.array(vxSize_xyz)
coords_xyz = (direction_a @ (cri_a * vxSize_a)) + origin_a
# coords_xyz = (direction_a @ (idx * vxSize_a)) + origin_a
return XyzTuple(*coords_xyz)
def xyz2irc(coord_xyz, origin_xyz, vxSize_xyz, direction_a):
"""病人坐标 → 数组坐标"""
# 逆操作: 平移 → 逆旋转 → 缩放 → CRI翻转为IRC
origin_a = np.array(origin_xyz)
vxSize_a = np.array(vxSize_xyz)
coord_a = np.array(coord_xyz)
cri_a = ((coord_a - origin_a) @ np.linalg.inv(direction_a)) / vxSize_a
cri_a = np.round(cri_a)
return IrcTuple(int(cri_a[2]), int(cri_a[1]), int(cri_a[0]))
⭐ 五、自定义Dataset(重点)
5.1 PyTorch Dataset的核心要求
自定义Dataset类必须继承torch.utils.data.Dataset并实现两个方法:
| 方法 | 作用 | 要求 |
|---|---|---|
__len__() |
返回数据集大小 | 返回常量N |
__getitem__(ndx) |
返回第ndx个样本 | 对0~N-1的所有输入返回有效值 |
5.2 __init__()的作用:建目录,不搬书
__init__() 是数据集的初始化/准备阶段,核心任务是构建样本索引列表,而不是加载实际数据。
类比理解
| 方法 | 类比 | 做什么 |
|---|---|---|
__init__() |
建立图书目录 | 知道"有哪些样本"、"在哪里找" |
__getitem__() |
按目录取书 | 真正加载并返回某个样本的数据 |
为什么不在__init__()中加载所有数据?
效率考虑:如果在 __init__() 中加载所有数据到内存,会:
- 初始化时间很长
- 内存可能不够用(本章的CT数据有几十GB)
正确做法:
def __init__(self):
# ✅ 只存储"如何找到数据"的信息
self.file_paths = ['data/img1.png', 'data/img2.png', ...]
self.labels = [0, 1, 0, 1, ...]
def __getitem__(self, idx):
# ✅ 用到时才加载
image = load_image(self.file_paths[idx])
label = self.labels[idx]
return image, label
💡 一句话总结:
__init__()准备"地图",__getitem__()按图索骥取数据。
5.3 LunaDataset完整实现
from torch.utils.data import Dataset
class LunaDataset(Dataset):
def __init__(self,
val_stride=0,
isValSet_bool=None,
series_uid=None,
):
"""
参数:
val_stride: 验证集采样步长,每val_stride个样本取一个作为验证集
isValSet_bool: True返回验证集,False返回训练集,None返回全部
series_uid: 指定单个CT扫描(用于调试/可视化)
"""
# 1. 获取候选者列表的副本(避免修改缓存)
self.candidateInfo_list = copy.copy(getCandidateInfoList())
# 2. 指定单个CT扫描(用于调试/可视化)
if series_uid:
self.candidateInfo_list = [
x for x in self.candidateInfo_list if x.series_uid == series_uid
]
# 3. 划分训练集/验证集
if isValSet_bool:
assert val_stride > 0, val_stride
# 保留每val_stride个样本作为验证集
self.candidateInfo_list = self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
elif val_stride > 0:
# 删除每val_stride个样本(剩余为训练集)
del self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
log.info("{!r}: {} {} samples".format(
self,
len(self.candidateInfo_list),
"validation" if isValSet_bool else "training",
))
def __len__(self):
"""返回数据集大小"""
return len(self.candidateInfo_list)
def __getitem__(self, ndx):
"""返回第ndx个样本"""
# 1. 获取候选者信息
candidateInfo_tup = self.candidateInfo_list[ndx]
width_irc = (32, 48, 48)
# 2. 从CT中裁剪候选区域
candidate_a, center_irc = getCtRawCandidate(
candidateInfo_tup.series_uid,
candidateInfo_tup.center_xyz,
width_irc,
)
# 3. 转换为PyTorch张量
candidate_t = torch.from_numpy(candidate_a)
candidate_t = candidate_t.to(torch.float32)
candidate_t = candidate_t.unsqueeze(0) # 添加通道维度: (32,48,48) → (1,32,48,48)
# 4. 构建分类标签(one-hot编码)
pos_t = torch.tensor([
not candidateInfo_tup.isNodule_bool, # 非结节
candidateInfo_tup.isNodule_bool # 结节
],
dtype=torch.long,
)
# 5. 返回样本元组
return (
candidate_t, # 候选区域张量
pos_t, # 分类标签
candidateInfo_tup.series_uid, # CT的唯一标识符
torch.tensor(center_irc), # 中心坐标
)
5.3 返回值解释
# 调用示例
sample = LunaDataset()[0]
# 返回元组:
# (
# tensor([[[[-899., -903., ...]]]]) # shape: (1, 32, 48, 48) 候选区域
# tensor([0, 1]), # 标签: [非结节概率, 结节概率]
# '1.3.6...287966244644280690737019247886', # series_uid
# tensor([91, 360, 341]) # 中心坐标 (I, R, C)
# )
5.4 自定义Dataset模板
如果你要实现自己的Dataset,参考这个模板:
from torch.utils.data import Dataset
import torch
class MyDataset(Dataset):
def __init__(self, data_list, transform=None):
"""
初始化数据集
- 加载/解析数据文件
- 构建样本索引列表
- 可选:划分训练/验证集
"""
self.data_list = data_list
self.transform = transform
def __len__(self):
"""返回数据集大小"""
return len(self.data_list)
def __getitem__(self, idx):
"""
返回第idx个样本
- 加载原始数据
- 数据预处理/转换
- 转换为PyTorch张量
- 返回(输入, 标签)元组
"""
data = self.data_list[idx]
# 加载和预处理
x = load_and_preprocess(data)
y = get_label(data)
# 转换为张量
x_tensor = torch.tensor(x, dtype=torch.float32)
y_tensor = torch.tensor(y, dtype=torch.long)
# 可选的数据增强
if self.transform:
x_tensor = self.transform(x_tensor)
return x_tensor, y_tensor
六、缓存策略(性能优化)
6.1 为什么需要缓存?
- 每个CT扫描约225个体素(约3200万数据点)
- 但每个训练样本只需要215个体素(约3万数据点)
- 没有缓存时,每次获取样本都要加载整个CT,慢约50倍!
6.2 两种缓存策略
# 第一层:内存缓存(LRU缓存,保留最近1个CT)
@functools.lru_cache(1, typed=True)
def getCt(series_uid):
return Ct(series_uid)
# 第二层:磁盘缓存(使用diskcache库)
raw_cache = getCache('part2ch10_raw')
@raw_cache.memoize(typed=True)
def getCtRawCandidate(series_uid, center_xyz, width_irc):
ct = getCt(series_uid)
ct_chunk, center_irc = ct.getRawCandidate(center_xyz, width_irc)
return ct_chunk, center_irc
缓存流程:
第一次访问样本:
磁盘(.mhd/.raw) → Ct对象(内存)、 裁剪块(磁盘缓存)
第二次访问相同样本:
缓存 → 直接返回,不需要重新执行函数
⚠️ 注意:如果修改了函数定义,需要清除缓存目录
data-unversioned/cache
七、训练集与验证集分隔
7.1 分隔策略
# 3. 划分训练集/验证集
if isValSet_bool:
assert val_stride > 0, val_stride
# 保留每val_stride个样本作为验证集
self.candidateInfo_list = self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
elif val_stride > 0:
# 删除每val_stride个样本(剩余为训练集)
del self.candidateInfo_list[::val_stride]
assert self.candidateInfo_list
7.2 分隔原则
- 代表性:两个集合都应包含预期输入的所有变体
- 无污染:训练集样本不应出现在验证集中(数据泄露)
- 一致性:依赖稳定的排序确保可重复性
💡 提示:某些任务需要确保同一病人的数据只出现在一个集合中
数据可视化
八、数据可视化
%matplotlib inline
from p2ch10.vis import findPositiveSamples, showCandidate
# 查找阳性样本
positiveSample_list = findPositiveSamples()
# 可视化特定CT
series_uid = positiveSample_list[11][2]
showCandidate(series_uid)
可视化的价值:
- 验证数据加载是否正确
- 直观了解输入数据的特征
- 调试时快速定位问题
九、本章小结
| 主题 | 要点 |
|---|---|
| 数据加载 | 使用SimpleITK加载MetaIO格式的CT扫描 |
| 数据清理 | 将HU值裁剪到[-1000, 1000]范围 |
| 坐标转换 | 在病人坐标(XYZ)和数组坐标(IRC)之间转换 |
| 自定义Dataset | 实现__len__()和__getitem__()两个必需方法 |
| 缓存策略 | 内存缓存(lru_cache) + 磁盘缓存(diskcache) |
| 数据分隔 | 使用步长采样划分训练集和验证集 |
十、补充
dict.setdefault()详解
你问的这行代码是 Python 中处理字典的经典技巧,核心作用是:给字典 diameter_dict 中键为 series_uid 的值(一个列表)追加新的元素,若该键不存在则先创建并初始化值为空列表。我拆成两步帮你彻底看懂:
第一步:先理解核心方法 dict.setdefault()
setdefault 是字典的内置方法,专门解决“键不存在时初始化,存在时直接取值”的问题,语法:
字典.setdefault(键, 默认值)
- 如果「键」已存在:返回字典中该键对应的现有值;
- 如果「键」不存在:先给字典添加「键: 默认值」,再返回这个默认值。
通俗类比
这就像你有一个抽屉柜(字典),每个抽屉对应一个 series_uid:
- 打开抽屉(取键)时,如果抽屉存在 → 直接用里面的东西(现有列表);
- 如果抽屉不存在 → 先新建一个空抽屉(初始化空列表),再用这个新抽屉。
第二步:整行代码的完整逻辑
我们结合代码上下文拆解:
diameter_dict.setdefault(series_uid, []).append(
(annotationCenter_xyz, annotationDiameter_mm)
)
拆解成两步(等价写法,更易读):
# 步骤1:获取/初始化列表(核心是setdefault)
# - 若series_uid在diameter_dict中,取对应的列表;
# - 若不存在,添加series_uid: [],并返回空列表
temp_list = diameter_dict.setdefault(series_uid, [])
# 步骤2:向列表追加新元素(元组形式)
temp_list.append( (annotationCenter_xyz, annotationDiameter_mm) )
实际场景示例(一看就懂)
假设处理CSV时遇到两行数据:
| series_uid | x | y | z | diameter |
|---|---|---|---|---|
| 123 | 10.5 | 20.3 | 30.1 | 5.2 |
| 123 | 11.8 | 22.5 | 31.7 | 4.9 |
代码执行过程:
-
处理第一行(series_uid=123):
123不在diameter_dict中 →setdefault创建123: [],返回空列表;- 追加元组
((10.5,20.3,30.1), 5.2)→diameter_dict[123] = [((10.5,20.3,30.1), 5.2)]。
-
处理第二行(series_uid=123):
123已存在 →setdefault返回现有列表[((10.5,20.3,30.1), 5.2)];- 追加元组
((11.8,22.5,31.7), 4.9)→diameter_dict[123] = [((10.5,20.3,30.1),5.2), ((11.8,22.5,31.7),4.9)]。
最终 diameter_dict 的结构:
{
"123": [
((10.5, 20.3, 30.1), 5.2),
((11.8, 22.5, 31.7), 4.9)
]
}
第三步:为什么不用普通的 dict[key]?
如果直接写 diameter_dict[series_uid].append(...),会有致命问题:
- 当
series_uid首次出现(字典中无该键),会直接报错KeyError: '123'; - 而
setdefault会自动初始化空列表,完美避免这个错误。
等价替代写法(新手更易理解)
# 效果和setdefault完全一致,但代码更长
if series_uid not in diameter_dict:
diameter_dict[series_uid] = [] # 不存在则初始化
diameter_dict[series_uid].append( (annotationCenter_xyz, annotationDiameter_mm) )
总结
setdefault(series_uid, []):核心是“无则建空列表,有则取现有列表”;.append(元组):向列表追加结节的“中心坐标+直径”信息;- 整体作用:按
series_uid分组存储每个结节的坐标和直径,一个series_uid对应多个结节信息(列表形式)。
这行代码是处理“一对多”字典结构的最优写法之一,比“先判断键是否存在再初始化”更简洁高效~


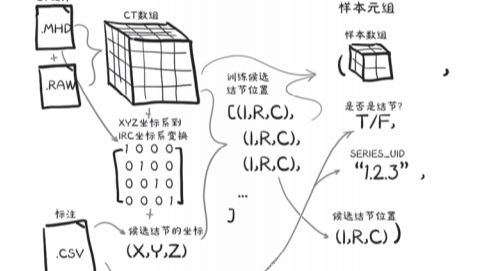 本章围绕医学影像数据加载展开,核心是将 LUNA16 数据集的 CT 扫描文件(.mhd/.raw)与 CSV 标注文件转换为 PyTorch 可用的训练样本。首先合并并匹配候选结节标注信息,通过 Ct 类加载 CT 数据并裁剪 HU 值至合理范围,完成病人坐标系(XYZ)与数组坐标系(IRC)的转换;接着自定义 LunaDataset 类实现数据集核心方法,采用内存 + 磁盘缓存策略优化性能,按步长采样划分训练 / 验证集;最终输出包含候选区域张量、分类标签等的标准化训练样本,形成完整的数据准备流水线。
本章围绕医学影像数据加载展开,核心是将 LUNA16 数据集的 CT 扫描文件(.mhd/.raw)与 CSV 标注文件转换为 PyTorch 可用的训练样本。首先合并并匹配候选结节标注信息,通过 Ct 类加载 CT 数据并裁剪 HU 值至合理范围,完成病人坐标系(XYZ)与数组坐标系(IRC)的转换;接着自定义 LunaDataset 类实现数据集核心方法,采用内存 + 磁盘缓存策略优化性能,按步长采样划分训练 / 验证集;最终输出包含候选区域张量、分类标签等的标准化训练样本,形成完整的数据准备流水线。

 浙公网安备 33010602011771号
浙公网安备 33010602011771号