文献阅读 | PlantDeepSEA, a deep learning-based web service to predict the regulatory effects of genomic variants in plants
Hu Zhao, Zhuo Tu, Yinmeng Liu, Zhanxiang Zong, Jiacheng Li, Hao Liu, Feng Xiong, Jinling Zhan, Xuehai Hu, Weibo Xie, PlantDeepSEA, a deep learning-based web service to predict the regulatory effects of genomic variants in plants, Nucleic Acids Research, 2021;, gkab383, https://doi.org/10.1093/nar/gkab383
表征植物中基因组变异的调节作用仍然是一个挑战。尽管已经有几种基于深度学习模型和大规模染色质分析数据的工具可用于预测调控元件和变异效应,但尚未报道植物中的专用工具或网络服务。
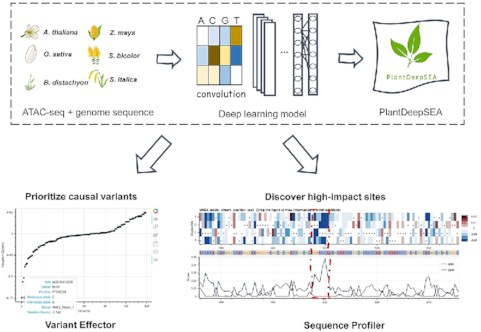
PlantDeepSEA 提供两个主要功能。一种称为变异效应器,旨在预测序列变异对染色质可及性的影响。另一个是 Sequence Profiler,这是一种执行“计算机饱和诱变”分析以在一个序列中发现高影响位点(例如,顺式-调节元素)。在独立测试集上进行验证时,PlantDeepSEA 中深度学习模型的接收器操作特征曲线下的面积范围为 0.93 到 0.99。PlantDeepSEA 可以帮助确定调节因果变异的优先级,并可能提高我们对它们在植物不同组织中的作用机制的理解。
数量性状位点 (QTL) 分析和全基因组关联研究 (GWAS) 已被广泛用于剖析植物复杂性状的遗传基础。然而,由于许多中性基因组变异也与 GWAS 中的性状显著相关,因此仅根据关联结果很难确定因果变异。此外,很难解决变异的潜在机制,尤其是对于非编码变异 (NCV)。最近的一篇综述文章总结了在六种主要作物中克隆的 364 个 QTL,并表明在玉米中,64% 的因果变异落在非编码区,证明了植物科学中 NCV 优先级和顺式调节元件 (CRE)注释的重要性。
基于深度神经网络 (DNN) 的模型已被证明是强大的,可以从基因组序列中自动提取复杂和相关的特征,并准确有效地学习和预测表观遗传状态。DeepSEA(基于深度学习的序列分析器)、DeepBind、Basset和 Basset 的继任者 Basenji是 DNN 的代表性框架。相比之下,DeepSEA 的结构更简单,可以在短时间内训练和注释基因组片段和变异。此外,基于 PyTorch 的深度学习库 Selene 可以轻松构建和训练 DNN 模型。
尽管大规模染色质分析数据已经可用,并且一些基于深度学习模型的工具已经在人类身上实现了最先进的性能,但在植物中还没有专门的工具或网络服务的报道。此外,对于那些只想优先考虑因果变异或识别基因组特定区域中的 CRE 的人来说,从头开始构建深度学习模型非常耗时且费力。因此,有必要构建一个在线 Web 服务,以基于植物中的深度学习模型来预测变异效应和 CRE。
作者从 NCBI 数据库中收集了拟南芥 (Arabidopsis thaliana)的 ATAC-seq 数据,并自己分析了一些其他物种的ATAC-seq数据。基于使用 Selene 实现的深度学习框架 DeepSEA,作者构建了DNN模型(架构见下图)。按照 训练集:验证集:测试集=8:1:1 的比例,进行了训练与评估。

截至目前,作者已收集了拟南芥、水稻、玉米、谷子、高粱、二穗短柄草等六种代表性植物的多个组织中的ATAC-seq数据。最终获得了七个经过训练的模型,水稻两个模型和每个其他物种一个模型。模型输出是一个由 0 到 1 的值组成的向量,表示该序列属于每个样本中的 OCR 的概率。
Web 服务器是使用 Django Web 框架实现的。服务器具有72 CPU cores and two GPU cores。
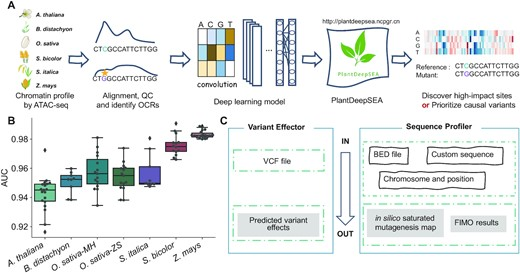
在实践中,作者进行了水稻DEP1基因非编码原因多态性的排序(prioritizing non-coding causal polymorphisms),并在玉米QTL UPA2中发现了高影响位点。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号