文献精读_2024.06.13
Universal and extensible language-vision models for organ segmentation and tumor detection from abdominal computed tomography
来源:https://doi.org/10.1016/j.media.2024.103226
GitHub仓库: https://github.com/ljwztc/CLIP-Driven-Universal-Model
-
第一眼,仓库上面放了两篇文章,而不是这篇,好奇什么情况🧐
看了一眼本篇文章,这两篇文章是 CLIP-Driven Universal Model 最开始出现的地方,一篇发表在ICCV 2023上面,另一篇发表在MICCAI 2023上面(章节2.5) -
已集成在MONAI框架里,可以调用
-
与之前发表的工作相比,这篇工作做了进一步扩展(见下)
- 设计了新模块(language-driven parameter generator),改善各种标签之间的纠缠(章节3.2.2)
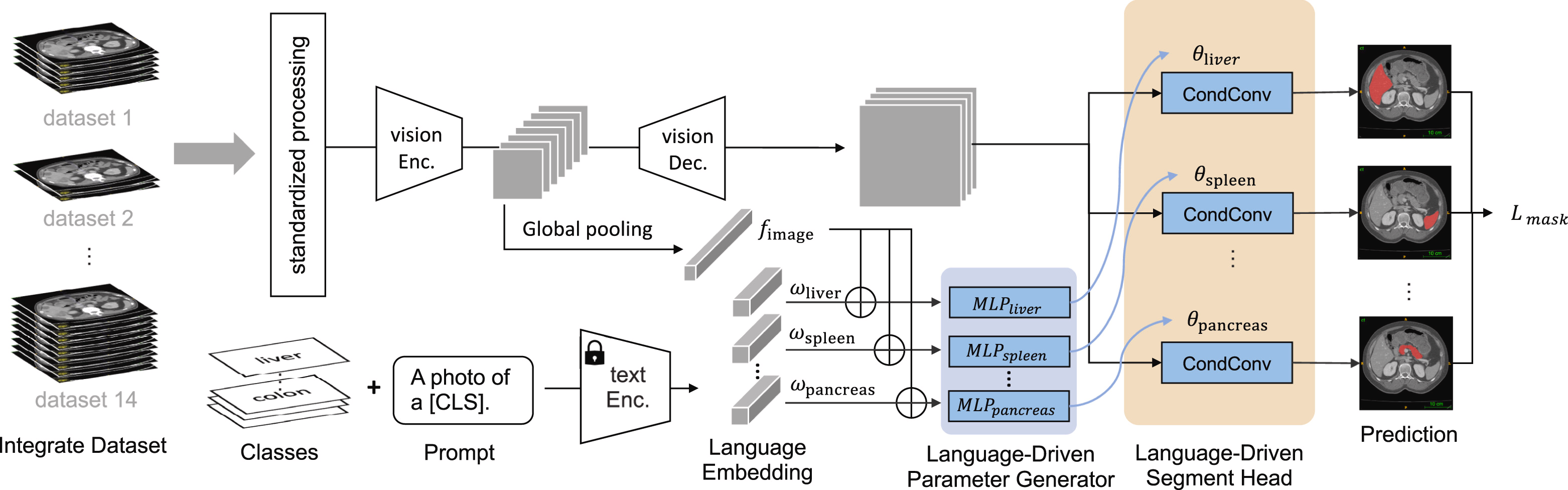
- [CLS]可以是器官的名字或肿瘤的名字,经过Text Enc.进行文本编码
- Text Enc.可以是CLIP(Radford et al.,2021)/BioBERT(Yasunaga et al.,2022)
- Vision Enc.之后的特征图经过GAP(global average pooling)得到全局特征
- 将编码后的文本特征和GAP之后的图像特征concat到一起(原文:where denotes the concatenation),传入MLP,得到对应[CLS]的参数,用于之后的分割网络的参数初始化
- LPG模块对每个[CLS]使用单独的MLP,以克服ICCV版本中存在的纠缠问题(依赖于单个MLP)
-
加入了四个外部数据集,测试泛化性(图4a)

-
进行了其他部分标记分割方法的基准测试(表4和5)
-
对具有挑战性(原文用词:challenging)的肿瘤进行了案例研究(原文用词:case studies),与医生(原文用词:human experts)的勾画结果进行了比较(图5)
左图:红线:GT1,绿线:GT2,蓝线:预测结果
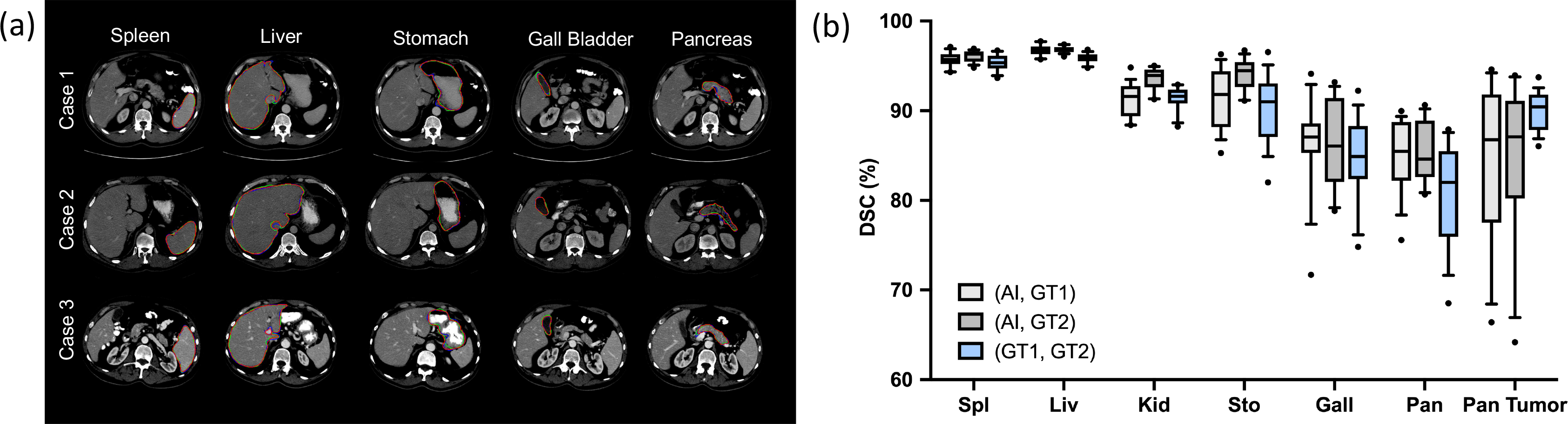
-
测试了临床数据,评估了模型对于临床上面被漏诊的小肿瘤的检测能力(章节4.5)
-
分析了模型分割胰腺肿瘤子类的可迁移性(表9)
- 设计了新模块(language-driven parameter generator),改善各种标签之间的纠缠(章节3.2.2)
-
其余文章要点
- Class-specific Segment Head
同一套架构,初始化不同,每个[CLS]有一个对应的初始化参数,于是产生了多个Class-specific Segment Head,一个头分割一个[CLS]
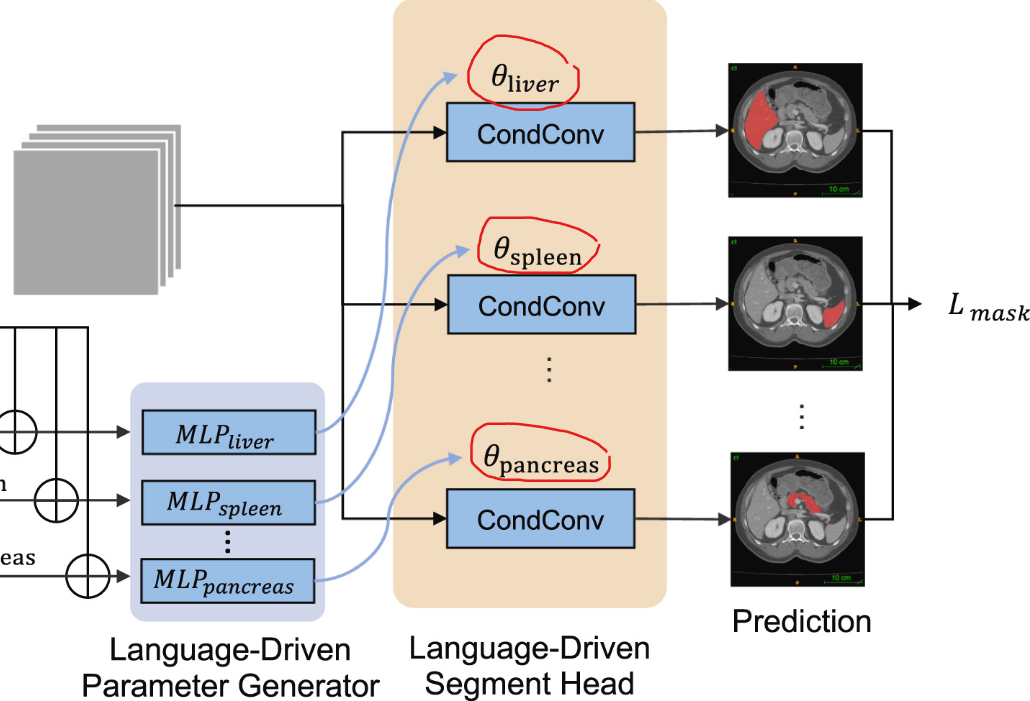
- 文章强调的增量学习,比较简单,就是利用Class-specific Segment Head,轻量化分割网络。这里的防遗忘策略是独立化每个类别的分割。另外,还有分割过程中的伪标签,可以看作是一种对于过去训练结果的一种保留。
- Class-specific Segment Head


 浙公网安备 33010602011771号
浙公网安备 33010602011771号