细胞图像数据的主动学习
通过细胞图像的标签对模型性能的影响,为数据设置优先级和权重。
许多机器学习任务的主要障碍之一是缺乏标记数据。而标记数据可能会耗费很长的时间,并且很昂贵,因此很多时候尝试使用机器学习方法来解决问题是不合理的。
为了解决这个问题,机器学习领域出现了一个叫做主动学习的领域。主动学习是机器学习中的一种方法,它提供了一个框架,根据模型已经看到的标记数据对未标记的数据样本进行优先排序。如果想
细胞成像的分割和分类等技术是一个快速发展的领域研究。就像在其他机器学习领域一样,数据的标注是非常昂贵的,并且对于数据标注的质量要求也非常的高。针对这一问题,本篇文章介绍一种对红细胞和白细胞图像分类任务的主动学习端到端工作流程。
我们的目标是将生物学和主动学习的结合,并帮助其他人使用主动学习方法解决生物学领域中类似的和更复杂的任务。
本篇文主要由三个部分组成:
- 细胞图像预处理——在这里将介绍如何预处理未分割的血细胞图像。
- 使用CellProfiler提取细胞特征——展示如何从生物细胞照片图像中提取形态学特征,以用作机器学习模型的特征。
- 使用主动学习——展示一个模拟使用主动学习和不使用主动学习的对比实验。
细胞图像预处理
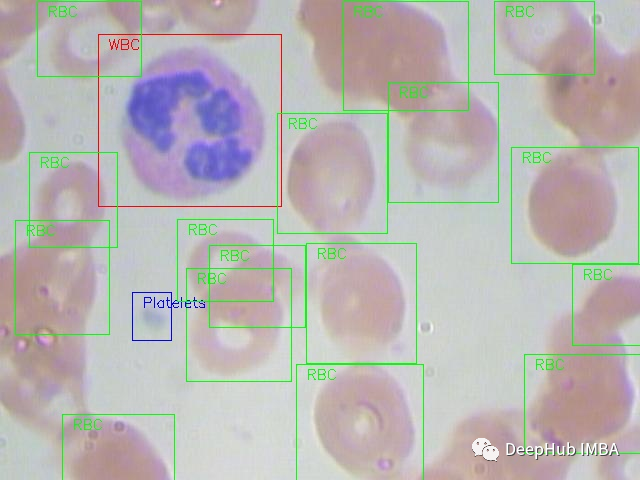
我们将使用在MIT许可的血细胞图像数据集(GitHub和Kaggle)。每张图片都根据红细胞(RBC)和白细胞(WBC)分类进行标记。对于这4种白细胞(嗜酸性粒细胞、淋巴细胞、单核细胞和中性粒细胞)还有附加的标签,但在本文的研究中没有使用这些标签。
下面是一个来自数据集的全尺寸原始图像的例子:
创建样本DF
原始数据集包含一个export.py脚本,它将XML注释解析为一个CSV表,其中包含每个细胞的文件名、细胞类型标签和边界框。
原始脚本没有包含cell_id列,但我们要对单个细胞进行分类,所以我们稍微修改了代码,添加了该列并添加了一列包括image_id和cell_id的filename列:
import os, sys, randomimport xml.etree.ElementTree as ETfrom glob import globimport pandas as pdfrom shutil import copyfileannotations = glob('BCCD_Dataset/BCCD/Annotations/*.xml')df = []for file in annotations:#filename = file.split('/')[-1].split('.')[0] + '.jpg'#filename = str(cnt) + '.jpg'filename = file.split('\\')[-1]filename =filename.split('.')[0] + '.jpg'row = []parsedXML = ET.parse(file)cell_id = 0for node in parsedXML.getroot().iter('object'):blood_cells = node.find('name').textxmin = int(node.find('bndbox/xmin').text)xmax = int(node.find('bndbox/xmax').text)ymin = int(node.find('bndbox/ymin').text)ymax = int(node.find('bndbox/ymax').text)row = [filename, cell_id, blood_cells, xmin, xmax, ymin, ymax]df.append(row)cell_id += 1data = pd.DataFrame(df, columns=['filename', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax'])data['image_id'] = data['filename'].apply(lambda x: int(x[-7:-4]))data[['filename', 'image_id', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax']].to_csv('bccd.csv', index=False)
裁剪
为了能够处理数据,第一步是根据边界框坐标裁剪全尺寸图像。这就产生了很多大小不一的细胞图像:
完整文章:
https://avoid.overfit.cn/post/e920ecde825b4136ae57fbcd325b9097




 浙公网安备 33010602011771号
浙公网安备 33010602011771号