划分聚类分析(K 均值聚类、围绕中心点的划分 PAM)
1 划分聚类分析
1.1 K 均值聚类
最常见的划分方法是K均值聚类分析。从概念上讲,K均值算法如下:
(1) 选择K个中心点(随机选择K行);
(2) 把每个数据点分配到离它最近的中心点;
(3) 重新计算每类中的点到该类中心点距离的平均值(也就说,得到长度为p的均值向量,这里的p是变量的个数);
(4) 分配每个数据到它最近的中心点;
(5) 重复步骤(3)和步骤(4)直到所有的观测值不再被分配或是达到最大的迭代次数(R把10次作为默认迭代次数)。
R软件使用Hartigan & Wong(1979)提出的有效算法,这种算法是把观测值分成k组并使得观测值到其指定的聚类中心的平方的总和为最小。
K均值聚类能处理比层次聚类更大的数据集。在R中K均值的函数格式是kmeans(x,centers),这里x表示数值数据集(矩阵或数据框),centers是要提取的聚类数目。函数返回类的成员、类中心、平方和(类内平方和、类间平方和、总平方和)和类大小。
由于K均值聚类在开始要随机选择k个中心点,在每次调用函数时可能获得不同的方案。使用set.seed()函数可以保证结果是可复制的。此外,聚类方法对初始中心值的选择也很敏感。
kmeans()函数有一个nstart选项尝试多种初始配置并输出最好的一个。例如,加上nstart=25会生成25个初始配置。通常推荐使用这种方法。
不像层次聚类方法,K均值聚类要求你事先确定要提取的聚类个数。同样,NbClust包可以用来作为参考。另外,在K均值聚类中,类中总的平方值对聚类数量的曲线可能是有帮助的。可根据图中的弯曲选择适当的类的数量。
图像可以用下面的代码产生:
wssplot<-function(data,nc=15,seed=123){
wss<-(nrow(data)-1)*sum(apply(data, 2, var))
for (i in 2:nc){
set.seed(seed)
wss[i]<-sum(kmeans(data,centers = i)$withiness)}
plot(1:nc,wss,type="b",xlab="Number of Clusters",
ylab="within groups sum of squares")}
data参数是用来分析的数值数据,nc是要考虑的最大聚类个数,而seed是一个随机数种子。
用K均值聚类来处理包含178种意大利葡萄酒中13种化学成分的数据集。该数据最初来自于UCI机器学习库(http://www.ics.uci.edu/~mlearn/MLRepository.html),但是可以通过rattle
包获得。在这个数据集里,观测值代表三种葡萄酒的品种,由第一个变量(类型)表示。
(1)葡萄酒数据的K均值聚类:
data(wine, package="rattle") head(wine)
df <- scale(wine[-1]) #标准化数据,均值未0,方差为1
wssplot(df)
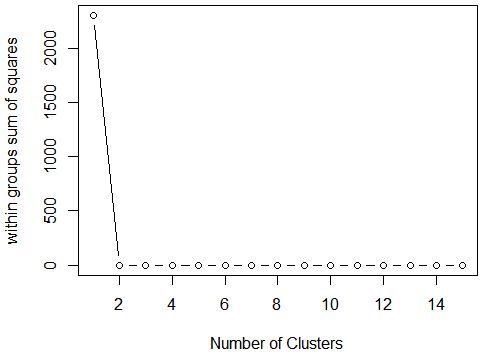
结果分析:画出组内的平方和和提取的聚类个数的对比。从一类到三类下降得很快(之后下降得很慢),建议选用聚类个数为二、三的解决方案。
library(NbClust) set.seed(1234) devAskNewPage(ask=TRUE) nc <- NbClust(df, min.nc=2, max.nc=15, method="kmeans")
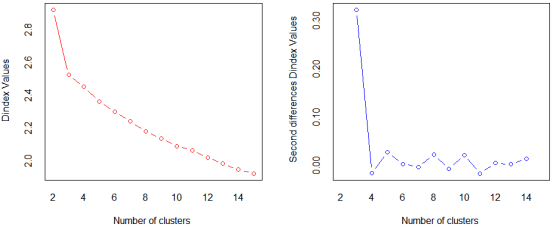
结果分析:左图表示从一类到三类变化时,组内的平方总和有一个明显的下降趋势。三类之后,下降的速度减弱,暗示着聚成三类可能对数据来说是一个很好的拟合。
table(nc$Best.n[1,]) #查看分组支持数
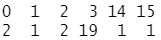
结果分析:这里,19个评判准则赞同聚类个数为3,2个判定准则赞同聚类个数为2,等等。
barplot(table(nc$Best.n[1,]), #分组支持数可视化
xlab="Number of Clusters", ylab="Number of Criteria",
main="Number of Clusters Chosen by 26 Criteria")
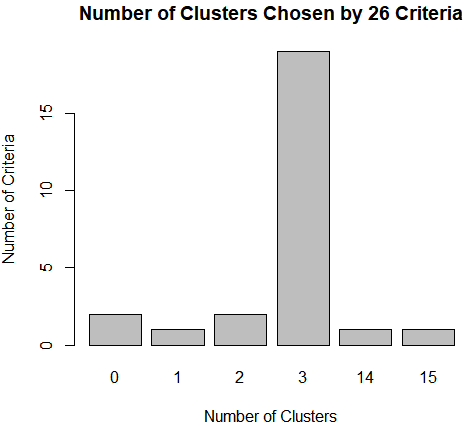
结果分析:使用NbClust包中的26个指标推荐的聚类个数,可以看到聚为3类拥有最多的支持数。
set.seed(1234) fit.km <- kmeans(df, 3, nstart=25) #进行K均值聚类分析 fit.km$size #查看聚成三类后,每类有多少变量
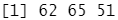
查看标准化之后数据据的维度,有178行,13列

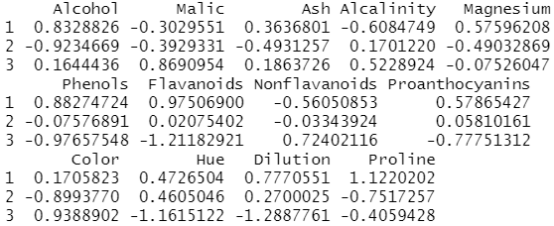
fit.km$centers #查看聚类的中心
aggregate(wine[-1], by=list(cluster=fit.km$cluster), mean) #使用aggregate()函数和类的成员来得到原始矩阵中每一类的变量均值
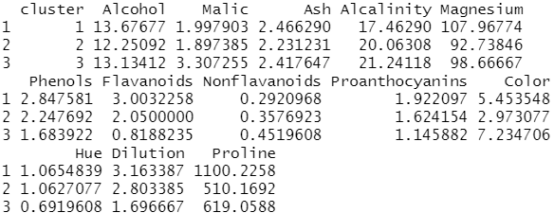
交叉列表类型(葡萄酒品种)和类成员由下面的代码表示:
ct.km <- table(wine$Type, fit.km$cluster) ct.km

可以用flexclust包中的兰德指数(Rand index)来量化类型变量和类之间的协议:
library(flexclust) randIndex(ct.km)
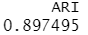
结果分析:调整的兰德指数为两种划分提供了一种衡量两个分区之间的协定,即调整后机会的量度。它的变化范围是从–1(不同意)到1 (完全同意)。葡萄酒品种类型和类的解决方案之间的协定约等于0.9,结果不坏。
1.2 围绕中心点的划分
因为K均值聚类方法是基于均值的,所以它对异常值是敏感的。一个更稳健的方法是围绕中
心点的划分(PAM),K均值聚类一般使用欧几里得距离,而PAM可以使用任意的距离来计算。
PAM算法如下:
(1) 随机选择K个观测值(每个都称为中心点);
(2) 计算观测值到各个中心的距离/相异性;
(3) 把每个观测值分配到最近的中心点;
(4) 计算每个中心点到每个观测值的距离的总和(总成本);
(5) 选择一个该类中不是中心的点,并和中心点互换;
(6) 重新把每个点分配到距它最近的中心点;
(7) 再次计算总成本;
(8) 如果总成本比步骤(4)计算的总成本少,把新的点作为中心点;
(9) 重复步骤(5)~(8)直到中心点不再改变。
可以使用cluster包中的pam()函数使用基于中心点的划分方法。格式如下:
pam(x, k, metric="euclidean", stand=FALSE)
x表示数据矩阵或数据框,k表示聚类的个数,metric表示使用的相似性/相异性的度量,而stand是一个逻辑值,表示是否有变量应该在计算该指标之前被标准化。
(1)对葡萄酒数据使用基于质心的划分方法:
library(cluster) set.seed(1234) fit.pam <- pam(wine[-1], k=3, stand=TRUE) #聚类数据的标准化 fit.pam$medoids #输出中心点
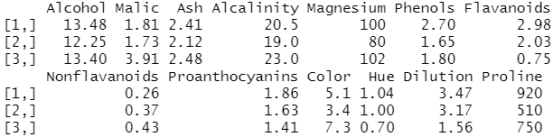
结果分析:这里得到的中心点是葡萄酒数据集中实际的观测值。
clusplot(fit.pam, main="Bivariate Cluster Plot") #画出聚类方案
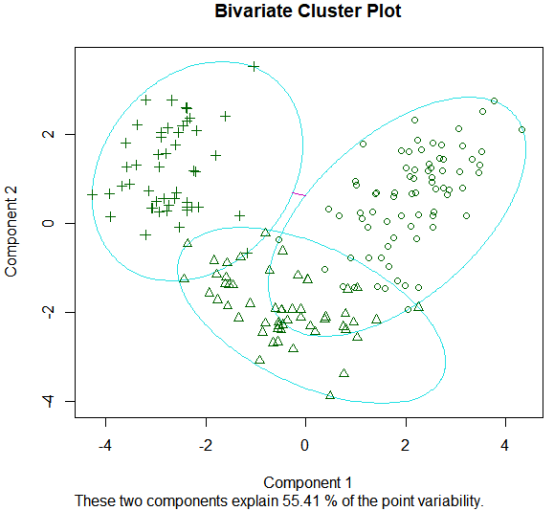
PAM在如下例子中的表现不如K均值:
ct.pam <- table(wine$Type, fit.pam$clustering) ct.pam

randIndex(ct.pam)
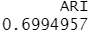
结果分析:flexclust包中的兰德指数(Rand index)来量化类型变量和类之间的协议,调整的兰德指数为两种划分提供了一种衡量两个分区之间的协定,即调整后机会的量度。它的变化范围是从–1(不同意)到1 (完全同意)。葡萄酒品种类型和类的解决方案之间的协定约等于0.7,结果没有很好。
本文来自博客园,作者:zhang-X,转载请注明原文链接:https://www.cnblogs.com/YY-zhang/p/15138191.html




 浙公网安备 33010602011771号
浙公网安备 33010602011771号