WCHN-CTseg
WCHN-CTseg
前两天我们装了 Matlab 和 SPM12 两个软件,今天我们就来讲怎么运行这个 CTseg (这是一个可以进行脑CT分割、归一化、颅骨剥离和总脑/颅内容积计算的一个算法~)
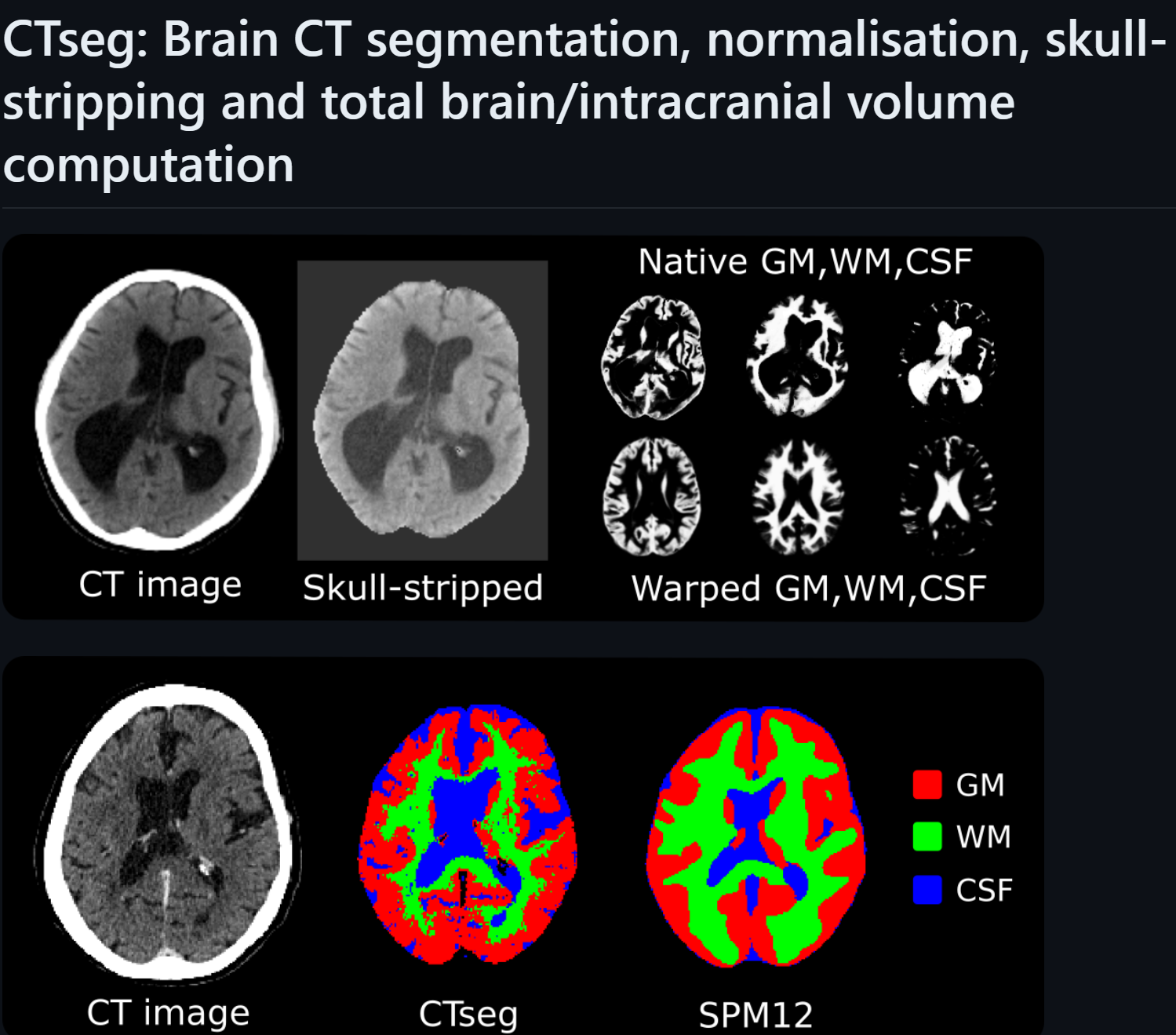
下载所需包
首先要下的两个软件: Matlab 和 SPM12 ,详情请看这里:Matlab 安装及添加 SPM 12 - Ray-Z - 博客园 (cnblogs.com)
下载CTseg 包
在 Github 上下载 CTseg 安装包 ,Github:https://github.com/WCHN/CTseg
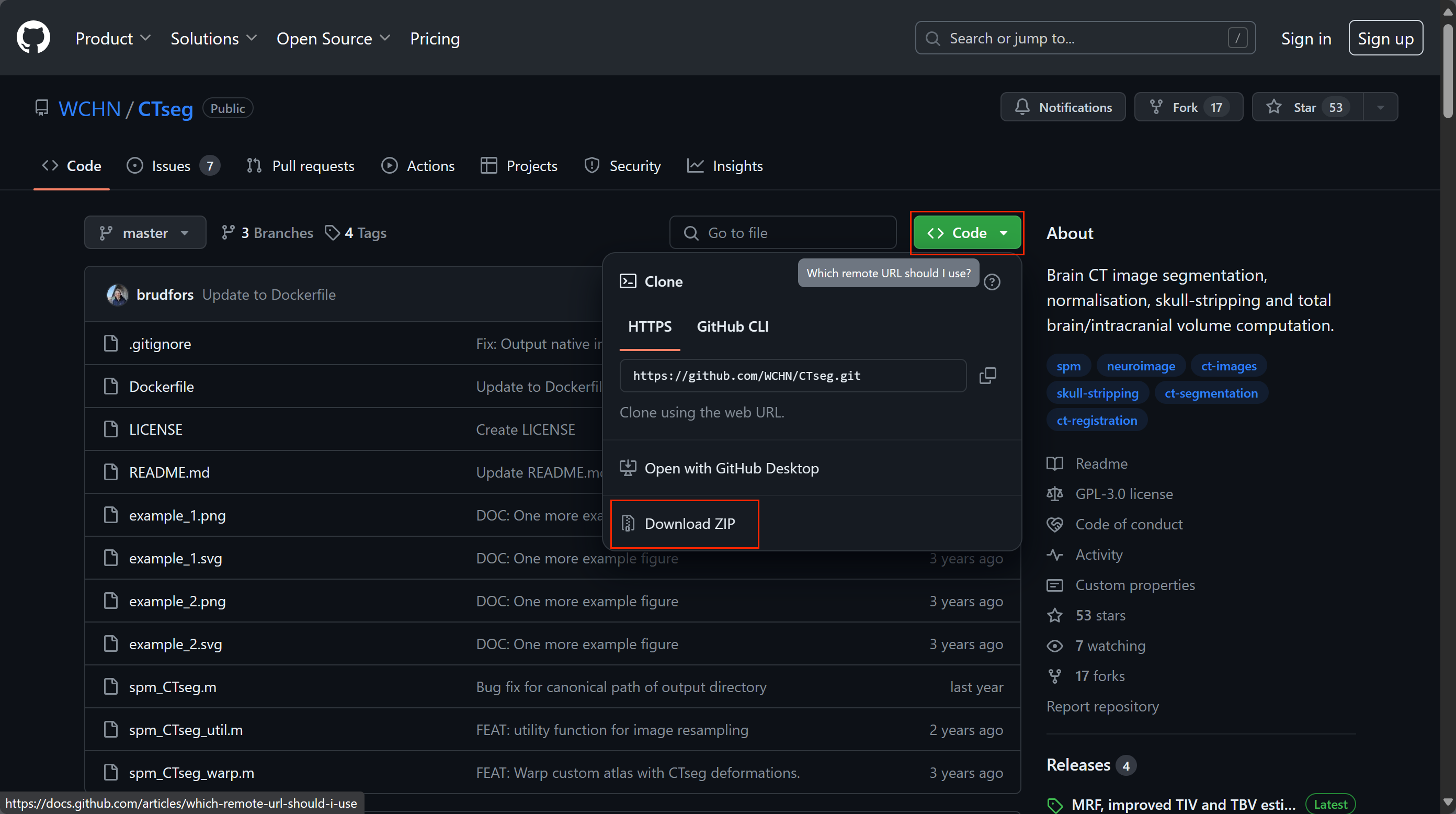
这个页面下面会有一些介绍,大概可以看一下
然后在依赖工具这里还需要下载一个包:Multi-Brain toolbox
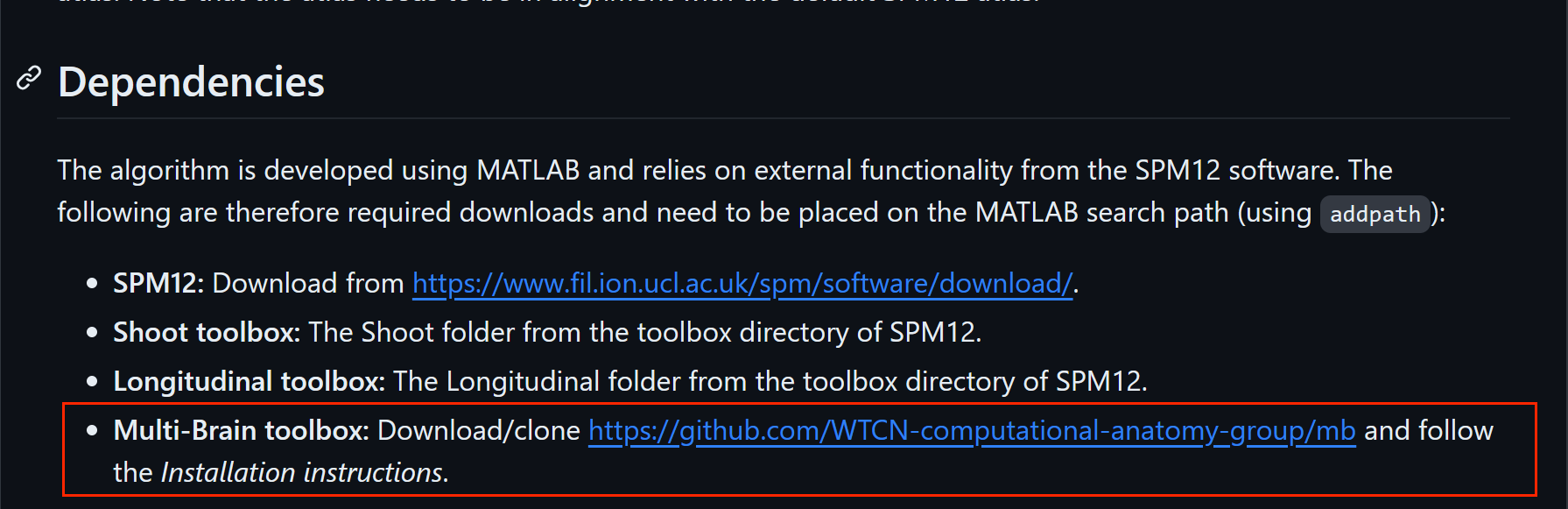
Tips:
我这边是在 Windows 系统下下载了 Matlab 软件使用的,如果你没有 Matlab ,他这里面有个 Docker ,这个我没试过,所以具体怎样我也不知道~~~
下载 Multi-Brain toolbox 包
然后,我们下载 Multi-Brain toolbox ,同 CTseg ,下载完会得到 mb-master 的压缩包。地址:https://github.com/WTCN-computational-anatomy-group/mb
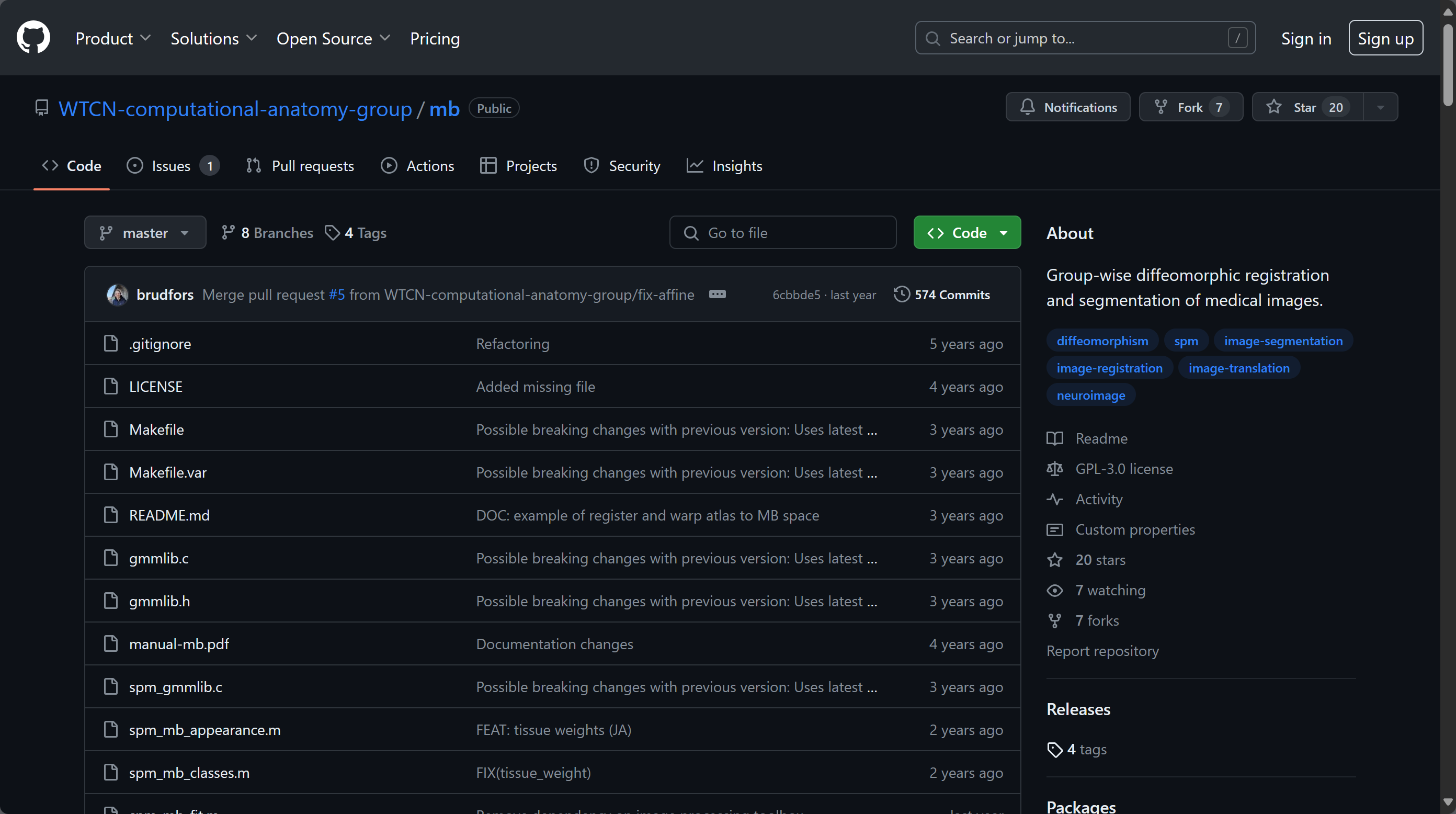
下载完以后,阅读这个页面的 Dependencies 和 Installation instructions ,会发现还需要安装一个编译器 MS ( Windows 系统下才会需要下 /(ㄒoㄒ)/~~ )
维基百科:https://en.wikibooks.org/wiki/SPM/SPM12_MEX_Compilation_on_Windows
下载 MS
从给的维基百科地址打开可以看到有下载 MSYS2 的网址,进去下载就可以了
下载完之后就安装就可以了,在这个下载页面也提供了具体的安装步骤~
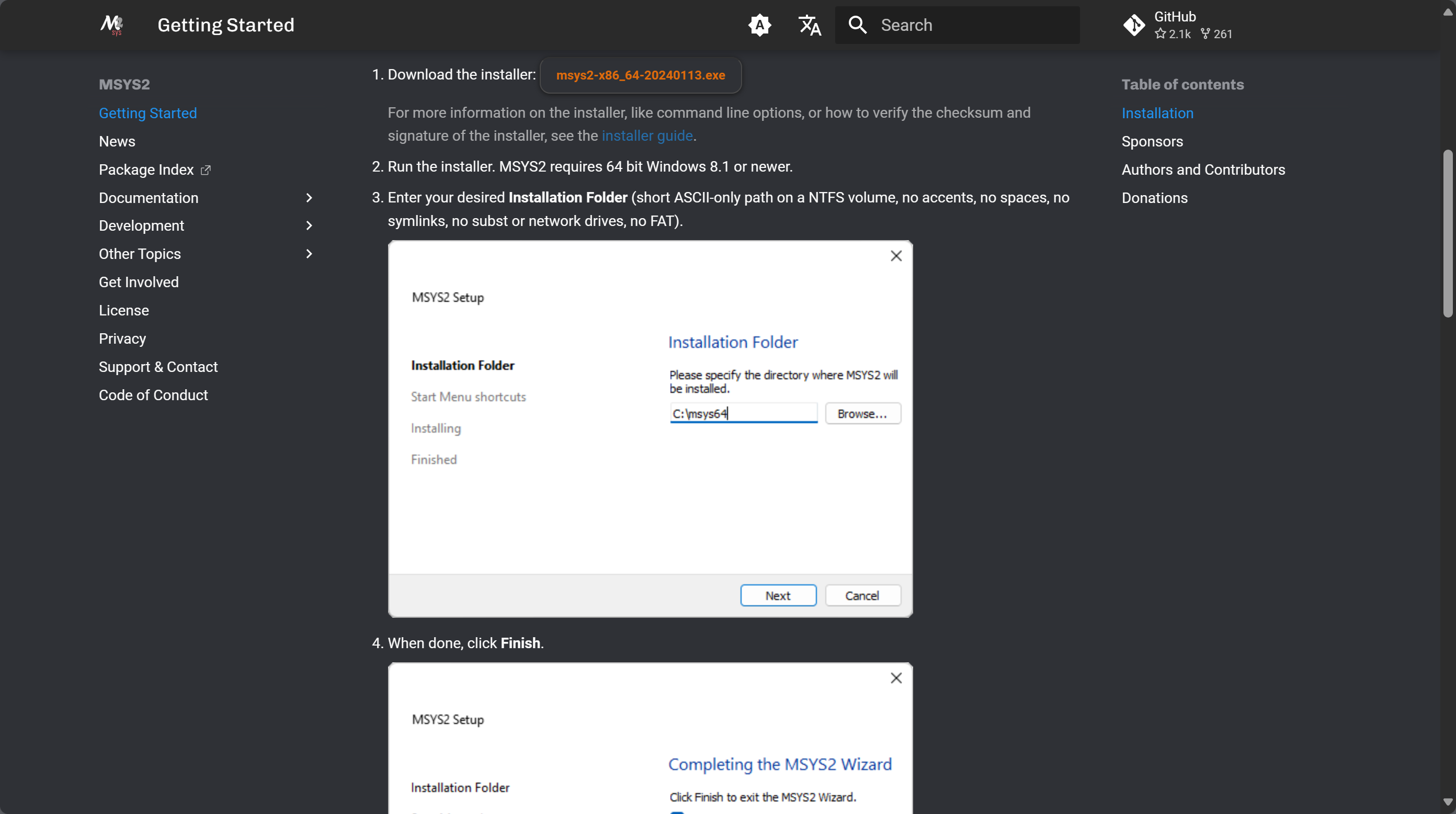
到这里,所有需要的包和软件都下载安装完毕啦~
配置
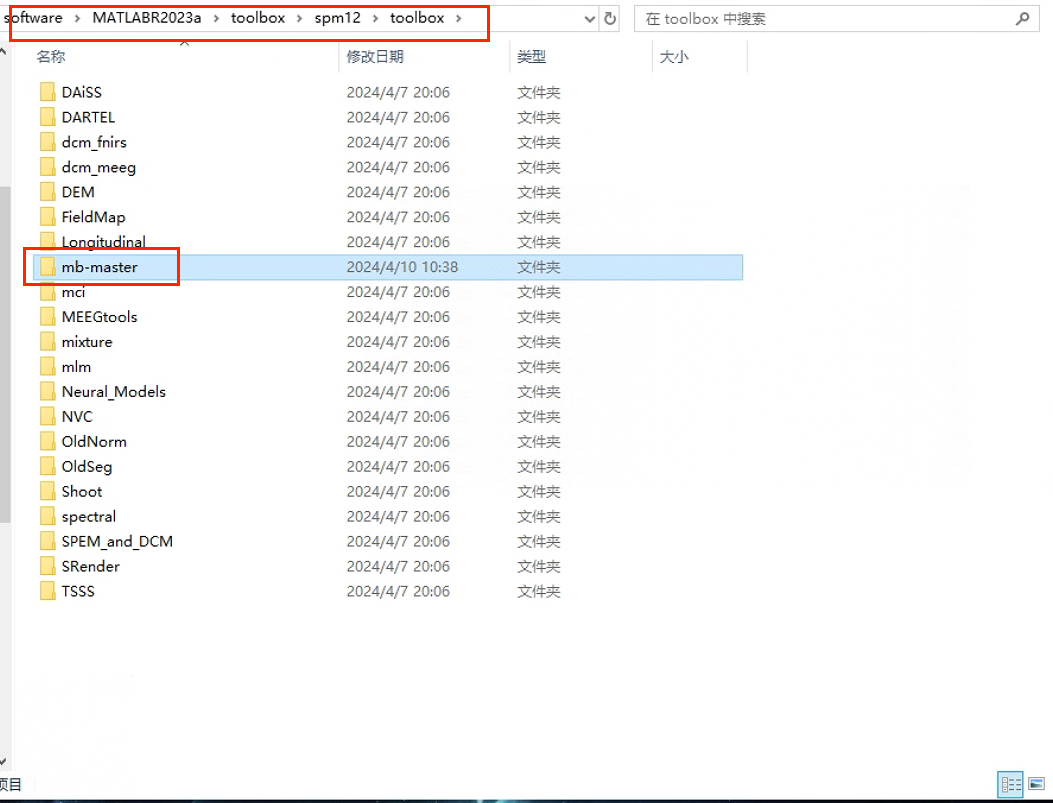
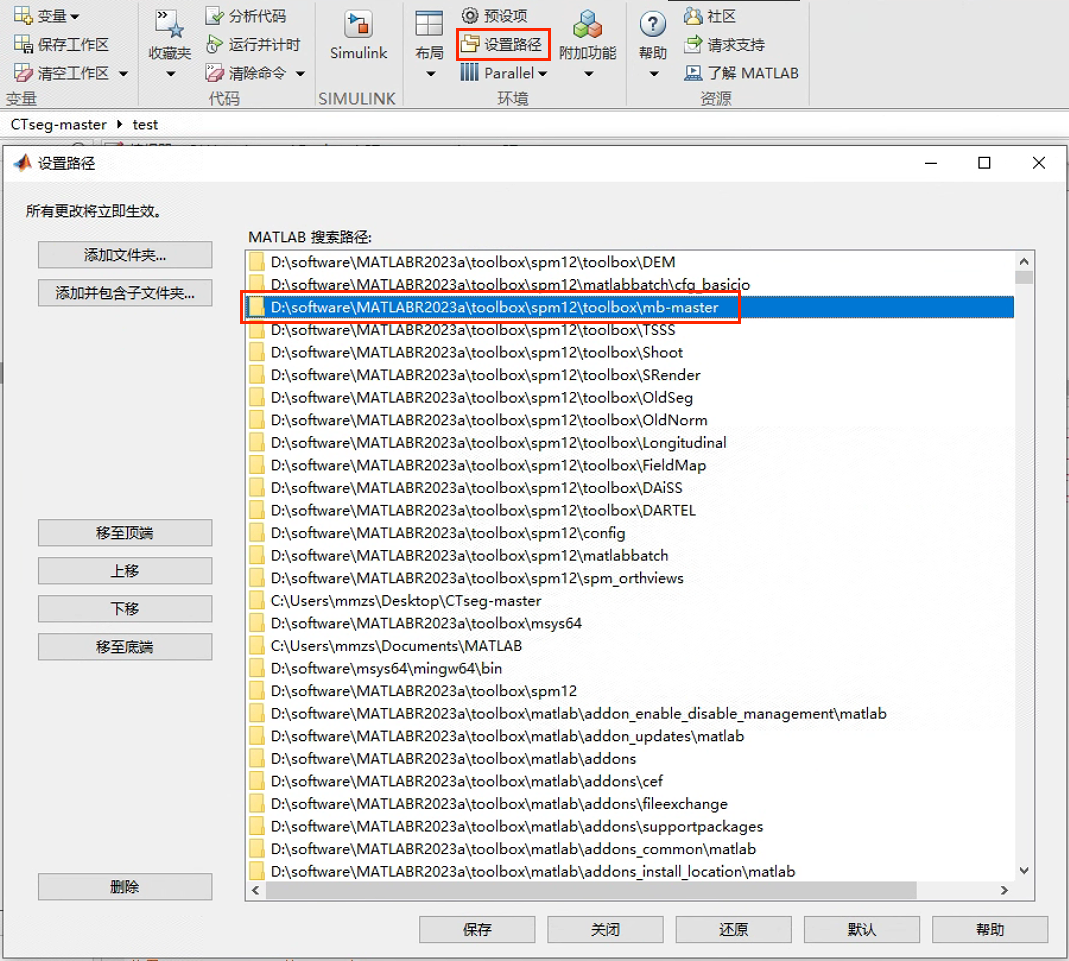
首先我们先把下载的 mb-master 压缩包解压,然后复制到 Matlab -> toolbox ->spm12 -> toolbox 中,记得在 Matlab 中设置路径,这是需要添加到环境路径中的,否则运行不了~
然后打开 MS ,按照维基百科页面输入以下代码:
pacman -Syu
pacman -Su
pacman -S --needed base-devel mingw-w64-x86_64-toolchain
这是在下载一些需要的包,然后下载慢或者网络不好的话,可以换源下载,这里我换的是清华源,代码如下:
sed -i "s#https\?://mirror.msys2.org/#https://mirrors.tuna.tsinghua.edu.cn/msys2/#g" /etc/pacman.d/mirrorlist*
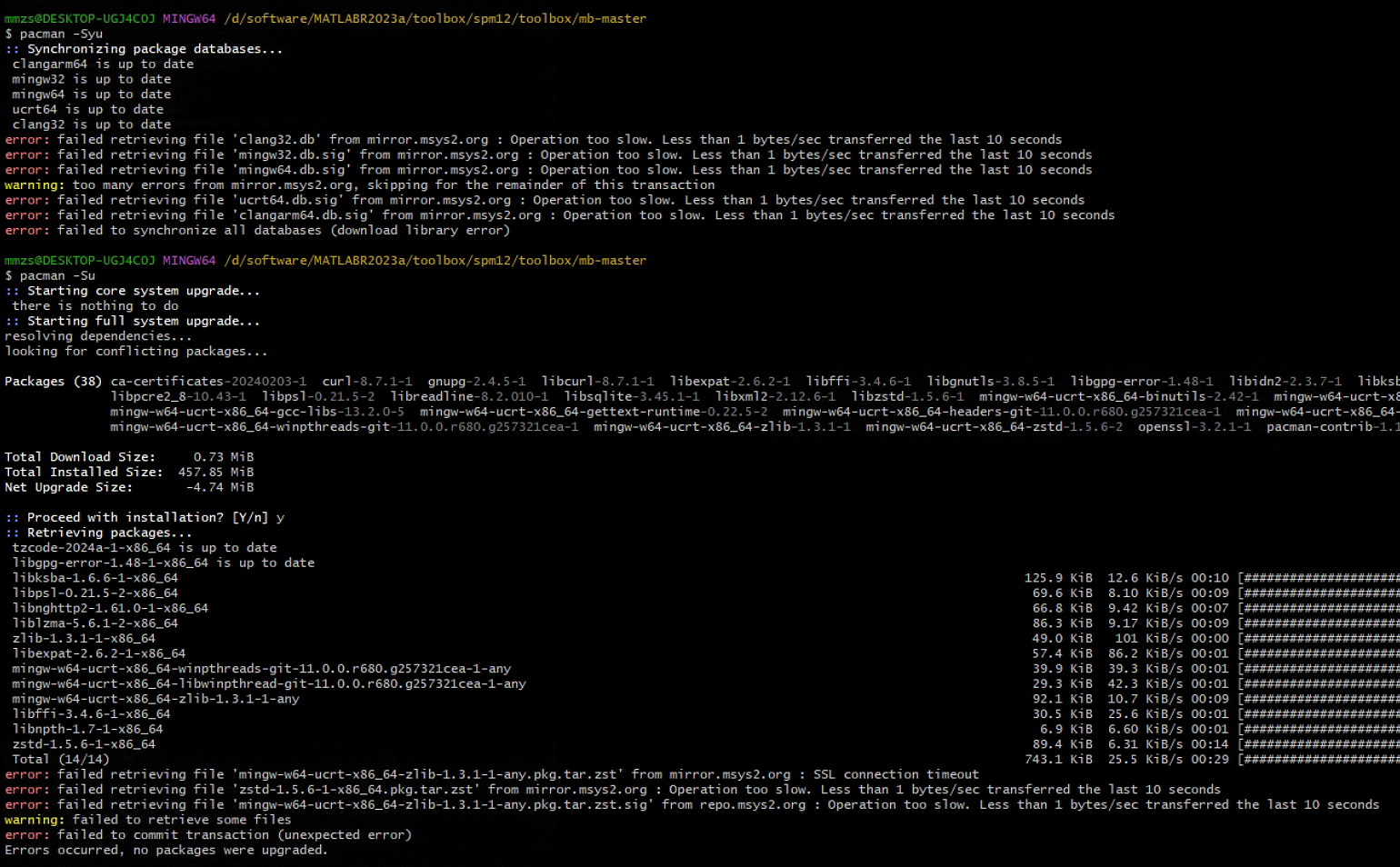
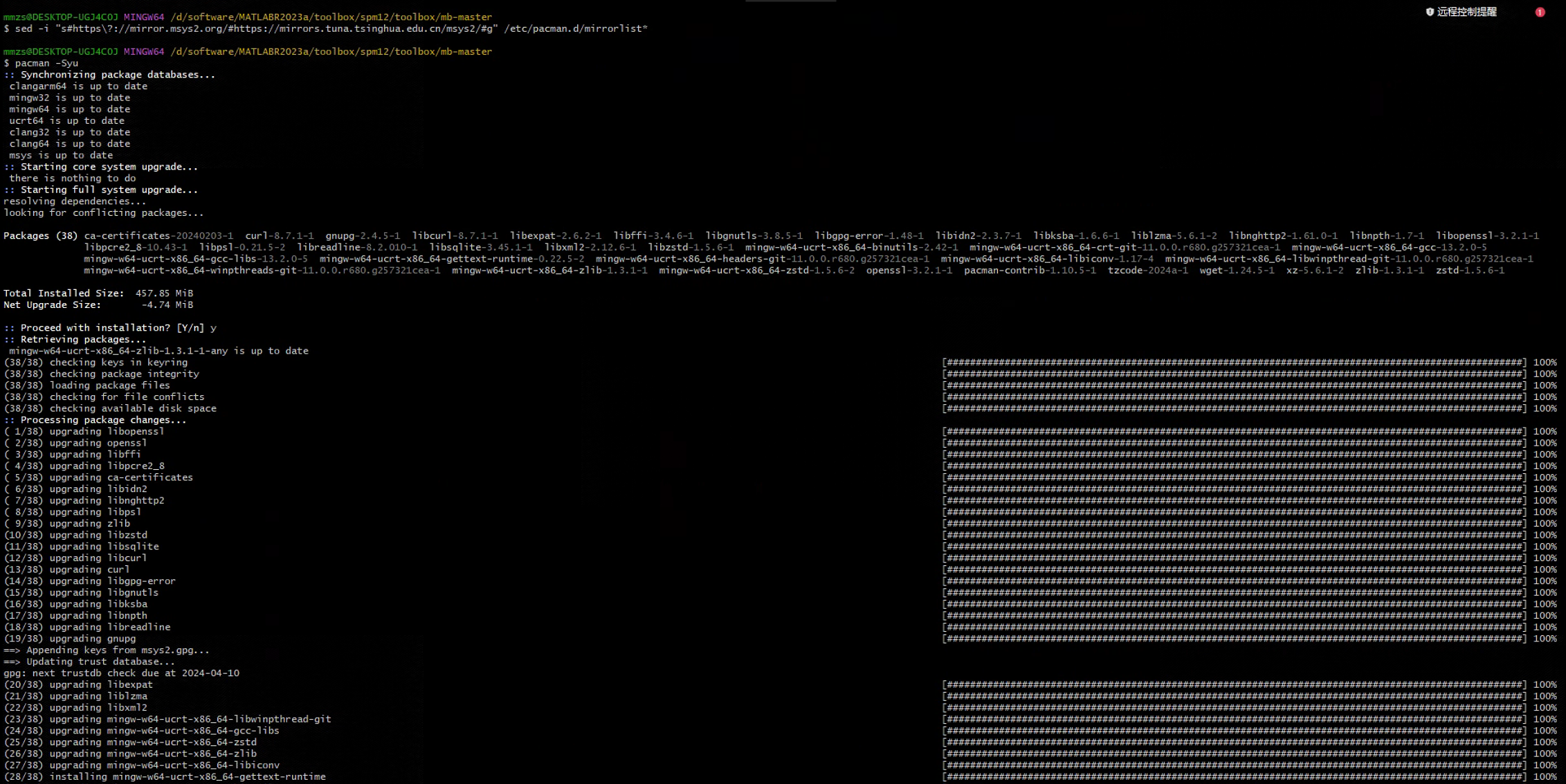
换完源之后就快很多啦~~~
之后去 Matlab 输入以下命令:
setenv('MW_MINGW64_LOC', '自己的msys64\mingw64地址')
mex -setup
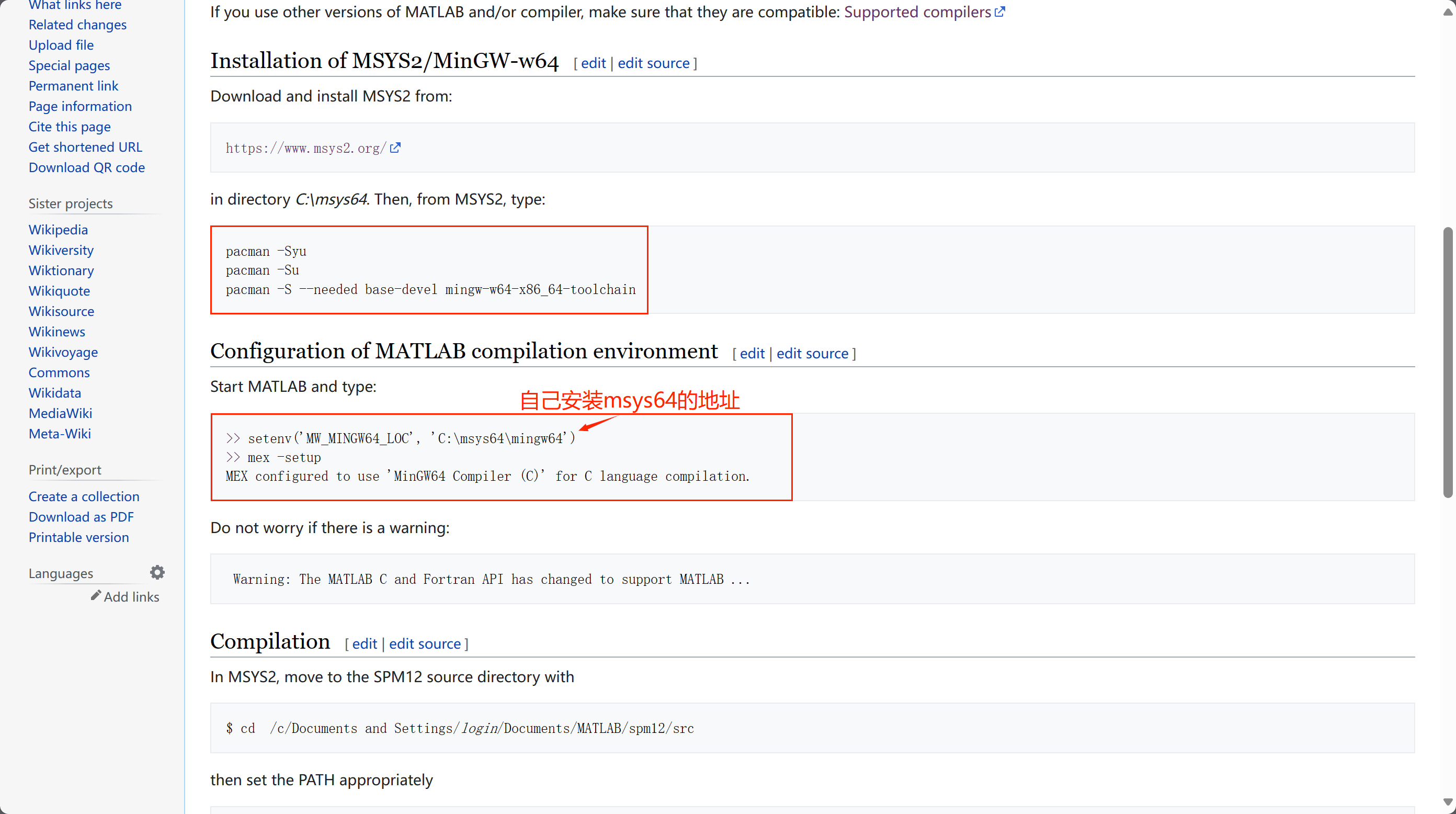
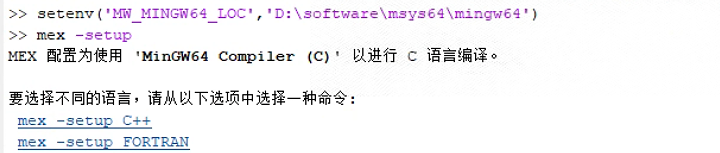
这样 MEX 就配置好了,就可以进行后续文件夹里面的 .c 文件的编译了。
然后根据页面,在 MS 中按要求输入以下代码:
cd 你自己下载的Matlab的位置/MATLAB/spm12/src
PATH=/你自己Matlab的位置/MATLAB/R2018b/bin/win64:${PATH}
$ export MW_MINGW64_LOC=/自己msysm64的位置/msys64/mingw64/
$ PATH=/自己msysm64的位置/msys64/mingw64/bin/:${PATH}
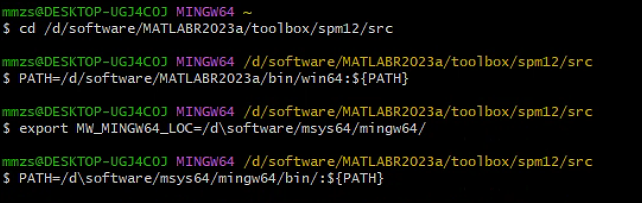
这是添加环境路径。
之后,下一步是需要输入以下代码:
make distclean
make && make install
make external-distclean
make external && make external-install
因为 Windows 没有 make 命令,所以需要自己安装, Linux 系统是不需要这一步的,直接 make 就行了~
但是这里我会一直报错,说找不到这个平台,弄了好久这里 /(ㄒoㄒ)/~~
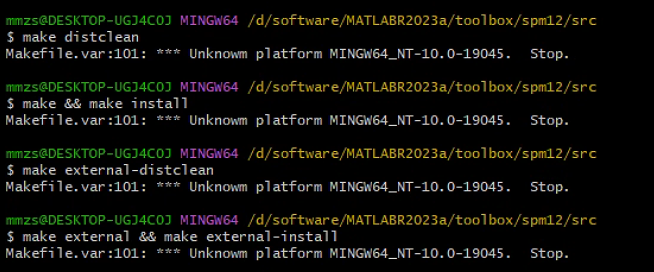
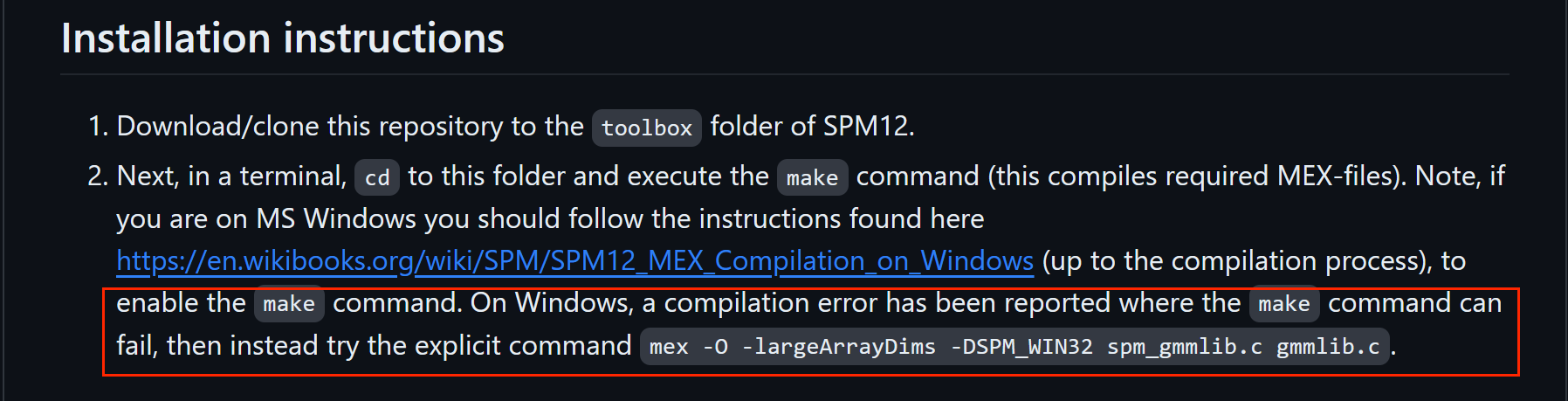
然后我们回到 Multi-Brain toolbox 的 Github ,里面有行代码特别重要!!!
mex -O -largeArrayDims -DSPM_WIN32 spm_gmmlib.c gmmlib.c
就是这个,他也说了,如果在 Windows 系统下报错就使用这行代码!
为啥我弄了好久呢,因为我用浏览器自带的翻译功能给他翻译了,然后这行代码就变成了这样:
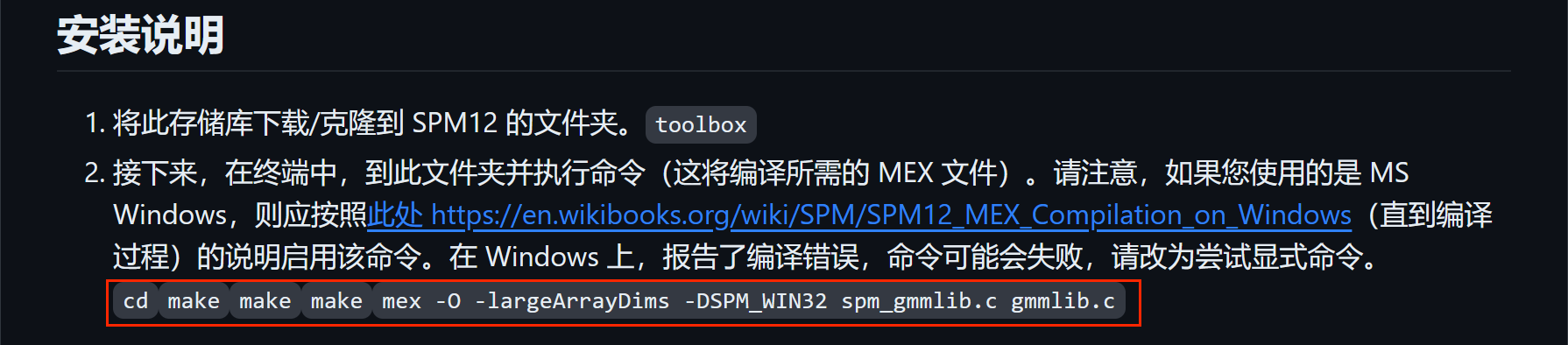
我是怎么输都不对呀!!!(╯▔皿▔)╯ 当时只是觉得这代码好奇怪呀,也没有细想了 /(ㄒoㄒ)/~~
在这里,以我自身的教训提醒大家,一定不要翻译,就算你翻译完后再显示原文也是不对的!!!可以重新进入一下!!!
我们去 Matlab 中使用这行命令就好啦!!!注意,要到含有 .c 的文件里里面使用这行代码哦,否则会报错!
spm_gmmlib.c gmmlib.c 这两个 .c 文件在你 Matlab 里面之前复制到 spm12 的 mb-master 文件夹中
Matlab地址\MATLABR2023a\toolbox\spm12\toolbox\mb-master
mex -O -largeArrayDims -DSPM_WIN32 spm_gmmlib.c gmmlib.c

这样就解析完成啦!!!
然后打开我们之前下载的 CTseg 包,解压后在 Matlab 中打开,记得先添加到路径中!
因为我们只需要用到归一化,所以我们使用的是示例一
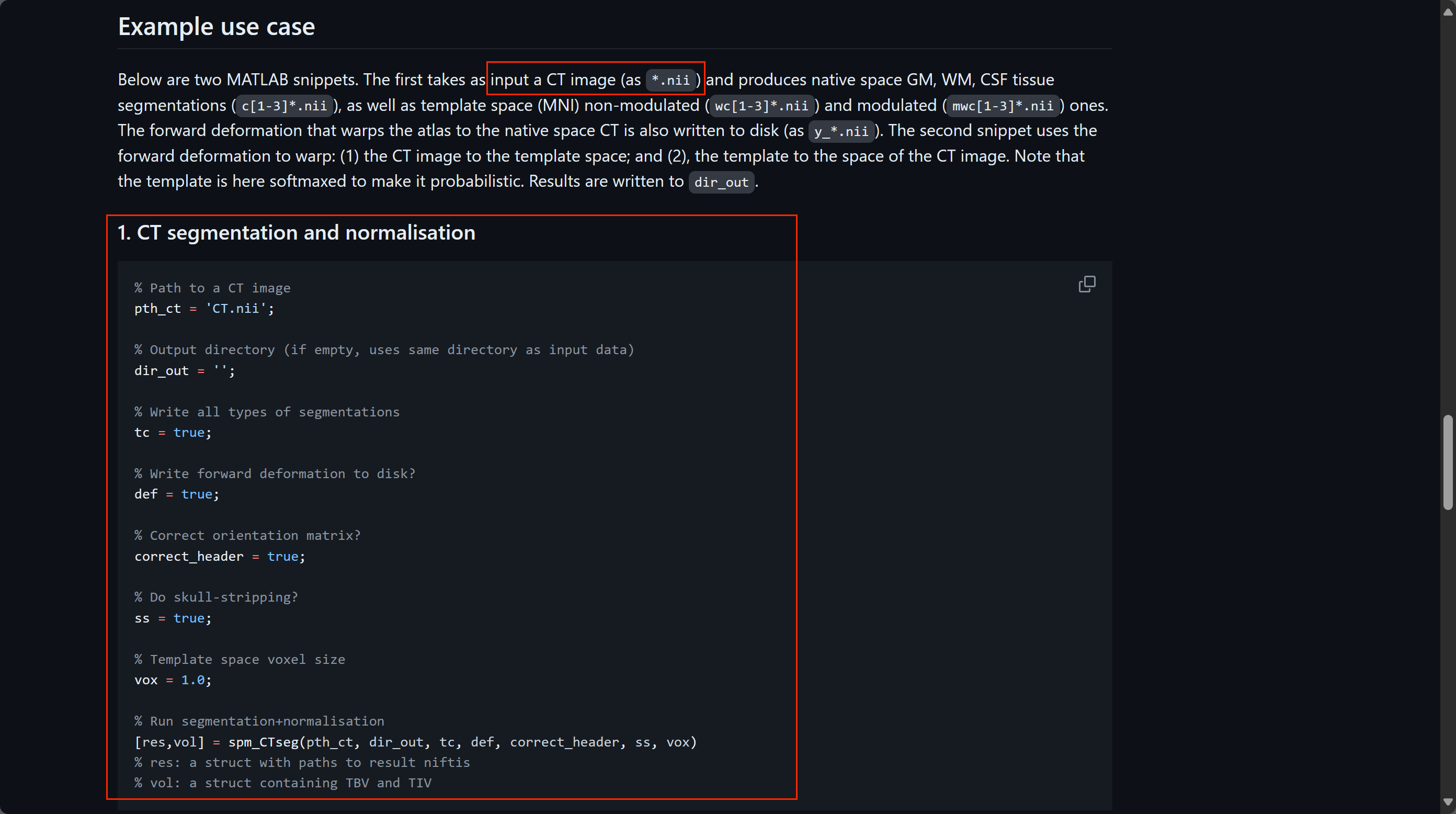
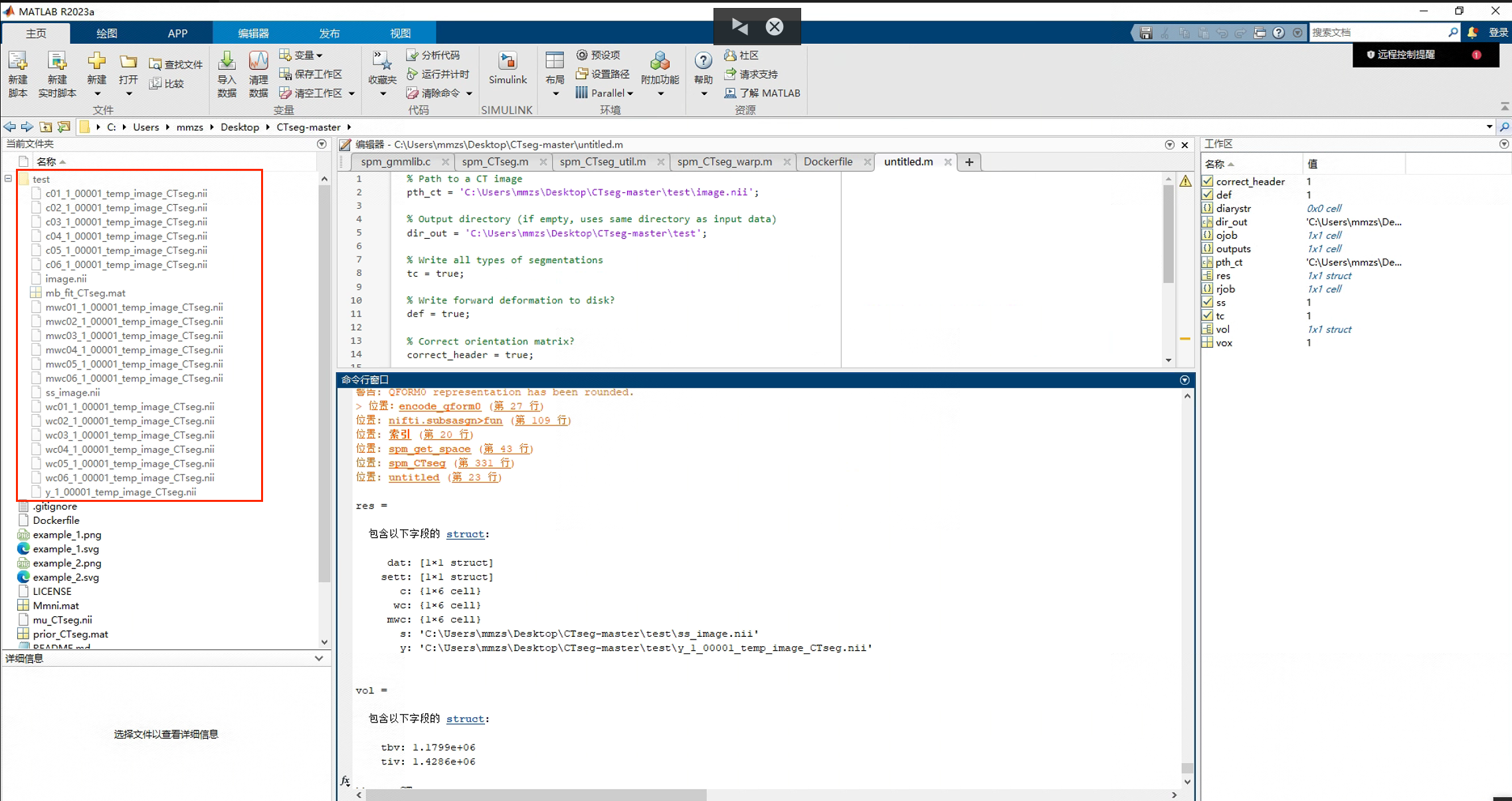
首先在 Matlab 中打开 CTseg-master,然后新建一个 .m 文件,在新建的文件里把示例一的代码复制粘贴,把地址换成自己的地址就可以啦!
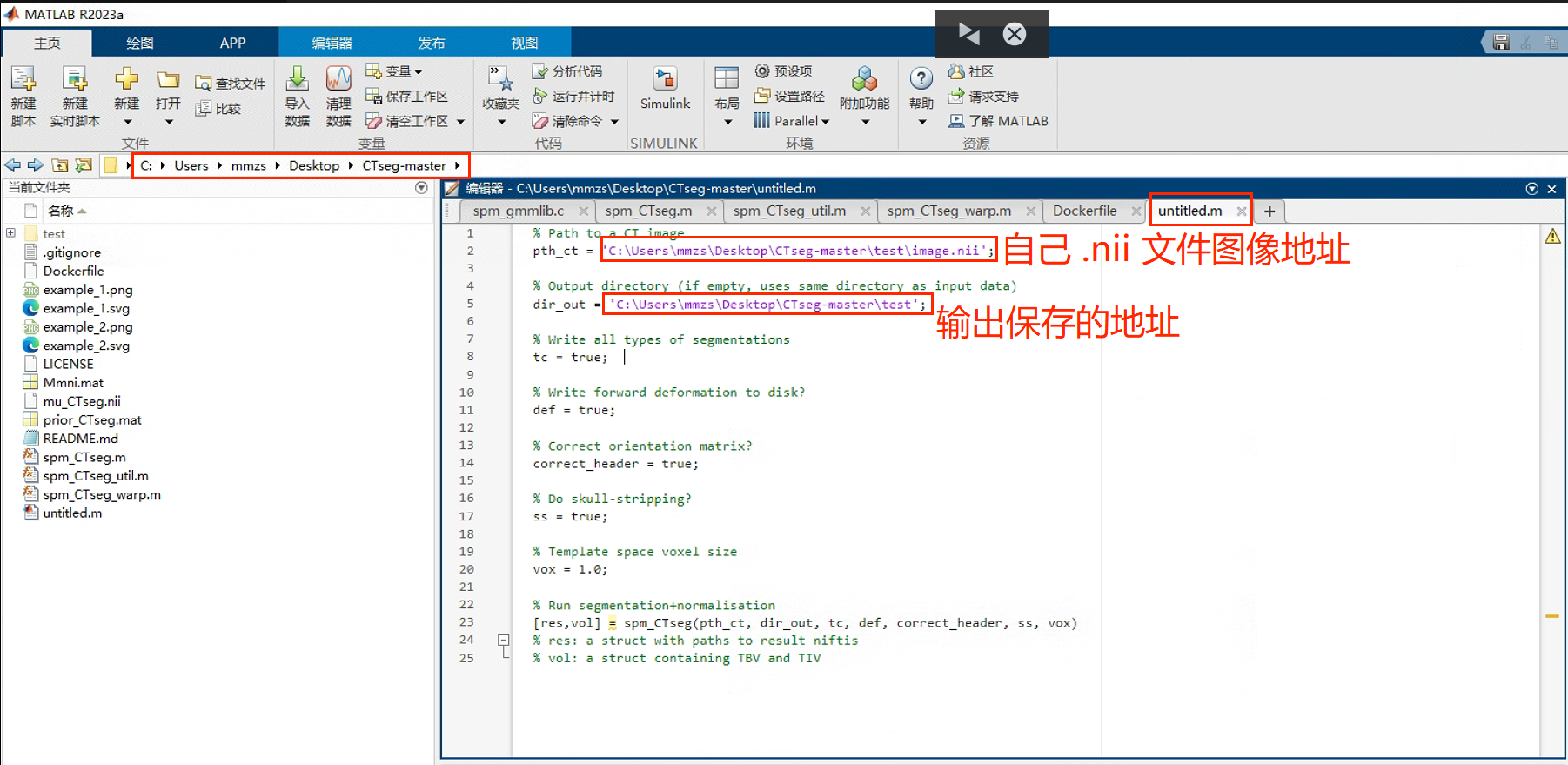
然后运行这个新建的 .m 文件就可以啦!
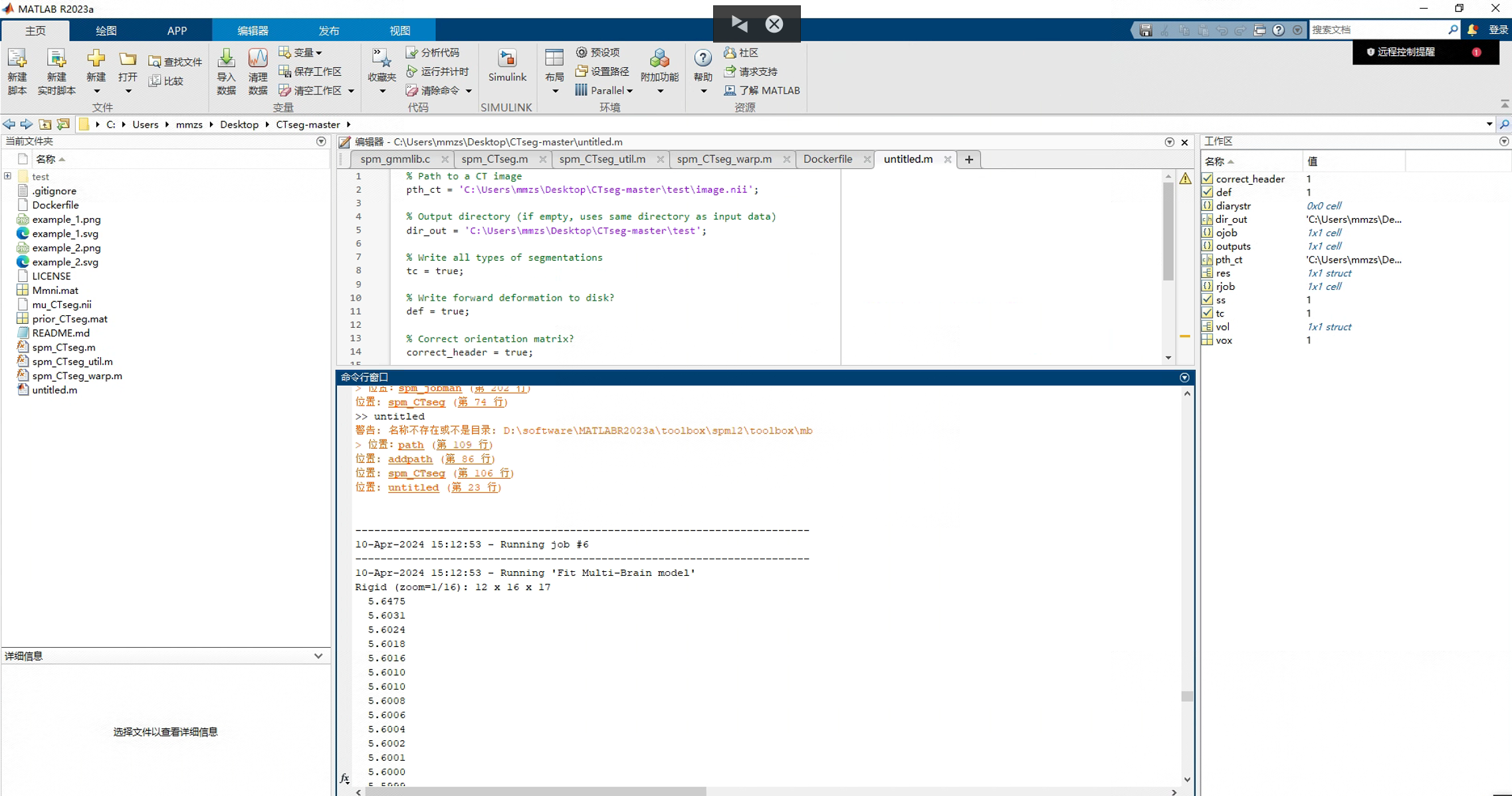
因为他这个代码里面会要下载一个 model.zip 文件,如果你网络不好的话,最好先下载下来,否则会报错,显示下载超时。
下载地址:https://www.dropbox.com/s/qjdqavysgqqhyzc/model.zip?dl=1
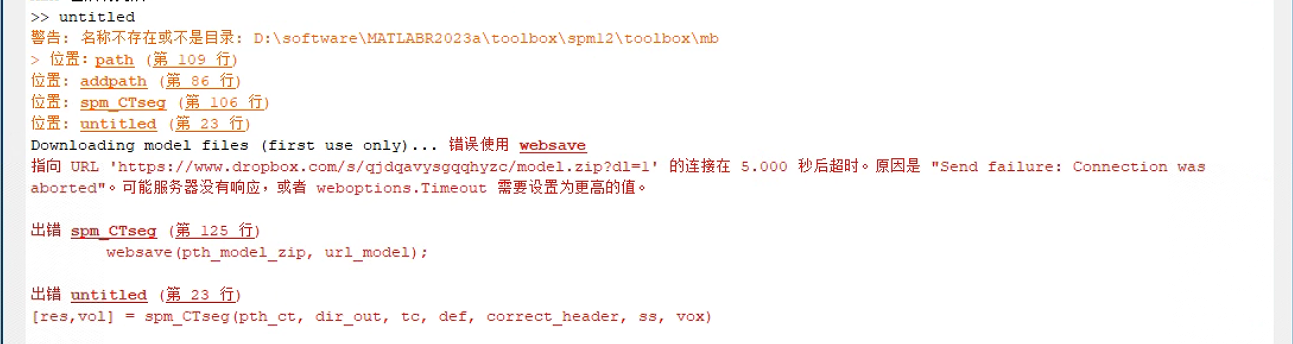
下载完不用解压,直接放到 CTseg-master 文件夹下就可以了
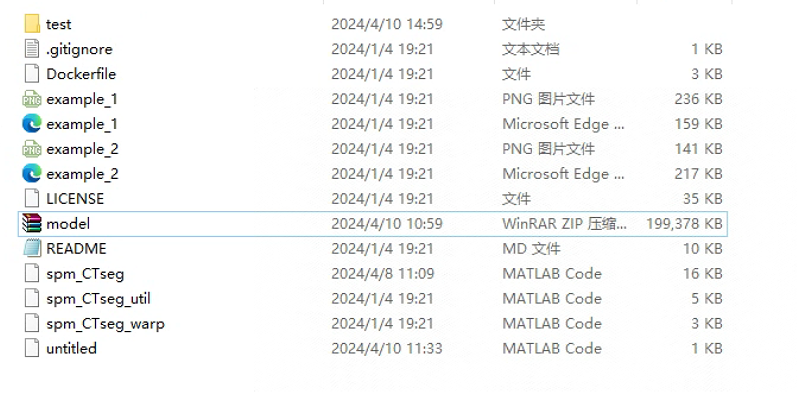
结束之后会生成这么多 .nii 文件, ss_ 是剥离颅骨的, mwc 是归一化和调制的(分的好细,分别是白质、黑质、脑脊液、骨、软组织和背景)
最后,有一个小Tips
Tips:在最后运行的时候如果出现错误的话,就重头把安装步骤再弄一遍,多试几次就好了~~~


 浙公网安备 33010602011771号
浙公网安备 33010602011771号