易基因:Nat Cell Biol/IF19.1:ChIP-seq+WGBS等分析揭示植物REM转录因子构建DNA甲基化谱的分子机制
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
近日,浙江大学/加州大学洛杉矶分校吴忠寿研究员为第一作者,加州大学洛杉矶分校Steven E. Jacobsen院士为通讯作者,在《Nature Cell Biology》(IF19.1/Q1)发表题为“REM transcription factors and GDE1 shape the DNA methylation landscape through the recruitment of RNA polymerase IV transcription complexes”的研究论文,揭示了植物生殖分生组织(REM)转录因子和GDE1蛋白在招募Pol Ⅳ复合体以产生siRNA和指导特定基因组位点DNA甲基化中的关键分子机制。
研究综合运用染色质免疫共沉淀测序(ChIP-seq)、全基因组重亚硫酸盐测序(WGBS)、小RNA测序(sRNA-seq)、IP-MS质谱等分析,在拟南芥中首次鉴定出一条全新RNA介导DNA甲基化(RdDM)通路调控机制:生殖分生组织(REM)家族特异性表达的转录因子(VDD、VAL、REM12、REM13)能够与GDE1蛋白协作,通过识别基因组上特定DNA 的motif序列(CLSY3/CLSY4 motif 1),将植物特有的RNA聚合酶IV(Pol IV)转录复合体精准招募到靶位点(雌性生殖组织中的siren位点)。Pol IV招募导致序列特异性24-nt siRNA产生,进而通过经典的RdDM通路建立特定区域DNA甲基化。本研究揭示了特异性转录因子与siRNA产生及DNA甲基化调控之间的直接关联,为表观遗传模式的遗传调控提供了新见解。

英文标题:REM transcription factors and GDE1 shape the DNA methylation landscape through the recruitment of RNA polymerase IV transcription complexes
中文标题:REM转录因子和GDE1通过招募RNA聚合酶IV转录复合体,构建DNA甲基化谱
发表时间:2025年6月27日
发表期刊:Nature Cell Biology
影响因子:IF19.1/Q1
技术平台:ChIP-seq、WGBS、RNA-seq等(易基因金牌技术)
DOI:10.1038/s41556-025-01691-0
易小结
本研究鉴定出不同于“表观遗传模式主要基于染色质特征(如H3K9甲基化)自我维持”的经典模式,发现了序列特异性转录因子直接编码DNA甲基化图谱的新机制,为理解植物发育与环境应答中表观遗传模式的时空特异性编程提供了关键线索。
该研究基于WGBS与ChIP-seq(易基因金牌技术)的联合分析,完整呈现“转录因子结合→复合体招募→siRNA产生→DNA甲基化建立”因果图谱,为跨物种表观基因组学研究提供可参考的技术框架,并为作物杂交育种的甲基化模式提供了方法学基础。
易基因相关拓展性产品案例
项目文章|Plant Physiol:郑州果树所王力荣团队ChIP-seq等揭示桃树需冷量和芽休眠调控的关键基因
项目文章 | Nature子刊:ChIP-seq等揭示c-di-AMP与DasR互作以调控细菌生长、发育和抗生素合成
项目文章|Plant Cell Environ:WGBS+RNA-seq助力揭示植物不定根再生的DNA甲基化调控机制
项目文章|DNA甲基化与转录组联合分析助力揭示花青素生物合成调控柑橘果实品质的表观新机制
项目文章|NG (IF29):颠覆认知!深圳仙湖植物园刘阳团队WGBS及超级泛基因组分析揭示苔藓植物基因家族比维管植物更丰富
项目文章|WGBS+RNA-seq揭示AtSAMS通过DNA甲基化和乙烯信号通路协同调控植物花器官发育的表观遗传机制
研究方法
(1)植物材料和突变体构建
转基因植物构建:Col-0拟南芥为对照,构建带有FLAG或Myc标签的十几种蛋白过表达转基因拟南芥株系。
突变体材料:
gde1-1(SALKseq_10069.1):GDE1基因T-DNA插入缺失突变体
clsy3-1(SALK_040366)和clsy4-1(SALK_003876):Pol IV招募因子双突变体
rem46/val/rem12三突变体:通过CRISPR-Cas9系统同时敲除三个串联重复的REM基因(REM46、VAL、REM12),产生部分功能丧失
rem8-cr:REM8基因CRISPR敲除突变体
(2)蛋白互作与定位分析
免疫沉淀-质谱(IP-MS):鉴定GDE1或VDD的互作蛋白。
蛋白质印迹与Co-IP:验证IP-MS结果,并更灵敏检测复合体组分间的互作。
(3)多组学综合分析
ChIP-seq:约2g花组织,对GDE1-3FLAG、CLSY3-9myc、CLSY4-9myc、Pol IV-9myc、VDD/VAL/REM13/REM19/REM22/REM8-9myc、VDD-3FLAG等12种蛋白标签抗体进行ChIP-seq,绘制全基因组蛋白结合图谱。
WGBS:计算各序列背景下(CG, CHG, CHH)的C/(C+T)比率,获得全基因组甲基化水平。直接量化gde1-1和clsy3clsy4突变体中Siren位点的CHG和CHH甲基化丧失,从功能层面证实REM-GDE1-CLSY3/4通路对非CG甲基化的依赖性。
小RNA测序(sRNA-seq):胚珠组织或花药样本,鉴定各突变体中的siRNA位点。
DNA亲和纯化测序(DAP-seq):野生型花蕾样本,验证体外直接结合能力和序列特异性。
结果图形
(1)GDE1编码一个位于RdDM位点的未表征蛋白
研究团队通过IP-MS筛选与RdDM通路相关蛋白MORC7互作的未知蛋白,鉴定并命名新蛋白为GDE1 (GENETICS DETERMINES EPIGENETICS 1)。利用ChIP-seq技术分析其在花组织的基因组结合位点,发现GDE1与RdDM通路的多个核心组分中强共定位(图1a-c)。motif(基序)富集分析显示,GDE1的结合peak中存在一个保守DNA基序,该基序与之前报道的CLSY3偏好基序一致,且GDE1表达模式也与主要在花组织中表达的CLSY3/4高度相似(图1d-f)。上述结果共同确立GDE1为RdDM通路中一个未知的、与CLSY3/4协同作用的核定位蛋白,其结合位点具有序列特异性。
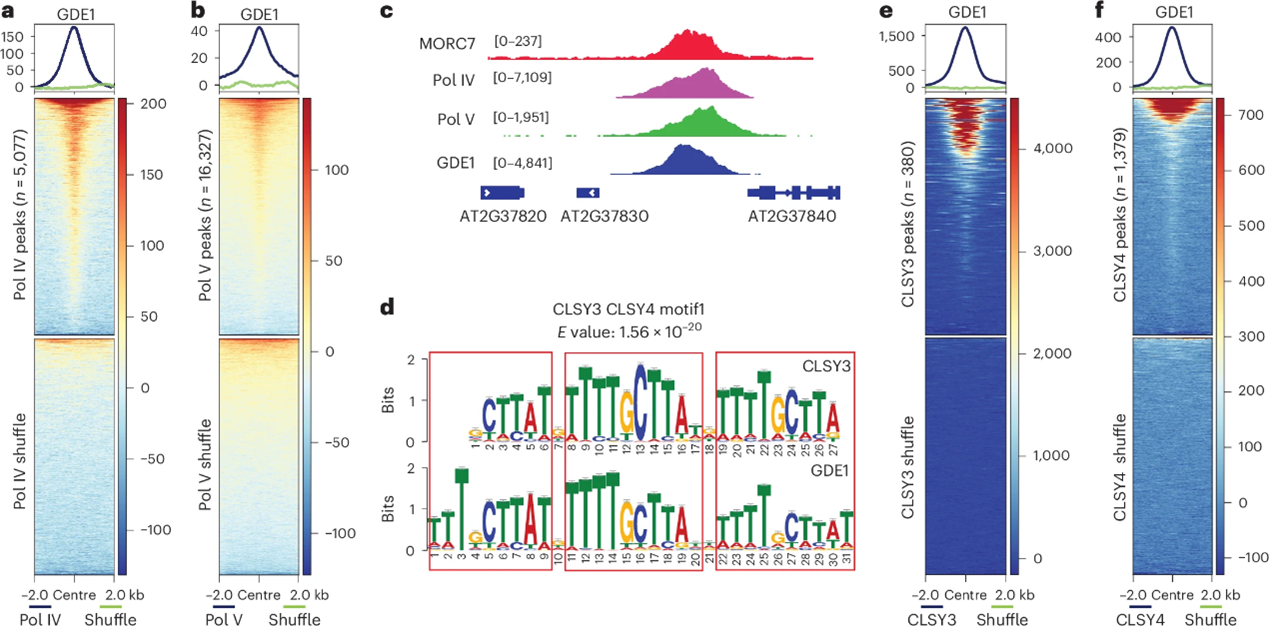
图1:GDE1是一个RdDM蛋白,并与Pol IV招募因子CLSY3/4共定位。
a-b. Meta图和热图显示GDE1的ChIP-seq信号在Pol IV(n=5,077)(a)和Pol V(n=16,327)(b)结合位点上的富集情况。
c. MORC7、Pol IV、Pol V和GDE1 ChIP-seq在代表性位点的截图。
d. TomTom分析显示CLSY3与GDE1结合motif的相似性。
e-f. Meta图和热图显示GDE1的ChIP-seq信号在CLSY3(n=380)(e)和CLSY4(n=1,368)(f)结合位点上的富集情况。
(2)GDE1指导CLSY3/4-Pol IV复合体调控siRNA产生
研究团队首先在胚珠组织中鉴定了753个CLSY3/4依赖的siRNA位点,且在clsy3clsy4双突变体中表达下调。为验证GDE1是否影响siRNA产生,研究团队在gde1-1突变体胚珠组织中进行了sRNA-seq。研究将依赖于CLSY3/4的siRNA位点根据在gde1-1中变化分为三组:Group 1(57%,siRNA减少)、Group 2(35%,siRNA增加)和Group 3(8%,不变)(图2a-c)。
关键的发现是,CLSY3/4和Pol IV的ChIP-seq信号在Group 1位点(常染色质区域,siren位点)上,于gde1-1背景下显著减弱;在Group 2位点(异染色质区域)上,却出现了增强(图2d)。GDE1自身的ChIP-seq信号在Group 1位点最强,Group 2位点最弱(图2e)。功能回补实验表明,GDE1-3FLAG转基因可完全恢复Group 1位点的siRNA水平(图2b-c),证实表型特异性。上述结果说明GDE1优先调控常染色质区的siRNA产生,并与异染色质区Pol IV招募存在竞争关系。
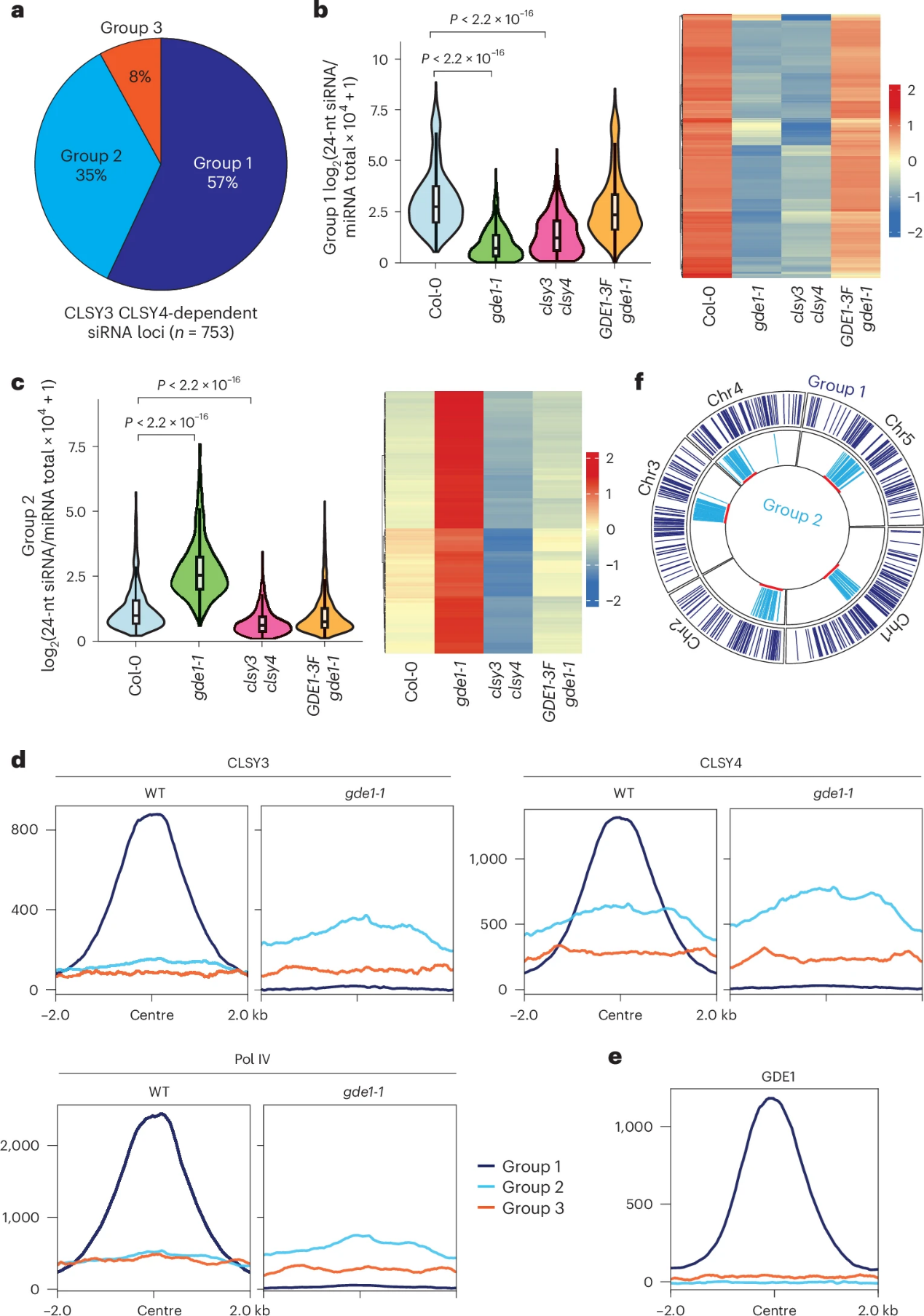
图2:GDE1定位于CLSY3/CLSY4依赖性siRNA位点子集以促进siRNA产生,而GDE1缺失会将CLSY3/4–Pol IV复合体重定位至其他位点。
- 饼图显示三组CLSY3/CLSY4依赖性siRNA位点的百分比分布。
b-c. 小提琴图和热图分别显示CLSY3/CLSY4依赖性位点中Group 1 (b) (n=429) 和Group 2 (c) (n=262) 的24-nt siRNA水平。
d. Meta图显示在野生型(WT)和gde1-1背景下,CLSY3/4和Pol IV的ChIP-seq信号在三组CLSY3/CLSY4依赖性位点上的富集情况。
e. Meta图展示了GDE1的ChIP-seq信号在三组CLSY3/CLSY4依赖性位点上的富集情况。
f. 圈图显示Group 1和2中CLSY3/CLSY4依赖性siRNA在所有五条染色体上的富集分布。
(3)GDE1与REM家族转录因子共定位
为探究GDE1如何发挥作用,研究人员对GDE1-3FLAG进行IP-MS分析,意外发现未富集到CLSY3/4-Pol IV组分,反而鉴定出多个REM转录因子家族成员(VDD、VAL、REM46等)(图3a)。随后,研究团队对VDD、VAL、REM19、REM22、REM8进行9myc标签ChIP-seq分析证实,四个REM转录因子(VDD, VAL, REM12, REM13)在Group 1位点存在显著且相似的富集(图3b-e、g),而另一些REM因子(如REM8)主要富集于Group 2位点(图3b-d、h)。进一步的IP-MS实验从VDD下拉获得了GDE1、CLSY3、Pol IV组分,证实了在体内存在由这些因子形成的复合体(图3f)。这些结果表明,REM家族转录因子(VDD/VAL/REM12/REM13/REM19为主,REM8/REM22为辅)与GDE1协同作用,负责识别特定DNA序列并与CLSY3/4-Pol IV复合体结合。
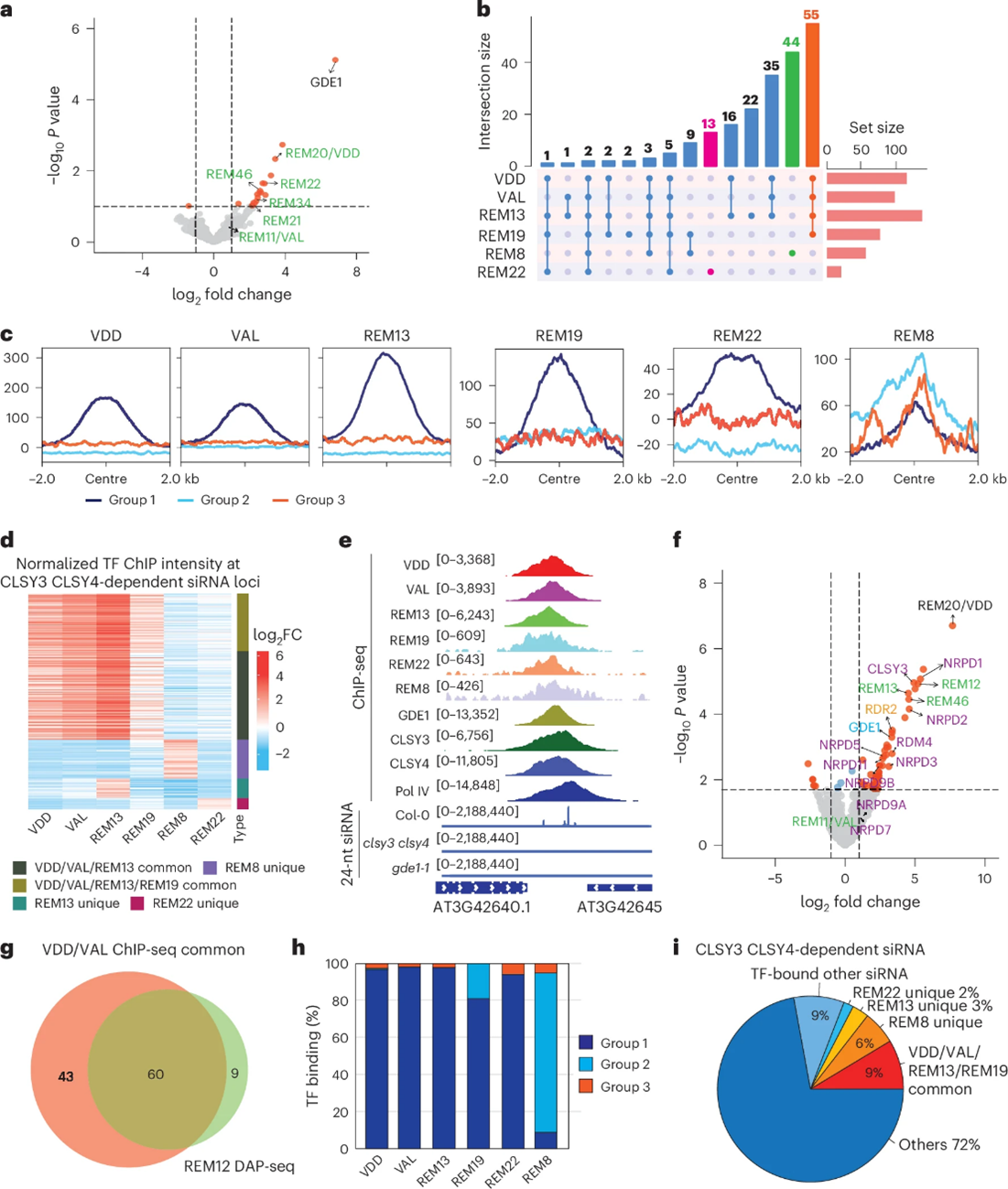
图3:REM转录因子在CLSY3/CLSY4依赖性siRNA位点与GDE1及Pol IV复合体结合
(4)GDE1在Siren位点调控siRNA产生与DNA甲基化
胚珠组织中主要的siRNA在Siren位点,86%的Siren位点含有CLSY3CLSY4 motif 1。研究团队发现VDD/VAL/REM13等REM转录因子和GDE1、CLSY3/4、Pol IV在该位点的ChIP-seq信号显著富集(图4a-b)。具体而言,在gde1-1突变体的胚珠组织中,siren位点的24-nt siRNA水平急剧下降,表型与clsy3clsy4双突变体相似。
研究人员应用WGBS分析了其DNA甲基化水平的变化,发现由于siRNA缺失,Siren位点的CHH和CHG甲基化水平在gde1-1中显著降低,模式与clsy3clsy4一致(图4d),这直接将GDE1介导的上游招募事件与最终的表观遗传修饰(DNA甲基化)降低相关联,完成了从“序列识别”到“甲基化缺失”的表型链验证。
ChIP-seq比较揭示,gde1-1中CLSY3/4和Pol IV在Siren位点的占用丢失,但在非Siren位点无变化,说明GDE1特异性稳定Pol IV复合体于Siren位点。
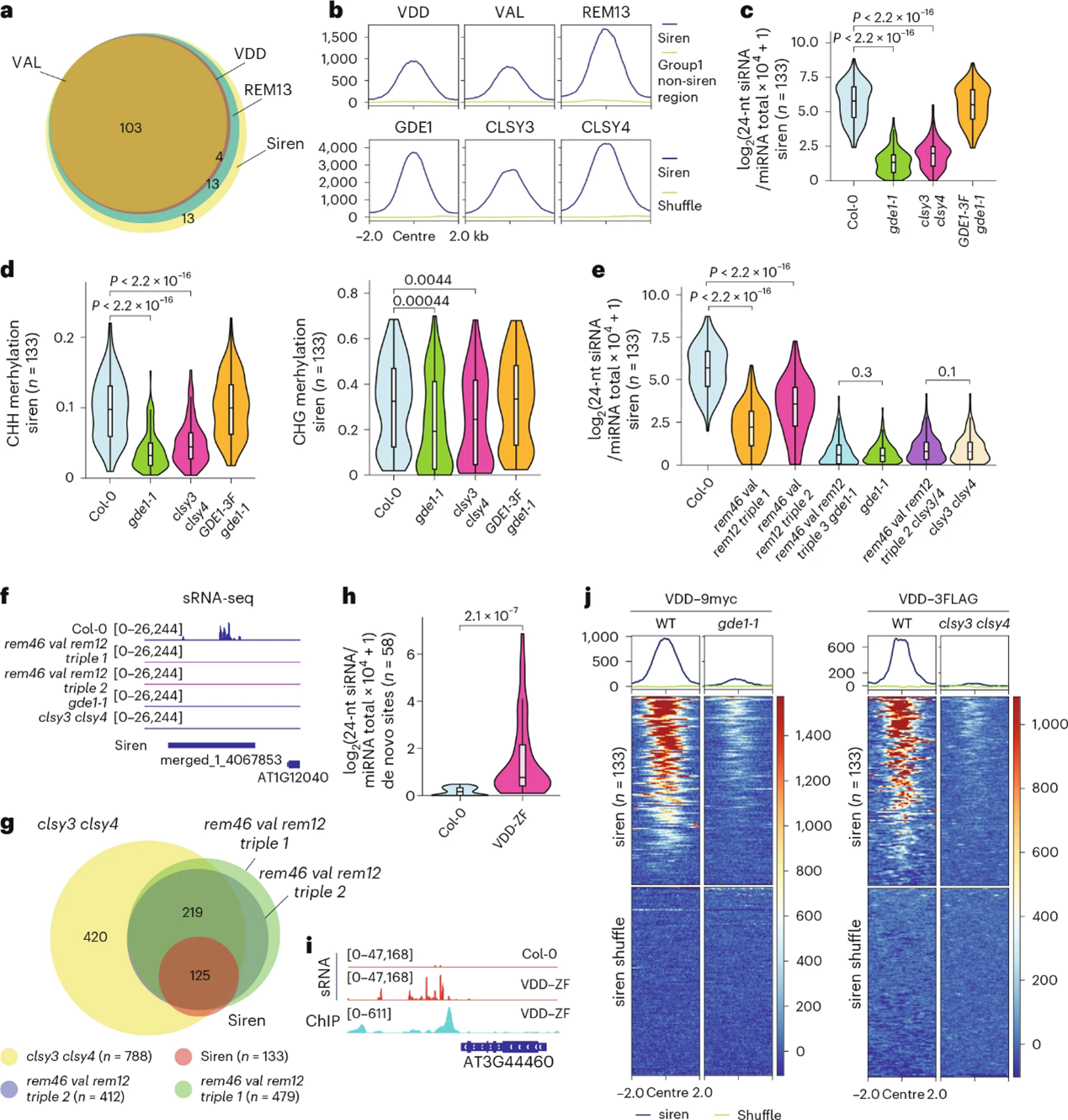
图4:VDD/VAL/REM13–GDE1–CLSY3/4复合体定位于siren位点以产生siRNA。
(5)REM转录因子正向调控siRNA产生
为直接验证REM转录因子的功能,研究团队构建了vdd单突变以及rem46/val/rem12三突变体。sRNA-seq分析显示,三突变体胚珠组织中Siren位点siRNA显著下调(图4e-f),与clsy3 clsy4双突变的表型在Group 1有相当程度的重合(图4g),表明GDE1依赖REM转录因子以介导siRNA产生。
(6)RBHG结构域在GDE1功能中至关重要
通过蛋白结构预测,研究团队发现GDE1的一个α螺旋区域(包含保守的RBHG基序)被预测与REM转录因子形成广泛的氢键和π键相互作用(图5a-c)。为验证这一发现,研究团队将此区域内三个关键氨基酸位点(E251A/Y252A/Y255A)突变后进行转基因功能回补实验。结果表明,虽然该突变体GDE1表达正常,但无法完全挽救gde1-1突变体中siren位点的siRNA缺失表型(图5d),Siren位点siRNA恢复程度显著低于野生型GDE1。上述结果表明该α螺旋结构域对于GDE1与REM转录因子的互作及其功能不可或缺,为复合体组装提供了分子层面的结构证据。
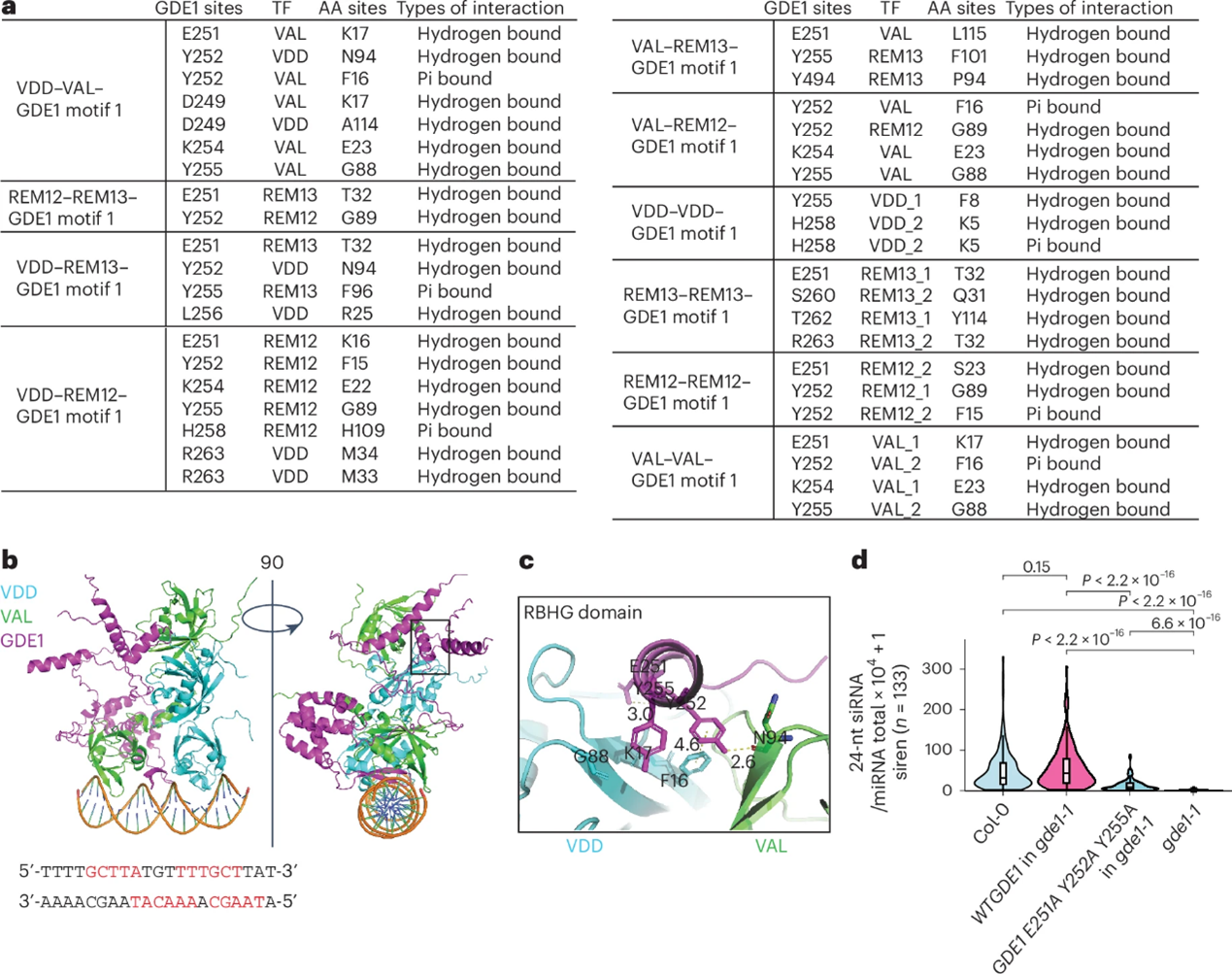
图5:GDE1的RBHG结构域对siRNA产生至关重要
(7)REM-GDE1介导CLSY3/4-Pol IV复合体的单向转录
对motif分析发现,CLSY3/4 motif 1由2-3个重复的TTTTGCTTAT序列组成。基因组中具有这种结构并产生显著ChIP信号的位点很少(二重复约48个,三重复约38个)。ChIP-seq分析显示,所有复合体组分都在这些位点富集,且REM-GDE1的peak中心位于基序中心,而CLSY3/4和Pol IV的峰则偏离中心约200-300 bp,且仅位于基序某一侧(图6a、c)。这表明REM-GDE1结合在DNA基序中心,然后招募CLSY3/4-Pol IV复合体,Pol IV随后将从基序一侧或另一侧起始单向转录,产生pre-siRNA(图6d)。DAP-seq实验进一步在体外验证VDD-VAL-GDE1复合体只结合单碱基间隔的基序(图6b)。
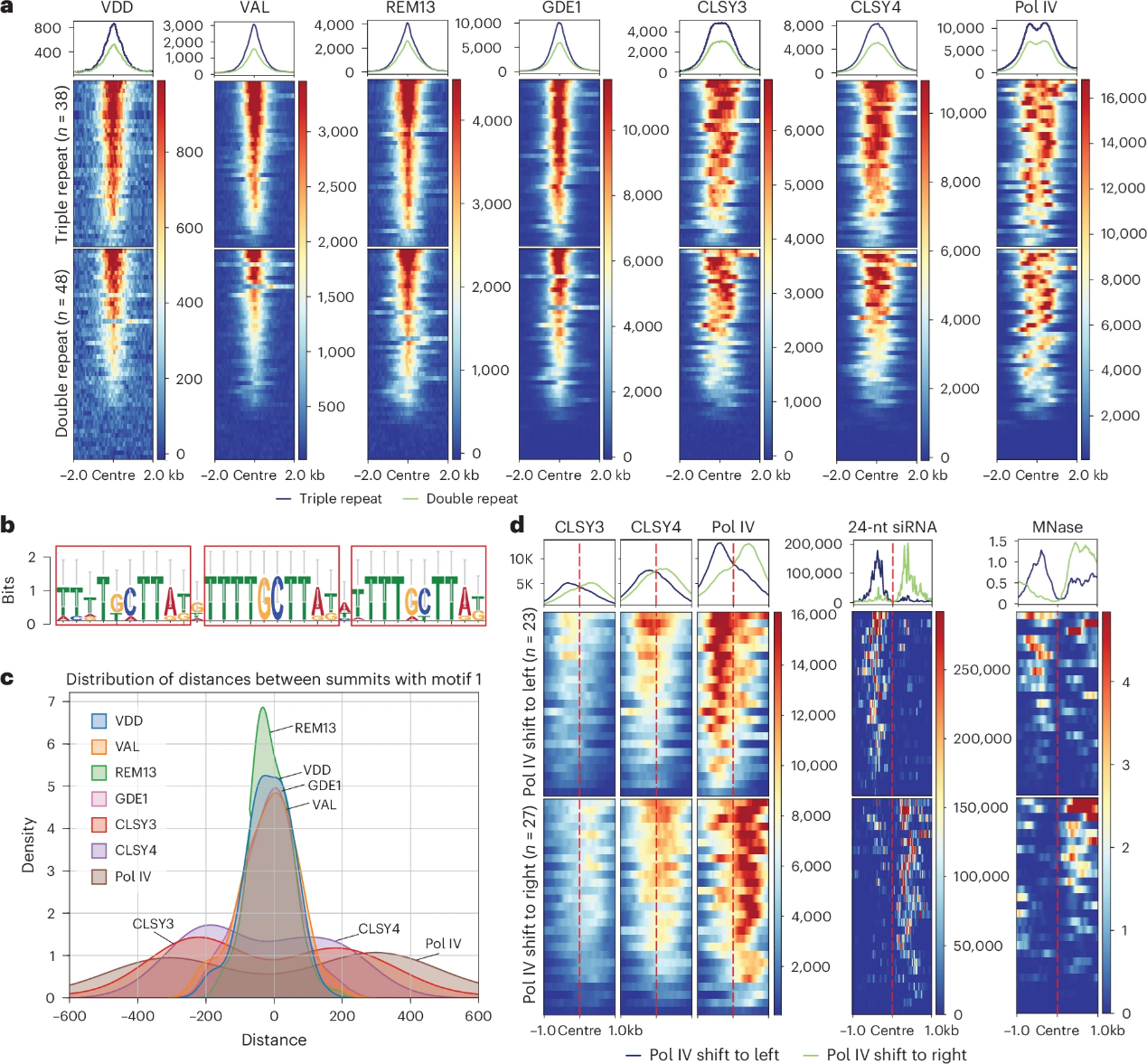
图6:Pol IV转录复合体识别CLSY3/4 motif 1并进行单向转录
(8)REM8识别CLSY3/4 Motif 2以在GDE1缺失时介导siRNA产生
此前结果显示Group 2位点在gde1-1中siRNA上调。研究团队聚焦在Group 2富集的REM8,发现其不结合CLSY3/4 motif 1,而是识别AAGCGGAT三重复序列(命名为CLSY3/4 motif 2)(图7b)。在gde1-1中,REM8结合的非Siren区域siRNA水平升高,且CLSY3/4和Pol IV占用增强(图7a),表明GDE1缺失释放的Pol IV复合体被REM8重定位至Group 2位点。
此外,CLSY3/4 motif 2位点的siRNA在clsy3clsy4中下降,在gde1-1中上升(图7c),证实这些位点确实受CLSY3/4-Pol IV调控,但招募机制不同于Siren位点。这表明REM8(可能与其他未知因子一起)也介导了对另一类DNA基序的识别和CLSY3/4-Pol IV的招募,是GDE1依赖路径之外的另一种序列特异性招募机制。
值得注意的是,虽然雄性组织中大多数REM转录因子表达量很低,但GDE1表达却非常高。在花药组织中,约一半的CLSY3/4依赖性siRNA位点在gde1-1中也表现出类似下调(图7d-f)。上述发现揭示GDE1在胚珠组织中与REM因子协作,在花药中可能与其他因子协作,实现组织特异性甲基化图谱的精准编程。
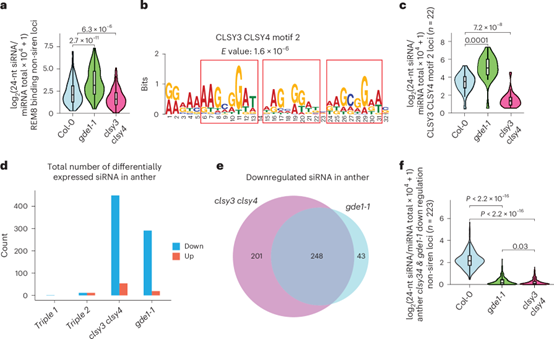
图7:GDE1缺失条件下,REM8将Pol IV复合体重新定位至CLSY34 motif 2位点,且GDE1是花药中siRNA产生必需
结论和启示
本研究首次发现并阐明了一个由生殖特异性REM转录因子与GDE1蛋白协作,通过识别特定DNA基序,直接招募Pol IV转录复合体,从而在特定基因组位点(如siren位点)启动siRNA产生和DNA甲基化的全新遗传和表观遗传关联通路。
ChIP-seq与WGBS在本研究中的重要作用
ChIP-seq通过对12种蛋白的精确定位,绘制了REM-GDE1-CLSY3/4-Pol IV的共定位网络;通过比较野生型与突变体,揭示了复合体的动态重定位;通过peak偏移分析,推断单向转录模型。
WGBS在siRNA产生后,直接单碱基分辨率检测DNA甲基化(CHG/CHH)修饰水平,从功能层面确认该通路的生物学结果。
WGBS与ChIP-seq的联合使用,使得从“蛋白定位”到“功能输出”的因果链条完整闭合,是揭示新机制的黄金标准。
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
|
技术类型 |
起始量 |
特点 |
覆盖度 |
|
常规WGBS |
1μg gDNA |
正常BS建库技术 |
95% |
|
Micro DNA-WGBS |
1-10000个细胞/1ng基因组DNA |
在常规WGBS技术上进行技术改进,使得起始量大大降低,适合珍稀样本的研究 |
95% |
|
scWGBS |
单细胞/1-10个细胞 |
克服了组织内部细胞异质性的影响,可以满足单个细胞层面的课题研究 |
20% |
|
Micro DNA-XRBS |
1ng gDNA、单细胞 |
为Micro DNA-WGBS的简化版本,特别适用于大样本量的珍稀样本DNA甲基化研究 |
20M CG |
参考文献:Wu Z, Xue Y, Wang S, Shih YH, Zhong Z, Feng S, Draper J, Lu A, Hoeke CA, Sha J, Li L, Wohlschlegel J, Wu K, Jacobsen SE. REM transcription factors and GDE1 shape the DNA methylation landscape through the recruitment of RNA polymerase IV transcription complexes. Nat Cell Biol. 2025 Jun 27. doi: 10.1038/s41556-025-01691-0.
相关阅读
项目文章|Sci Adv:WGBS+ChIP-seq揭示DNA甲基化在异基因造血干细胞移植后延迟血小板恢复中的关键作用
项目文章|Plant Cell Environ:WGBS+RNA-seq助力揭示植物不定根再生的DNA甲基化调控机制
项目文章|DNA甲基化与转录组联合分析助力揭示花青素生物合成调控柑橘果实品质的表观新机制
项目文章|JAR (IF13):西农陈玉林团队多组学分析揭示绵羊早期胚胎发育的分子与表观遗传调控机制
项目文章 | 植入前胚胎的全基因组DNA甲基化和转录组分析揭示水牛胚胎基因组激活进展
项目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘细胞DNA甲基化与羟甲基化表观基因组图谱
PNAS:南方科技大学朱健康团队多组学揭示协同调控植物DNA甲基化与Polycomb沉默的表观遗传新机制
NC(IF15.7):华农王应祥/杨存义ChIP-seq等揭示大豆开花时间调控及区域适应性的表观分子机制
PBJ/IF10.5:西农园艺学院DNA甲基化+组蛋白修饰综合分析揭示苹果干旱胁迫的表观基因组调控机制
Genome Biol:ChIP-seq等揭示植物H3K27me3相关转座元件沉默模式及与DNA甲基化动态转换


 浙公网安备 33010602011771号
浙公网安备 33010602011771号