llamafactory使用记录
(1)# 创建Python隔离环境
conda create -n llama_factory python=3.10
conda activate llama_factory
(2)# 安装核心依赖
pip install torch==2.1.2 --index-url https://download.pytorch.org/whl/cu118
pip install git+https://github.com/hiyouga/LLaMA-Factory.git
# 为了安装依赖包,所以直接clone了这个库到本地
cd /data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/
git clone https://github.com/hiyouga/LLaMA-Factory.git # 第一次的时候没有成功,第二次的时候 成功了
cd /data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/
pip install -e ".[torch,metrics,deepspeed,flash-attn,bitsandbytes,vllm]"
#检查是否安装成功
llamafactory-cli version # Welcome to LLaMA Factory, version 0.9.4.dev0 安装成功
(3)数据准备
(3.1)json格式,每个样本包括instruction,input和output
例如:{"instruction": "法语问候翻译", "input": "早上好", "output": "Bonjour"}
在我的研究中,我的instruction都是相同的,prompt设置。input是影像报告,output是希望预测的病理指标。我的output是多个病理指标。所以我的准备格式是这样的
[
{
"instruction": "你是一名专业的医学影像分析助手。请根据患者的影像所见、诊断结论和病灶结构化信息,预测以下四个病理指标的阴性和阳性:\n1. 病理指标A\n2. 病理指标B\n3. 病理指标C\n4. 病理指标D\n请严格按照'指标A:阴性,指标B:阳性,指标C:阴性,指标D:阳性'的格式输出结果。",
"input": "影像所见:肺部可见磨玻璃结节,大小约1.2cm,边界清晰。诊断结论:疑似早期炎症。病灶信息:结节大小1.2cm,密度均匀,无分叶征,无毛刺征。",
"output": "指标A:阴性,指标B:阴性,指标C:阳性,指标D:阴性"
},
{
"instruction": "你是一名专业的医学影像分析助手。请根据患者的影像所见、诊断结论和病灶结构化信息,预测以下四个病理指标的阴性和阳性:\n1. 病理指标A\n2. 病理指标B\n3. 病理指标C\n4. 病理指标D\n请严格按照'指标A:阴性,指标B:阳性,指标C:阴性,指 标D:阳性'的格式输出结果。",
"input": "影像所见:肝脏占位性病变,增强扫描呈快进快出。诊断结论:考虑恶性肿瘤。病灶信息:肿块大小3.5cm,边界不清,有坏死区域。",
"output": "指标A:阳性,指标B:阳性,指标C:阴性,指标D:阳性"
}
]
重要!!!json文件确认好后,文件存放位置放在data文件夹下:/data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/data/XXX.json
同时,该目录下的data_info.json需要同步修改。比如,我的json文件是medical_classification_all_train.json,我的json文件中有instruction,input和output,我的json设置
{
"medical_classification_all_train": { #这里是json文件的名字
"file_name": "medical_classification_all_train.json", # 这里是json文件全称
"columns": {
"prompt": "instruction",
"query": "input",
"response": "output",
"history": ""
}
},
"identity": {
"file_name": "identity.json"
},
}
(3.2)训练配置设置(.yaml)
1).yaml文件的存放路径位于 /data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/examples/train_lora/AAA.yaml
2) .yaml里面包括 model、method、dataset、output、train的设置。
其中,model是下载的大语言模型的位置,我的模型下载放到了RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory_Model/Qwen3-8B这里。关于模型下载,可以用魔塔,这里贴个图,需要下什么模型,就去官网找,然后他也会告诉你安装的代码
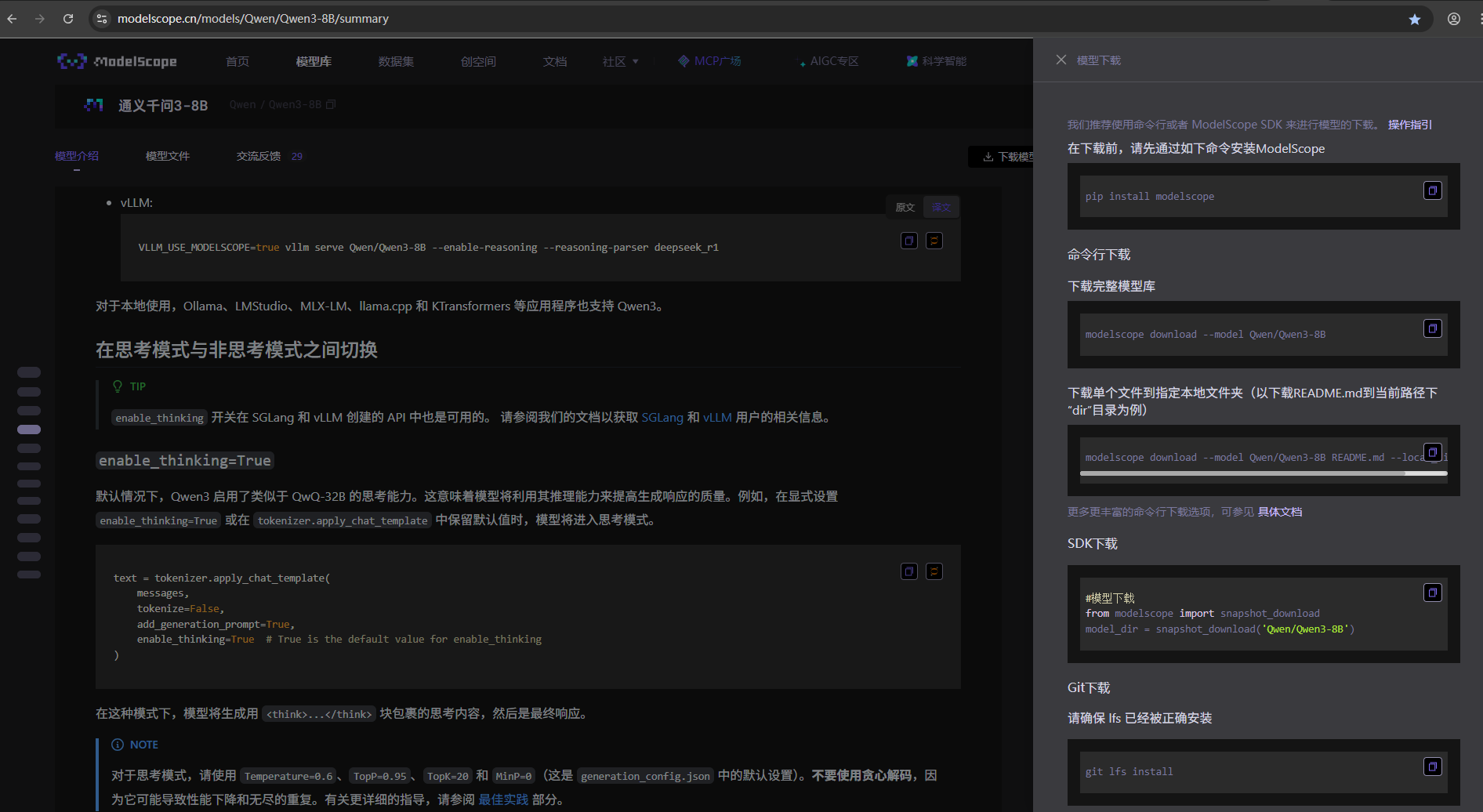
所以,在yaml的model设置中,关于model_name_or_path,就填写大语言模型所在的路径
另外,tmplate的设置,可以看LLAMA-Factory官网的配置信息
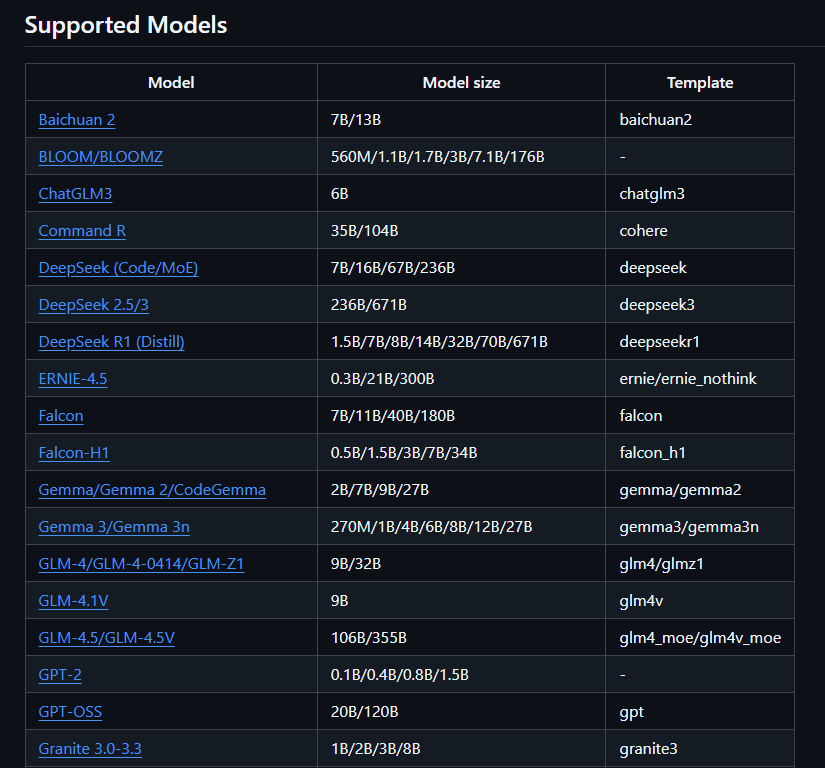
method常规设置即可
dataset设置里面,dataset的设置就是训练集json文件的名称(注意不要.json的后缀)
output设置里面,output_dir是你自定义的模型保存的位置
设置好后,可以试着开始训练
(4)模型训练
llamafactory-cli train "/data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/examples/train_lora/llama3_lora_hcc_mvi.yaml"
注意,需要cd到"/data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/"目录再开始训练
(5)模型推理
模型推理稍微复杂一点,因为对于验证集和测试集患者想批量处理,所以在linux系统环境下,需要先激活API,然后运行批处理的.py文件。
首先,激活API可以在terminal直接输入:(注意,都是在LLamafactory的目录下运行)
CUDA_VISIBLE_DEVICES=2 API_PORT=8000 llamafactory-cli api --model_name_or_path /data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory_Model/Qwen3-8B/ --adapter_name_or_path /data1/wangfang_data/RC_Collaboratives/project/fang.wang03/0_Models/LLaMA-Factory/LLaMA-Factory/models/Abdominal_QWen/Batch1/checkpoint-10000/ --template qwen3 --finetuning_type lora
激活后,写进行批处理的python文件,.py文件中,核心是配置信息,链接到API
# 配置
API_URL = "http://localhost:8000/v1/chat/completions"
HEADERS = {"Content-Type": "application/json"}
def query_model(prompt):
"""向模型发送查询"""
data = {
"model": "default",
"messages": [
{"role": "user", "content": prompt}
],
"temperature": 0.1,
"max_tokens": 2048
}
try:
response = requests.post(API_URL, headers=HEADERS, json=data, timeout=60)
if response.status_code == 200:
return response.json()["choices"][0]["message"]["content"]
else:
print(f"请求失败,状态码: {response.status_code}, 响应内容: {response.text}")
return f"Error: {response.status_code}"
except Exception as e:
print(f"请求异常: {str(e)}")
return f"Exception: {str(e)}"
其他的就是读取json文件,设置好自己的输入、输出,然后在LLM这个环境下,运行这个.py文件即可。
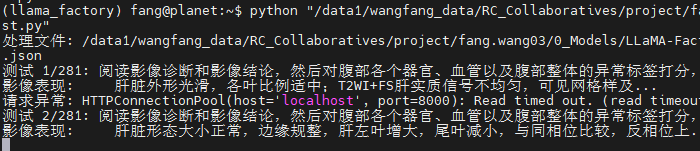
又有新的注意了,模型没训练完的时候,直接去某个epoch下做测试,会报错,显示connection refused。我把训练的模型关掉,再测就正常了。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号