【机器学习】GMM和EM算法
机器学习算法-GMM和EM算法
1. GMM模型
聚类问题是一个经典的无监督任务,其目标是将 \(N\) 个 \(D\) 维数据 \(\{\bf{x}_i\}_{i=1}^N\) 分成\(K\)个簇,使得每个簇中的样本尽可能相似。GMM算法对数据分布做了一些假设:
- 第\(k\)个簇数据点服从正态分布,即\(\mathbf{x}|\mathbf{z}\sim \mathcal{N}(\mu_k, \Sigma_k)\)
- 簇标签\(\mathbf{z}\sim \mathrm{Dir}(\alpha)\)
其中簇标签\(\mathbf{z}\)满足\(\mathbf{z}\in \{0, 1\}^K, \quad \sum_{k=1}^K z_k = 1,\quad p(z_k=1)=\alpha_k\)。根据条件概率和边缘分布可得:
代入相关的概率分布得到:
需要求解的模型参数为\(\{\alpha_k, \mu_k, \sigma_k^2\}_{k=1}^K\)共计\(K\times(1+D+D^2)\)个参数。
2. GMM模型参数求解
为了估计GMM分布中的参数,采用MLE或者MAP即可实现。以MLE为例,目标函数为:
2.1 参数\(\alpha\)的求解
参数\(\alpha\)需要满足\(\sum_k \alpha_k=1\)的条件,拉格朗日函数为:
对梯度等式两边同乘上\(\alpha_k\)并对k求和得到\(\lambda = -\frac{1}{N}\)。定义模型对数据\(\mathbf{x}_n\)的响应\(\gamma_{nk}\)为:
得到参数\(\alpha\)的估计为:
2.2 参数\(\mu\)和\(\Sigma\)的求解
参数\(\mu\)和\(\Sigma\)没有任何限制,对似然函数直接求导即可得到:
代入正态分布的表达式\(\mathcal{N}(\mathbf{x}|\mu_k, \Sigma_k) = \frac{1}{(2\pi)^{\frac{D}{2}}|\Sigma|^{\frac{1}{2}}}\exp\{-\frac{1}{2}(\mathbf{x}-\mu_k)^T\Sigma_k^{-1}(\mathbf{x}-\mu_k)\}\)得到:
根据矩阵行列式的拉普拉斯展开\(|A| = \sum_{j=1}^M a_{ij}M_{ij}\)和逆矩阵计算公式\(A^{-1} = \frac{1}{|A|}A^*\)得到:
将上式和\(\frac{\partial \mathbf{x}^T A \mathbf{x}}{\partial A} = \mathbf{x}\mathbf{x}^T\)带入到梯度表达式得到:
将上述求导结果代入到似然函数的梯度中得到:
3. GMM算法的实现
根据模型参数求解的结果可知,在更新参数时需要知道\(\gamma_{nk}\),而计算\(\gamma_{nk}\)又需要知道模型参数,陷入到了一个循环中。GMM采用了EM算法更新这两部分参数:
- E-step:固定模型,计算\(\gamma_{nk}\)
- M-step:运用极大似然估计更新模型参数
重复迭代E-step和M-step直到模型收敛。
3.1 gmm类的定义和实现
gmm类需要记录数据的维度和簇的数目,为了后续的方便,将样本数量也作为初始化参数记录;然后生成了三个模型参数,为了简化将协方差矩阵限定为对角矩阵。方法包含了两个主要方法和三个辅助函数:
- train:训练函数,根据数据集计算出模型参数
- step:单步的训练,包含了E-step和M-step
- _prob:多元高斯密度计算
- _log_likelihood:计算当前模型下的对数似然
- cluster:预测函数,将数据集进行聚类
class gmm():
def __init__(self, dims, K, N):
self.dims = dims
self.K = K
self.N = N
self.mu = np.random.randn(K, dims)
self.sigma = np.ones((K, dims))
self.alpha = np.ones(K)/self.K
def train(self, X, maxIter, verbos):
for i in range(maxIter):
self.step(X)
if i % verbos == 0:
logH = self._log_likelihod(X)
print("In loop: %d, log likelihood : %f" %(i, logH))
def step(self, X):
# E-step
p = self._prob(X) # N x K matrix
alpha_p = self.alpha * p
Z = np.sum(alpha_p, axis = 1, keepdims=True)
gamma = alpha_p / Z
N_k = np.sum(gamma, axis = 0)
# M-step
self.alpha = N_k / self.N
tmp_mu = np.zeros_like(self.mu)
tmp_sigma = np.zeros_like(self.sigma)
for k in range(self.K):
tmp_mu[k] = np.average(X, axis = 0, weights = gamma[:,k])
tmp_sigma[k] = np.average((X - self.mu[k])**2, axis = 0, weights = gamma[:, k])
self.mu = tmp_mu
self.sigma = tmp_sigma
def _log_likelihod(self, X):
n_points, n_clusters = len(X), self.K
pdfs = (self.alpha*self._prob(X)).sum(axis = 1)
return np.mean(np.log(pdfs))
def _prob(self, X):
n_points, n_clusters = len(X), self.K
pdfs = np.zeros(((n_points, n_clusters)))
for i in range(n_clusters):
pdfs[:, i] = multivariate_normal.pdf(X, self.mu[i], np.diag(self.sigma[i]))
return pdfs
def cluster(self, X):
p = self._prob(X)
labels = np.argmax(p, axis = -1)
return labels
3.2 测试
导入相关包
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.patches import Ellipse
from scipy.stats import multivariate_normal
from GMM import *
生成样本数据并可视化
#%% 生成数据
num1, mu1, var1 = 400, [0.5, 0.5], [1, 3]
X1 = np.random.multivariate_normal(mu1, np.diag(var1), num1)
num2, mu2, var2 = 600, [5.5, 2.5], [2, 2]
X2 = np.random.multivariate_normal(mu2, np.diag(var2), num2)
num3, mu3, var3 = 1000, [1, 7], [6, 2]
X3 = np.random.multivariate_normal(mu3, np.diag(var3), num3)
X = np.vstack((X1, X2, X3))
#%% 可视化数据点
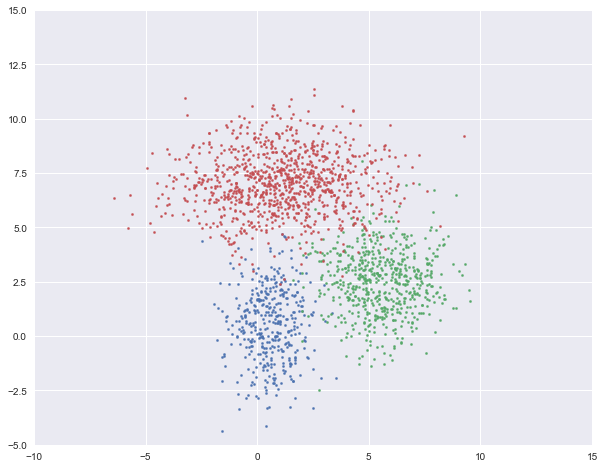
plt.figure(figsize=(10, 8))
plt.axis([-10, 15, -5, 15])
plt.scatter(X1[:, 0], X1[:, 1], s=5)
plt.scatter(X2[:, 0], X2[:, 1], s=5)
plt.scatter(X3[:, 0], X3[:, 1], s=5)
plt.show()
实例化一个模型并训练
model = gmm(X.shape[1], 3, X.shape[0])
max_Iter = 60
verbos = 10
model.train(X, max_Iter, verbos)
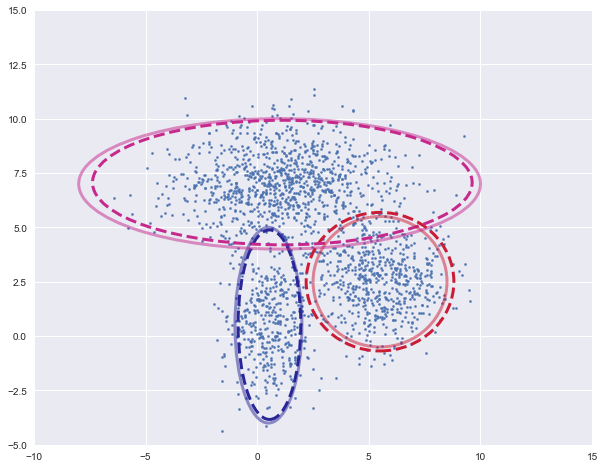
可视化聚类结果,将各个高斯密度函数的等高线画出来,虚线为训练的模型,实线为实际的模型
plot_clusters(X, model.mu, model.sigma, [mu1, mu2, mu3], [var1, var2, var3])
为了实现可视化过程,定义了绘制簇的函数
def plot_clusters(X, Mu, Var, Mu_true=None, Var_true=None):
assert X.shape[1] == 2, "this function can't plot 3D figure"
n_clusters = len(Mu)
# colors = ['r', 'g', 'b']
colors = [0]*n_clusters
for i in range(n_clusters):
colors[i] = randomcolor()
plt.figure(figsize=(10, 8))
plt.axis([-10, 15, -5, 15])
plt.scatter(X[:, 0], X[:, 1], s=5) # markersize = 5
ax = plt.gca() # get current axis
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 3, 'edgecolor': colors[i], 'ls': '--'}
ellipse = Ellipse(Mu[i], 3 * Var[i][0], 3 * Var[i][1], **plot_args)
ax.add_patch(ellipse)
if (Mu_true is not None) & (Var_true is not None):
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 3, 'edgecolor': colors[i], 'alpha': 0.5}
ellipse = Ellipse(Mu_true[i], 3 * Var_true[i][0], 3 * Var_true[i][1], **plot_args)
ax.add_patch(ellipse)
plt.show()
def randomcolor():
colorArr = ['1','2','3','4','5','6','7','8','9','A','B','C','D','E','F']
color = ""
for i in range(6):
color += colorArr[np.random.randint(0,14)]
return "#"+color
结果
4. EM算法
GMM仅仅是EM算法运用的一个特例。在EM算法中,假设数据是由两个部分组成:可观测部分\(\mathbf{x}\)和隐变量\(\mathbf{z}\),两者组合在一起\(\{\mathbf{x}, \mathbf{z}\}\)称为完全数据。在实际应用中,完全数据是无法获取的,根据完全数据进行MLE或者MAP是不现实的,而用完全数据进行估计比用可观测数据对模型进行估计简单。EM算法提供了一种转换办法。
定义记号:
-
数据集:\(\mathbf{X} = \{\mathbf{x}_n\}_{n=1}^N\)
-
隐变量:\(\mathbf{Z} = \{z_n\}_{n=1}^N\)
-
数据分布:\(p(\mathbf{X}) = \prod_{n=1}^N p(\mathbf{x}), \quad p(\mathbf{X}, \mathbf{Z})=\prod_{n=1}^Np(\mathbf{x_n}, \mathbf{z_n})\),可以验证这种定义方式满足条件概率和边缘概率公式:
\[p(\mathbf{X}) = \sum_{\mathbf{Z}}p(\mathbf{X}, \mathbf{Z}), \quad p(\mathbf{X}|\mathbf{Z}) = \frac{p(\mathbf{X}, \mathbf{Z})}{p(\mathbf{Z})} \] 在完全数据上的似然函数为:
\[\mathcal{L} = \log p(\mathbf{X}|\theta) \]
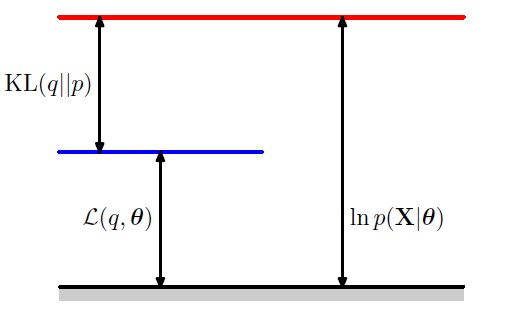
假设隐变量的条件分布为\(q(\mathbf{Z})\),似然函数可以被写作:
根据以上公式可知,可观测数据的似然函数与证据下界之间差了一个KL散度。
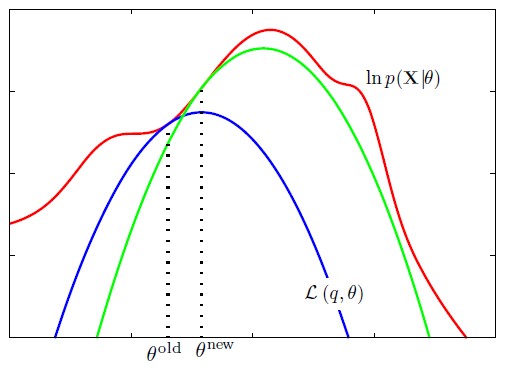
从该角度可以理解EM算法的流程:
- E-step:求解似然函数的下界,该过程是一个关于函数q的变分问题
- M-step:最大化似然函数的下界
该过程需要重复进行,这是因为最大化下界之后,模型的参数会变为\(\theta^{new}\),此时似然函数的下界已经发生了改变,需要重新计算。

EM算法同样适用于MAP框架。在MAP中,目标函数变为:
相比于MLE,MAP多出了一项关于模型参数的似然(数据先验分布似然不影响参数的求解,可以忽略),这一项可以被看作是对模型参数的正则化。
-------------------------------------------
个性签名:只要想起一生中后悔的事,梅花便落满了南山
如果觉得这篇文章对你有小小的帮助的话,记得在右下角点个“推荐”哦,博主在此感谢!


 浙公网安备 33010602011771号
浙公网安备 33010602011771号