详细介绍:【文献分享】HydraRNA:一种基于混合架构的全长 RNA 语言模型
2025-12-08 18:08 tlnshuju 阅读(0) 评论(0) 收藏 举报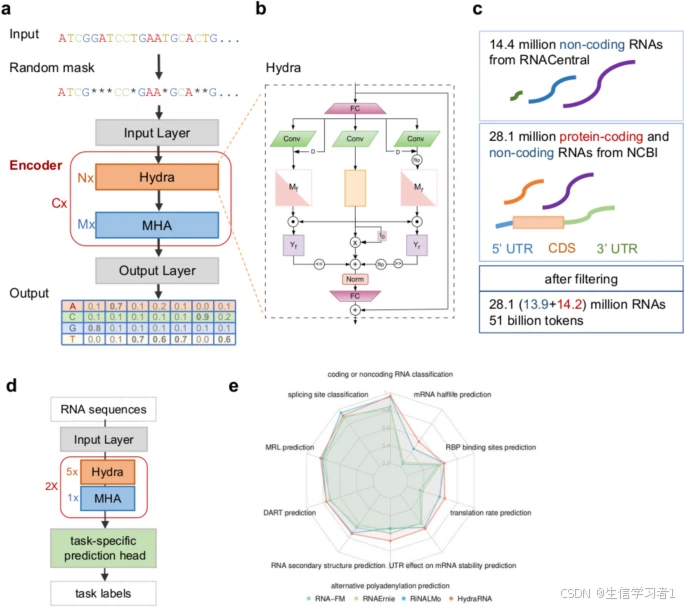
介绍
RNA 是分子生物学中心法则的重要组成部分,在所有细胞过程中都发挥着多种作用。RNA 大规模语言模型(LLM)作为 RNA 研究中强大的方法,有助于解析其复杂的效果和调控网络。然而,之前的 RNA LLM 通常基于 Transformer 模型,并且是基于短片段的非编码 RNA 进行预训练的,这限制了它们的通用适用性。在此,我们介绍了首个全长 RNA 基础模型——HydraRNA,它基于双向状态空间模型和多头注意力机制的混合架构。
HydraRNA 是基于大量的蛋白质编码 mRNA 和非编码 RNA 进行预训练的。尽管其参数最少且使用的 GPU 资源最少,但 HydraRNA 学习到了更优的 RNA 表示,并在各种与 mRNA 相关的任务(包括编码/非编码 RNA 分类、RNA 二级结构预测、RNA 结合蛋白结合位点、剪接和聚腺苷酸化位点、mRNA 稳定性和翻译效率预测)中表现优于现有的基础模型。此外,HydraRNA 能够准确预测突变的影响,并估算不同 mRNA 区域对 RNA 稳定性和翻译的相对贡献。
我们的研究结果表明,此种混合架构在 RNA 语言建模方面优于纯 Transformer 架构。我们预计 HydraRNA 将能够解析 mRNA 的各种特性,加速 mRNA 调控的研究,并有助于优化 mRNA 疗法的设计。
核糖核酸(RNA)可分为两类:编码蛋白质的 RNA 和非编码 RNA。前者在分子生物学的中心法则中充当遗传信息的载体,而后者在各种重要的细胞过程中发挥着多种作用[1]。RNA 的重要性在众多领域得到了强调,从基础的分子生物学和遗传学到生物技术和医学[2, 3]。例如,mRNA 在翻译过程中的作用使其成为基于 RNA 的疗法的焦点,特别是在针对诸如 COVID-19 等传染病的 mRNA 疫苗的背景下[4, 5]。鉴于 RNA 的重要性以及 RNA 生物学的复杂性,开发先进的计算方法来解析其繁琐的能力和调控网络的兴趣日益浓厚。
大型语言模型(LLMs),一种源自自然语言处理的人工智能形式,在 RNA 研究中已成为一种强大的方法。通过对大量 RNA 序列进行预训练,并对实验数据进行微调,用于 RNA 的 LLM 在各种与 RNA 相关的下游任务中表现出良好的性能,例如预测 RNA 功能和结构。RNA-FM [6] 是首个 RNA 基础模型,它在 RNAcentral [7] 数据库中的非编码 RNA 片段上进行了预训练,并经过微调以解决包括 RNA 二级结构预测在内的多个任务。RNAErnie [8] 是基于增强知识整合表示(ERNIE) [9] 框架构建的,它在 RNAcentral 数据库上使用了基于模式的预训练策略,并在多个任务中表现出优于其他基准模型的优越性能 [8],包括 RNABERT [10]、RNA-MSM [11] 和 RNA-FM。具有 650 万个参数的 RiNALMo 在非编码 RNA 上进行了预训练,是迄今为止公布的最大的 RNA 语言模型,并在包括 RNA 二级结构预测在内的多个下游任务中实现了最先进的性能 [12]。所有这些方法本质上都是基于 Transformer [13] 架构的。然而,由于注意力机制会随着序列长度的增加而呈平方级增长 [13],因此这些模型会受到输入大小的限制,并且通常无法将完整的 mRNA 序列作为一个整体进行处理。例如,RNA-FM、RiNALMo 和 RNAErnie 的最大输入长度分别为 1024、1024 和 512 个核苷酸。此外,当前的模型是在非编码 RNA 的截短短片段上进行预训练的,这可能会妨碍其在完整的 mRNA 相关任务上的表现。除了这些通用的 RNA 语言模型之外,还有专门针对 mRNA 未翻译区(UTR)的 RNA 语言模型,如 3’UTRBERT [14] 和 UTR-LM [15]。这些办法的设计并不适用于一般任务。实际上,这类专门的 RNA 语言模型在 UTR 相关任务上是否比通用的 RNA 基础模型更具优势也是有争议的。此外,编码序列众所周知会影响 mRNA 的翻译 [16, 17]。因此,尽管预测 5’UTR 或 3’UTR 对 RNA 特性(如 mRNA 翻译效率和稳定性)的影响对于设计 mRNA 疗法序列具有核心意义,但准确预测全长 mRNA 的特性对于进一步优化序列则是必不可少的。随着基于 mRNA 的疗法的兴起,这一点变得愈发重要。为了加快由人工智能驱动的 RNA 生物学和医学研究,对一种能够处理全长非编码 RNA 和蛋白质编码 mRNA 的新型 RNA 基础模型的需求日益增长。
为解决这些问题,大家创建了一种全新的全长 RNA 语言模型,名为 HydraRNA。该模型基于一种结合双向状态空间模型[18]和多头注意力机制[13]的混合架构,并且在 mRNA 和非编码 RNA 序列上进行了预训练。我们将 HydraRNA 应用于 10 种与 RNA 相关的任务中,包括 RNA 分类、RNA 二级结构预测、RNA 结合蛋白结合位点、剪接和聚腺苷酸化位点、mRNA 稳定性和翻译效率。在 10 个下游任务中,HydraRNA 在 8 个任务中均优于当前的 RNA 语言模型。此外,HydraRNA 能准确预测突变的影响,并估计 mRNA 不同部分对 RNA 稳定性和翻译的相对贡献。总的来说,我们证明了 HydraRNA 是一种有价值的工具,用于剖析 mRNA 的多种特性,这将进一步加速基于人工智能的 RNA 生物学和医学研究。
代码
https://github.com/GuipengLi/HydraRNA
参考
- HydraRNA: a hybrid architecture based full-length RNA language model
- https://github.com/GuipengLi/HydraRNA



 浙公网安备 33010602011771号
浙公网安备 33010602011771号