dataframe 和 numpy 数组有什么不同?
在cropformer相关的基因组预测任务场景中,DataFrame(通常指Pandas DataFrame)与NumPy数组均为数据处理核心格式,但二者在数据结构、功能定位、适用场景上存在显著差异,具体区别可结合文献中Cropformer的数据流处理需求(如基因型编码、特征选择、模型输入)展开分析:
1. 数据结构与存储特性
- DataFrame:
是一种二维表格型数据结构,兼具行索引(Index)和列索引(Columns),支持存储不同数据类型(如字符串型的SNP碱基、数值型的编码结果、文本型的样本ID)。例如,读取[test.ped](https://www.cnblogs.com/seryn/p/19171842)文件时,DataFrame可同时保留前6列的样本系谱信息(字符串型,如个体ID“MG_998_X_MG_1522”)和第7列及以后的SNP碱基数据(字符型,如“C”“T”),且通过列索引可直观区分不同数据类型的含义。
核心特点是结构化、带标签,便于人类理解数据含义,适合数据预处理阶段的筛选(如base_from_ped函数提取第7列及以后的SNP序列)、清洗和标注。
import pandas as pd
ser = pd.Series([4,7,5,3])
print(ser)
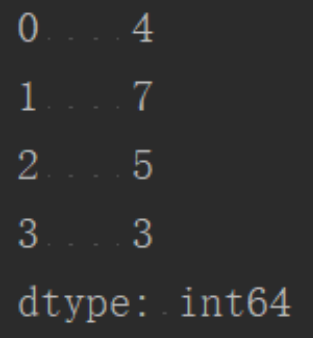
如上所示:0,1,2,3是索引,4,7,5,3是值,其中索引是可以指定的.
import pandas as pd
data = {'color':['blue', 'green', 'yellow', 'red', 'white'],
'object':['ball', 'pan', 'pencil', 'paper', 'mug'],
'price':[1.2, 1.0, 0.6, 0.9, 1.7]}
frame = pd.DataFrame(data)
print(frame)
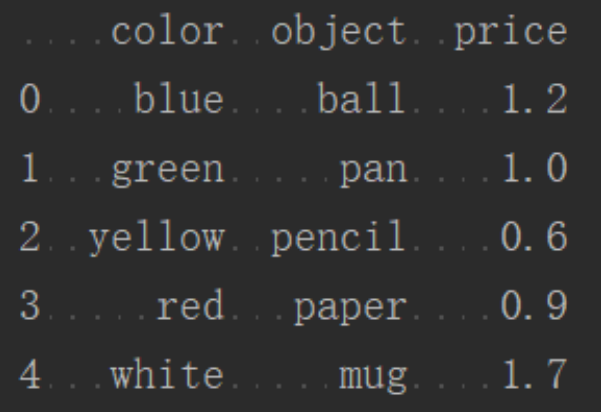
- NumPy数组:
是一种同构多维数组(通常为二维,即“样本×特征”矩阵),仅支持存储单一数据类型(如纯数值型、纯字符型),且无显式行列标签,通过索引(如X[:, 0])定位数据。例如,Cropformer模型训练时,需将DataFrame格式的SNP编码结果(0-9的数字)转换为NumPy数组,确保输入特征均为数值型,适配深度学习框架(如PyTorch、TensorFlow)的计算要求。
核心特点是非结构化、无标签,专注于数值计算效率,适合模型训练阶段的矩阵运算(如CNN卷积、互信息计算)。
import numpy as np
e = np.array([[1,2,3],[4,5,6],[7,8,9]])
print(e)

2. 功能定位与操作场景
-
DataFrame:适配“数据预处理”阶段
在Cropformer的数据流中,DataFrame主要用于原始数据读取、特征筛选、结构化管理,对应文献中“基因型数据编码前的准备工作”:- 读取非规整数据:如
test.ped中混合了系谱信息(前6列)和SNP碱基(第7列后),DataFrame可通过header=None和列索引灵活区分,避免数据混乱; - 直观特征操作:如
base_from_ped函数通过chr_merge.iloc[i,6:]直接提取SNP列,无需关注具体列数,操作可读性远高于NumPy数组的切片; - 数据类型兼容:处理SNP编码时,可先以DataFrame存储字符型碱基对(如“AA”“AT”),再通过
base_trans_num函数转换为数值型,过程中无需频繁转换数据格式。
- 读取非规整数据:如
-
NumPy数组:适配“模型计算”阶段
NumPy数组主要用于数值计算、模型输入,对应文献中“特征选择与模型训练”的核心流程:- 高效矩阵运算:如
MICSelector类中mutual_info_regression函数计算互信息时,需输入NumPy数组以实现快速的向量运算,避免DataFrame的标签解析开销; - 适配深度学习框架:Cropformer的CNN层、自注意力层(或Hyena算子)均需接收NumPy数组格式的输入(再转换为张量),例如将筛选后的Top-10000个SNP特征(
X_train_selected)以NumPy数组传入模型,确保计算效率; - 维度一致性保障:NumPy数组的“同构性”可避免数据类型混杂导致的模型报错,例如SNP编码后的数字(0-9)需统一为数值型数组,才能进行后续的卷积特征提取。
- 高效矩阵运算:如
3. 与Cropformer任务的适配性对比
| 对比维度 | DataFrame | NumPy数组 |
|---|---|---|
| 数据类型支持 | 多类型(字符串、数值、文本) | 单类型(如纯数值、纯字符) |
| 行列标签 | 有显式标签(便于数据理解) | 无标签(依赖索引定位) |
| 核心优势 | 结构化管理、灵活预处理 | 高效数值计算、适配模型输入 |
| Cropformer场景 | 读取test.ped/test_label.csv、SNP序列提取、编码结果暂存 |
互信息计算、特征选择、模型训练输入 |
| 对应文献步骤 | 基因型数据格式转换前的准备 | 特征选择与模型输入 |
总结
在Cropformer的基因组预测流程中,DataFrame与NumPy数组是“预处理→计算”流水线的两个关键环节:
- DataFrame以“结构化、带标签”的优势,解决原始基因型数据(如
test.ped)的读取、筛选和标注问题,确保数据预处理的直观性和灵活性; - NumPy数组以“同构、高效计算”的优势,满足互信息特征选择、深度学习模型训练的数值运算需求,是连接数据预处理与模型核心计算的桥梁,二者协同支撑文献中“从基因型到表型预测”的完整技术路径。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号