转:用K-Means聚类分析做客户分群
转:
https://www.cnblogs.com/niniya/p/8784947.html
聚类指的是把集合,分组成多个类,每个类中的对象都是彼此相似的。K-means是聚类中最常用的方法之一,它是基于点与点距离的相似度来计算最佳类别归属。
在使用该方法前,要注意(1)对数据异常值的处理;(2)对数据标准化处理(x-min(x))/(max(x)-min(x));(3)每一个类别的数量要大体均等;(4)不同类别间的特质值应该差异较大
一、K-means聚类步骤:
(1)选择k个初始聚类中心
(2)计算每个对象与这k个中心各自的距离,按照最小距离原则分配到最邻近聚类
(3)使用每个聚类中的样本均值作为新的聚类中心
(4)重复步骤(2)和(3)直到聚类中心不再变化
(5)结束,得到k个聚类
二、评价聚类的指标:
(1)inertias:是K-Means模型对象的属性,它作为没有真实分类结果标签下的非监督式评估指标。表示样本到最近的聚类中心的距离总和。值越小越好,越小表示样本在类间的分布越集中。
(2)兰德指数:兰德指数(Rand index)需要给定实际类别信息C,假设K是聚类结果,a表示在C与K中都是同类别的元素对数,b表示在C与K中都是不同类别的元素对数,则兰德指数为:

RI取值范围为[0,1],值越大意味着聚类结果与真实情况越吻合。
对于随机结果,RI并不能保证分数接近零。为了实现“在聚类结果随机产生的情况下,指标应该接近零”,调整兰德系数(Adjusted rand index)被提出,它具有更高的区分度:

ARI取值范围为[−1,1],值越大意味着聚类结果与真实情况越吻合。从广义的角度来讲,ARI衡量的是两个数据分布的吻合程度。
(3)互信息(Mutual Information,MI):指的是相同数据的两个标签之间的相似度,即也是在衡量两个数据分布的相似程度。利用互信息来衡量聚类效果需要知道实际类别信息。
假设U与V是对N个样本标签的分配情况,则两种分布的熵分别为:
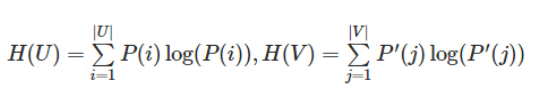 ,其中
,其中![]()
U与V之间的互信息(MI)定义为:
![]() ,其中
,其中![]() 。
。
标准化后的互信息(Normalized Mutual Information):
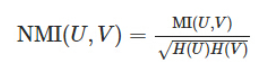
调整互信息(Adjusted Mutual Information):
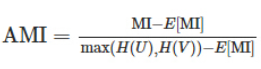
MI与NMI取值范围[0,1],AMI取值范围[-1,1],都是值越大说明聚类效果越好。
(4)同质化得分(Homogeneity):如果所有的聚类都只包含属于单个类的成员的数据点,则聚类结果满足同质性。取值范围[0,1],值越大意味着聚类结果与真实情况越符合。
(5)完整性得分(Complenteness):如果作为给定类的成员的所有数据点是相同集群的元素,则聚类结果满足完整性。取值范围[0,1],值越大意味着聚类结果与真实情况越符合。
(6)v_meansure_score:同质化和完整性之间的谐波平均值,v=2*(同质化*完整性)/(同质化+完整性),取值范围[0,1],值越大意味着聚类结果与真实情况越符合。
(7)轮廓系数(Silhouette):适用于实际类别信息未知的情况,用来计算所有样本的平均轮廓系数。对于单个样本,设a是该样本与它同类别中其他样本的平均距离,b是与它距离最近不同类别中样本的平均距离,轮廓系数为:

对于一个样本集合,它的轮廓系数是所有样本轮廓系数的平均值,轮廓系数取值范围是[−1,1],0附近的值表示重叠的聚类,负值通常表示样本被分配到错误的集群,分数越高,说明同类别样本间距离近,不同类别样本间距离远。
(8)calinski-harabaz Index:适用于实际类别信息未知的情况,为群内离散与簇间离散的比值,值越大聚类效果越好。
三、KMeans 主要参数
(1)n_clusters:k值
(2)init:初始值选择方式,可选值:'k-means++'(用均值)、'random'(随机)、an ndarray(指定一个数组),默认为'k-means++'。
(3)n_init:用不同的初始化质心运行算法的次数。由于K-Means是结果受初始值影响的局部最优的迭代算法,因此需要多跑几次以选择一个较好的聚类效果,默认是10,一般不需要改,即程序能够基于不同的随机初始中心点独立运行算法10次,并从中寻找SSE(簇内误差平方和)最小的作为最终模型。如果k值较大,则可以适当增大这个值。
(4)max_iter: 最大的迭代次数,一般如果是凸数据集的话可以不管这个值,如果数据集不是凸的,可能很难收敛,此时可以指定最大的迭代次数让算法可以及时退出循环。
(5)algorithm:算法,可选值:“auto”, “full” or “elkan”。"full"指K-Means算法, “elkan”指elkan K-Means算法。默认的"auto"则会根据数据值是否是稀疏的,来决定如何选择"full"和“elkan”。一般数据是稠密的,那么就是 “elkan”,否则就是"full"。一般来说建议直接用默认的"auto"。
四、k值的选择方法
基于簇内误差平方和,使用肘方法确定簇的最佳数量,肘方法的基本理念就是找出聚类偏差骤增是的k值,通过画出不同k值对应的聚类偏差图,可以清楚看出。
#导入库
import numpy as np import pandas as pd import matplotlib.pyplot as plt from sklearn.cluster import KMeans
#导入数据 df=pd.read_csv(r'E:\data analysis\test\cluster.txt',header=None,sep='\s+') print(df.head()) x=df.iloc[:,:-1] y=df.iloc[:,-1]
#肘方法看k值 d=[] for i in range(1,11): #k取值1~11,做kmeans聚类,看不同k值对应的簇内误差平方和 km=KMeans(n_clusters=i,init='k-means++',n_init=10,max_iter=300,random_state=0) km.fit(x) d.append(km.inertia_) #inertia簇内误差平方和 plt.plot(range(1,11),d,marker='o') plt.xlabel('number of clusters') plt.ylabel('distortions') plt.show()
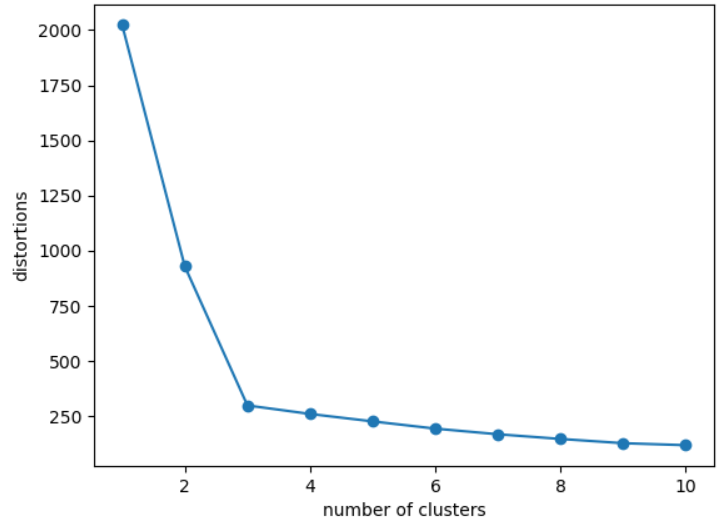
从图中可看出,k取3合适。
五、python做K-Means
继续使用上例中导入的数据。
#训练聚类模型
from sklearn import metrics model_kmeans=KMeans(n_clusters=3,random_state=0) #建立模型对象 model_kmeans.fit(x) #训练聚类模型 y_pre=model_kmeans.predict(x) #预测聚类模型
#评价指标 inertias=model_kmeans.inertia_ #样本距离最近的聚类中心的距离总和 adjusted_rand_s=metrics.adjusted_rand_score(y_true,y_pre) #调整后的兰德指数 mutual_info_s=metrics.mutual_info_score(y_true,y_pre) #互信息 adjusted_mutual_info_s=metrics.adjusted_mutual_info_score (y_true,y_pre) #调整后的互信息 homogeneity_s=metrics.homogeneity_score(y_true,y_pre) #同质化得分 completeness_s=metrics.completeness_score(y_true,y_pre) #完整性得分 v_measure_s=metrics.v_measure_score(y_true,y_pre) #V-measure得分 silhouette_s=metrics.silhouette_score(x,y_pre,metric='euclidean') #轮廓系数 calinski_harabaz_s=metrics.calinski_harabaz_score(x,y_pre) #calinski&harabaz得分 print('inertia\tARI\tMI\tAMI\thomo\tcomp\tv_m\tsilh\tc&h') print('%d\t%.2f\t%.2f\t%.2f\t%.2f\t%.2f\t%.2f\t%.2f\t%d'% (inertias,adjusted_rand_s,mutual_info_s,adjusted_mutual_info_s,homogeneity_s, completeness_s,v_measure_s,silhouette_s,calinski_harabaz_s))
inertia ARI MI AMI homo comp v_m silh c&h
300 0.96 1.03 0.94 0.94 0.94 0.94 0.63 2860
#聚类可视化
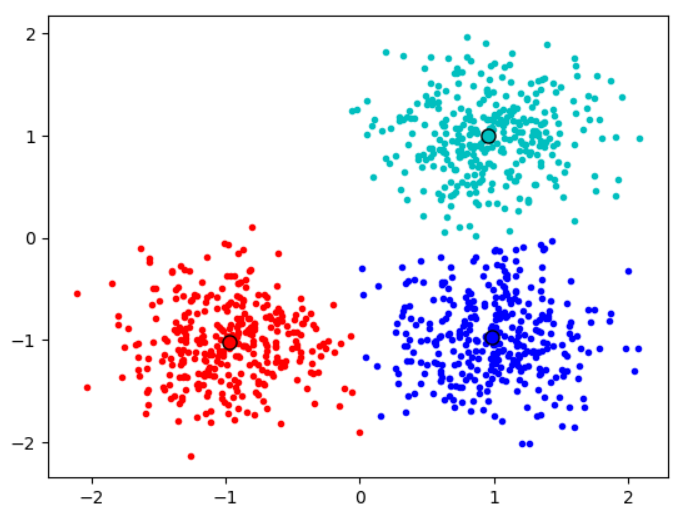
centers=model_kmeans.cluster_centers_ #类别中心
print(centers) colors=['r','c','b'] plt.figure() for j in range(3): index_set=np.where(y_pre==j) cluster=x.iloc[index_set] plt.scatter(cluster.iloc[:,0],cluster.iloc[:,1],c=colors[j],marker='.') plt.plot(centers[j][0],centers[j][1],'o',markerfacecolor=colors[j],markeredgecolor='k',markersize=8) #画类别中心 plt.show()
[[-0.97756516 -1.01954358]
[ 0.9583867 0.99631896]
[ 0.98703574 -0.97108137]]
参考:
《python数据分析与数据化运营》——宋天龙
https://blog.csdn.net/sinat_26917383/article/details/70577710
https://www.jianshu.com/p/b5996bf06bd6
https://blog.csdn.net/sinat_26917383/article/details/51611519
http://www.cnblogs.com/pinard/p/6169370.html




 浙公网安备 33010602011771号
浙公网安备 33010602011771号