【翻译】Species distribution modeling 3 不存在和背景点
3.”不存在“和”背景“点
一些早期的物种分布模型算法,比如Bioclim和Domain只在建模过程中使用‘presence’数据。其他方法也使用‘absence’数据和‘background’数据。逻辑回归是分析‘presence’和‘absence’数据的传统途径(而且仍然被大量使用,经常在一个广义线性建模(GLM)框架中实现)。如果你有一个从精心设计的调查中获得的巨大的包含presence/absence数据的数据集,你应该用一个可以使用这些数据的方法(例如,不要使用一个只考虑presence数据的建模方法)。如果你只有presence数据,你仍然可以使用一个需要absence数据的方法,用background数据去取代absence数据。
“背景”数据(e.g. Philips et al. 2009)不是在“不存在”地点试图去猜测,而是要描述研究区域环境的特征。在这个意义上,不管物种是在哪里被发现,背景都是相同的。“背景”数据建立了这个研究的环境范围,同时“存在”数据应该建立起物种相对于平均值更趋于存在的条件。一个有些相关但不同的概念,叫做“假设不存在”,也被用于生成逻辑模型中的非“存在”阶元。在这种情况下,研究者们有时试着猜测“不存在”可能发生的位置——他们会抽样除了“存在”地点的所有区域,或者抽样看起来不适合该物种生存的地点。我们倾向于“背景”概念,因为它请求更少的假设而且在处理“存在”和“背景”点之间的“overlap”时拥有连贯的统计方法(e.g. Ward et al. 2009;Philips and Elith, 2011)。
“调查不存在”数据是有价值的,结合“存在”记录,建立起了调查的区域,为调查工作提供了物种的流行度(prevalence )。在建模“仅存在”数据时,信息中缺乏“仅存在”数据,可能会造成实质性困难。然而,“不存在”数据仍然可能存在偏差或不完整,如关于可探测性的文献中所讨论的(e.g. Kéry et al., 2010)。
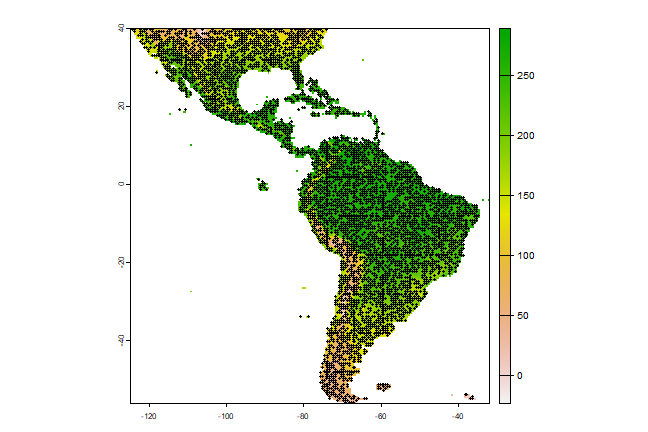
Dismo包中有一个用于在某个研究区域抽样随机点(“背景”数据)的函数。你可以使用‘mask’去排除无数据NA的区域,比如非陆地的区域。你可以使用‘extent’从所绘制的随机地点中去进一步限制区域。
在下方的例子中,我们首先得到了预测栅格数据(细节讨论见下一章)的文件名列表。我们在randomPoints函数中使用栅格作为一个‘mask’这样可以使背景点来自同一个地理区域,且仅在有价值的地方(在我们的情况下,指陆地)。
注意如果蒙板(mask)拥有经纬度坐标参考系统,函数randomPoints根据cell区域选择cell,因纬度而异(同 Elith et al., 2011)。
library(dismo)
# get the file names
files <- list.files(path=paste(system.file(package="dismo"), '/ex',
sep=''), pattern='grd', full.names=TRUE )
# we use the first file to create a RasterLayer
mask <- raster(files[1])
# select 500 random points
# set seed to assure that the examples will always
# have the same random sample.
set.seed(1963)
bg <- randomPoints(mask, 500 )
然后通过绘图检查结果。

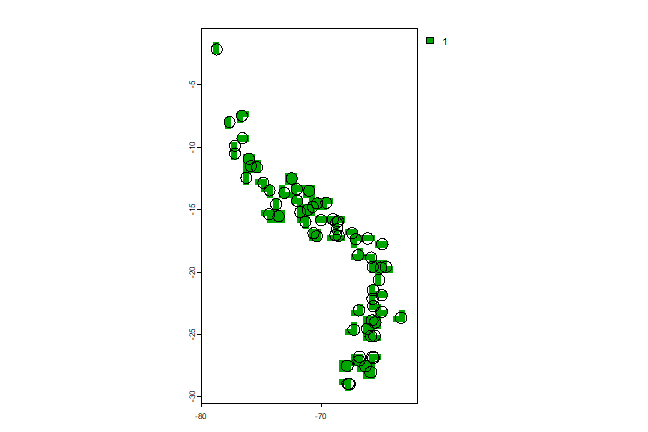
有一些途径可以用于抽样”假设-不存在“的点,也就是来自比‘background’更为限制性的区域的点。VanDerWal et al.(2009) 的抽样就是以“存在”点为半径。这里有一条途径,使用Solanum acaule数据来实现它。
我们首先读取在先前的章节中清理过的来自dismo包自带的csv文件中的S. acaule的子集数据:
file <- paste(system.file(package="dismo"), '/ex/acaule.csv', sep='') ac <- read.csv(file)
ac是一个data.frame。让我们将它转变成一个SpatialPointsDataFrame。
coordinates(ac) <- ~lon+lat
projection(ac) <- CRS('+proj=longlat +datum=WGS84')
我们首先创建一个‘circles’模型(参见有关geographic models的章节),使用一个50km的任意半径。
# circles with a radius of 50 km x <- circles(ac, d=50000, lonlat=TRUE) ## Loading required namespace: rgeos pol <- polygons(pol) ## Error in polygons(pol): object 'pol' not found
注意你应该已经为circles函数安装好rgeos包,去‘dissolve’这些圈(在圈重叠的地方去除边界)。
# sample randomly from all circles samp1 <- spsample(pol, 250, type='random', iter=25) ## Error in spsample(pol, 250, type = "random", iter = 25): object 'pol' not found # get unique cells cells <- cellFromXY(mask, samp1) ## Error in cellFromXY(mask, samp1): object 'samp1' not found length(cells) ## Error in eval(expr, envir, enclos): object 'cells' not found cells <- unique(cells) ## Error in unique(cells): object 'cells' not found length(cells) ## Error in eval(expr, envir, enclos): object 'cells' not found xy <- xyFromCell(mask, cells) ## Error in .doXYFromCell(object@ncols, object@nrows, e@xmin, e@xmax, e@ymin, : object 'cells' not found
绘图以检查结果:
plot(pol, axes=TRUE) ## Error in plot(pol, axes = TRUE): object 'pol' not found points(xy, cex=0.75, pch=20, col='blue') ## Error in points(xy, cex = 0.75, pch = 20, col = "blue"): object 'xy' not found
注意蓝色的点不都是包含在图形(圆圈)里,因为它们现在表示从掩模(mask)中所选的单元格的中心。我们可以选择仅使用具有圆心的单元格,使用overlay(叠加)功能。
spxy <- SpatialPoints(xy, proj4string=CRS('+proj=longlat +datum=WGS84'))
## Error in coordinates(coords): object 'xy' not found
o <- over(spxy, geometry(x))
## Error in over(spxy, geometry(x)): object 'spxy' not found
xyInside <- xy[!is.na(o), ]
## Error in eval(expr, envir, enclos): object 'xy' not found
通过raster函数rasterize或extract同样可以得到相似的结果。
# extract cell numbers for the circles v <- extract(mask, x@polygons, cellnumbers=T) # use rbind to combine the elements in list v v <- do.call(rbind, v) # get unique cell numbers from which you could sample v <- unique(v[,1]) head(v) ## [1] 15531 15717 17581 17582 17765 17767 # to display the results m <- mask m[] <- NA m[v] <- 1 plot(m, ext=extent(x@polygons)+1) plot(x@polygons, add=T)


 浙公网安备 33010602011771号
浙公网安备 33010602011771号